
引言
抗生素耐药性,是人类社会中最严重的医疗问题之一,目前每年在欧洲造成超过2.5万人死亡,在美国造成3.5万人死亡。几十年来,具有抗菌素耐药性的微生物数量一直在增加。这些微生物引起的感染缺乏有效的治疗方案。在过去几十年里,人们发现抗生素缺乏,严重影响了经济和人类的福祉。因此,当前重要的是,人类必须开发出能够对抗耐多药微生物,并减缓抗生素耐药性的进化和传播的抗菌素。
抗菌肽(AMPs),基本上是地球上所有生物自然产生的,通过多种作用机制作为防御系统,来抵御入侵的病原体。作为免疫系统的一部分,这些分子已经进化了数十亿年,并提供了广谱保护,以抵抗各种病原微生物,包括细菌、真菌、病毒和寄生虫等。然而,由于一些障碍,如毒性、有限的生物利用度、对病原体的特异性不足以及难以大规模生产等,阻碍了AMPs作为治疗传染病的治疗药物的发展。然而,最近,在设计策略、合成方法和输送系统方面的进展,正在扩大将合成AMPs转化为治疗耐药感染的下一代标准护理抗生素的可能性。在这里,研究者的综述提供了最新的计算和合成生物学工具的发展,以推进抗菌药物的应用,以帮助补充人类目前的抗生素库。
AMPs:用于工程的多功能支架
AMPs具有许多固有的医学特性:它们可以杀死细菌,靶向耐药微生物生物膜,促进伤口愈合,并调节免疫反应。这些分子的多功能特性(图1A),使它们成为药物开发的极好模板,包括有希望成为临床可用抗生素的替代品。例如,阳离子AMPs,如多粘菌素B和gramicidin S,已经在临床和外用非处方药中使用了很长时间,阳离子lantibiotic nisin通常被用作抗菌食品添加剂。然而,到目前为止,只有D2A21、SGX942、p2TA、omiganan等少数AMPs,在三期临床试验中证明了其有效性(表1),凸显了传统AMPs的局限性,这一点仍有待解决。在这里,研究者描述了这些不同分子的前景和挑战,并描述了通过合成生物学和计算生物学技术的发现、设计和生产,来最大化其潜力的途径。
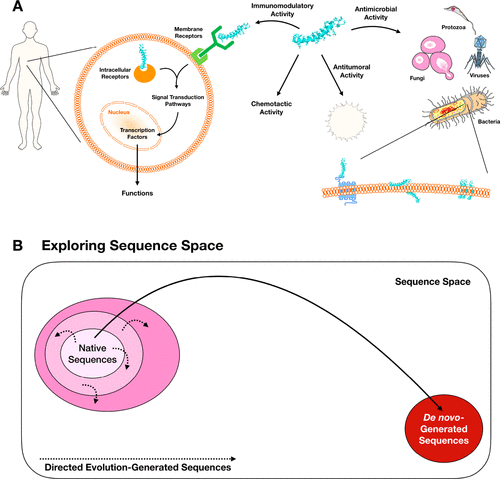
图1. AMPs的多功能和多样性的性质和序列空间探索的机会
图片来源于ACS Nano

表1. 最先进的AMPs在临床试验的范例
表格来源于ACS Nano
真正具有生物活性的肽分子的发现,可能有助于改变将AMPs转化为临床的成功率;然而,这仍然是一个突出的挑战。广阔的肽序列空间(图1B),提供了许多未被探索的可能性,并可能产生创新的肽序列,从而构成治疗。由于人类对不同肽序列的生物学、物理和化学性质知之甚少,因此很难预测肽所具有的众多生物活性。在过去的几十年里,一些计算资源被开发来促进AMP的设计,包括数据库、预测工具和设计模型。
分子描述符
计算方法和启发式代换,是预测AMP结构和功能的常用方法。例如,定量构效关系研究(QSAR),已被用于描述氨基酸残基及其特征。在这种情况下,物理化学描述符和结构描述符,可能相互关联或依赖,在QSAR建模和最终研究的生物活性中具有权重。Supady等人识别了结构描述符,如低能量构象,利用遗传算法,识别了表现出低能量剖面的多肽构象空间片段。作者预测了在全局最小值之上的一个能量窗口内的所有构象,而不是寻找全局最小能量。这一策略,防止了对特定力场参数化的潜在偏差结果,从而使搜索适用于更广泛的参数选择,并使对构象空间的探索更广泛。
计算机辅助设计
到目前为止,几乎所有的肽工程都涉及到,对自然产生的分子的修饰。然而,通过在自然序列中提出突变,计算机指导的肽设计允许探索更大的序列空间区域,这是以前没有在实验室或整个进化中分析过的。计算机指导设计的另一个优点是,它不需要对生成的每一个变体进行综合和筛选,从而节省了时间、劳动力和费用(图2A)。目前,计算机指导的预测AMP功能的方法的主要局限性之一是:需要标准化和可靠的生物数据作为有效的设计过程的输入。计算方法已经发展到可以训练计算机开发和增强分析,以优化传统的非计算设计方法,如不同类型的SAR策略。这些技术可以,与各种基于自然序列的结构相结合。肽可以精确地生成,也可以通过进化模板来生成生物活性肽来改进。
预测技术
当分子的潜在活性谱未知时,基于配体的模型对于AMP的预测是有效的。通过数据挖掘,Mardirossian等人从宽吻海豚身上识别出了Tur1A和Tur1B,这是两种富含脯氨酸的AMPs。作者发现,Tur1A通过内膜转运体SbmA和YjiL/MdM被大肠杆菌内化,而不造成膜损伤,并通过与核糖体结合,阻断起始期到延伸期的过渡,抑制细菌蛋白合成。另一方面,Tur1B适度地抑制蛋白质合成,似乎通过一种不同的,目前尚未清楚的作用机制,来发挥其抗菌活性。越来越多的大型数据库,使得机器学习(ML)/人工智能(AI)算法能够用于AMP预测。例如,Yoshida等人提出了一项概念证明研究,描述了通过序列空间探索发现AMP的有效策略。作者使用一个闭环的方法结合遗传算法,ML,和体外评价,以提高抗菌活性的多肽。这些努力从一个小的天然阳离子模板Temporin-Ali (FFPIVGKLLSGLL-NH2)中发现了44个先导肽。仅经过三次迭代,获得的击中数比原始型天然AMP活性高160倍。基于支持向量机(SVM)的分类器,也可以通过其作用机制有效地预测AMPs。Lee等人开发了一种分类器,用于研究由不稳定膜作用的螺旋AMPs。该方法认为螺旋结构是分析类似肽,最重要的活性描述符。作者观察到AMPs增加了细胞膜负高斯曲率,这是磷脂膜裂变过程的特征。
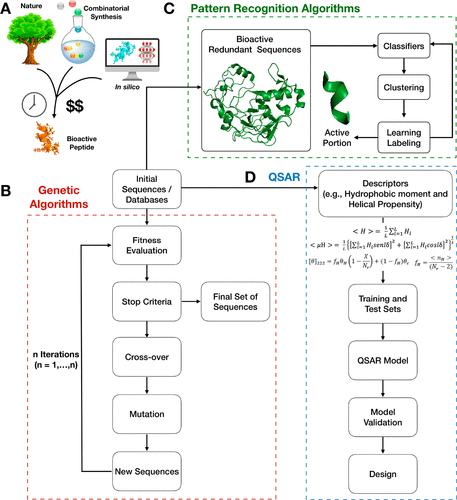
图2. 抗菌肽的开发工具
图片来源于ACS Nano
从头设计
AMPs的从头设计,导致产生与数据库中AMPs几乎没有相似之处的完全序列。从头技术,如从分类器和改进的算法所做的预测推断,已经产生了生物活性序列。该技术是药物活性肽的重要来源,如Bhardwaj等人提出的超稳定受限肽。作者设计了高度稳定的18-47残基,二硫化物交联肽,其中一部分是杂手性和/或N-C骨干环化的。
Hosseinzadeh等人,采用了从头设计混合手性肽大环的方法。作者通过几乎完全的主干取样、序列设计和能量绘景计算,列举了由L-和D-氨基酸组成的大环肽所采用的稳定结构。这种技术可以识别超过200个预计折叠成单一稳定结构的肽,当分析时,发现它们与计算模型预测的肽相似。
遗传算法
用于阐明结构-功能关系的计算方法,正在成为研究多肽与微生物膜相互作用的有用工具。最近的努力“训练”计算机执行达尔文的进化算法,并通过突变、选择和重组输入的肽序列,机器能够进化这些分子,构建具有体内抗感染活性的人工肽抗生素(图2B)。这些工具很有前途,但可以进一步优化。例如,遗传算法(GA)可以生成最优解,其中包含已有数据库中描述的大约一半的自然AMPs,从而为探索完全人工序列留下改进空间。Porto等人采用了一种令人兴奋的计算机辅助设计策略,来优化肽Pg-AMP120,其结果是将植物序列作为AMP模板,用于随后的计算机辅助合成序列设计。通过算法的中断来探索之前未探索的组合序列空间,从而使其达到稳定解。计算机制造的guavanin 2是一种很有前途的AMP,它在水中是无结构的,并且在疏水环境中经历了螺旋到螺旋的转变。最重要的是,guavanin 2在一个具有临床前相关性的小鼠感染模型中,显示了有希望的抗感染活性。
模式识别算法
算法也可以用于具有确定结构的肽的预测(图2C)。模式识别算法通常用于图像和语音识别,用于识别在较大蛋白质或前体结构中加密的氨基酸模式,以及分析大AMPs的内部结构,使识别特定的结构特征和抗菌活性的分子决定因素成为可能。先前对抗菌活性的序列要求的了解,使人类能够在大型数据库中搜索这些模式。例如,Pane等人利用模式识别技术,发现了人胃蛋白酶A中隐藏的AMPs。这些多肽对人类细胞具有低毒性,并在小鼠模型中表现出抗感染活性。序列比对是比较生物序列的常用方法,也是在包含数千种蛋白质的大型数据库中,搜索相似序列的最简单方法。有两种主要的方法来搜索AMP模式:隐马尔科夫模型(profile-HMM)和正则表达式(REGEX)。校准结果然后提交给特定的计算机程序,如HMMER或Pratt。REGEX的一个重要特征是,有助于功能的氨基酸理化性质可以通过经验的方式整合到平台中。例如,Silverstein等人表明,来自不同肽家族的一些序列可以很好地对抗配置-HMMs,正如观察到的硫蛋白和snakins/GASA肽。
除了用于AMP的鉴定外,REGEX还可以用于探索不同的生物功能和设计非常规的AMPs。一旦确定了这些肽的主序列,就可以设计具有相似氨基酸组成的序列,并保持相似的物理化学特征平均值,如平均疏水性或净电荷。该方法用于分析混杂肽,即在不同环境或条件下具有多种生物学功能的多肽。这种模式匹配的通用性,得到了Loose等人的证实,他们开发了一种不同的合理地AMP设计方法,从多个数据库中提取了大约700个AMPs的序列,产生了一系列具有类似抗菌应用特性的不同AMPs。
近年来,数据库中可用的信息越来越多,使得算法的设计,除了执行序列比对和氨基酸含量比较,能够提供结构信息和结构分类之间的其他一些结构工具。因此,由于大量的描述符依赖于结构信息,通过对肽和蛋白质的氨基酸序列进行非常精确的分析,可以实现对模式的准确预测。Pane等人报道AMPs的抗菌活性与产物Cm×Hn×L呈线性相关,其中C为净电荷,H为整个序列平均疏水性的一致平均值,L为AMP的长度。作者报告了一种有效的计算策略,来识别在较大蛋白质或前体结构中加密的AMPs。
Cardoso等人利用Joker算法从E. coli MerP片段中提取了AMP EcDBS1R5,这是一种18个氨基酸的肽,此前该算法预测它是一种抗菌剂。E. coli MerP片段展示了一个螺旋含量高而不是标准的可预测的物理化学特性,典型赋予小型螺旋阳离子抗菌活性肽,如低净正电荷和长度(+ 2)或高疏水性(∼80%的氨基酸序列的疏水残基)。这种修饰是通过插入一个富含赖氨酸的基序来实现的,该基序导致EcDBS1R5的疏水性(约58%)和更高的疏水矩,这些特性有助于EcDBS1R5在体内具有高的抗菌和抗感染活性,以及高的抗菌膜活性。
基于统计学肽设计
基于统计的计算方法代表了传统计算机引导的肽设计的替代策略。这些方法使用生物信息学工具,如统计建模、SAR研究、神经网络和ML,来分析和增强数据库中描述的AMPs的活性。Porto等人最近发表了AMP数据库和数据挖掘的广泛概述。计算方法结合结构和物理化学特征的使用可以导致抗菌分子的准确预测。
分子动力学
分子模型(MM)和分子动力学(MD),是分析AMPs在生物膜或模拟它们的人工膜上的结构与活性关系的有效方法(图2D)。MD可以用来推测AMPs的作用机制,也可以用来提取影响抗菌活性的生物描述符(图3A)。然而,时间尺度和通过经验分析验证的需要,仍然是这项技术的限制因素。最重要的生物过程,包括AMP的内化或在膜失稳之前与膜的相互作用,都是几毫秒长的。然而,目前的MD模拟运行在纳米到微秒的量级上。因此,对于复杂生物过程的精确建模,仍然存在几个数量级的差距。目前,只有少数几个小组能够在小范围内,进行验证MD模拟预测的实验。研究者预计MD模拟将能够预测AMP的结构,并提供这些制剂在与微生物接触时的完整原子细节轨迹,从而使计算机辅助机制阐明研究成为可能。例如,Melo等人通过结合量子力学和分子力学,开发了一套完整而全面的套件,用于对复杂过程进行精确的计算机辅助分析。作者将分子动力学和可视化软件(NAMD和VMD)与量子化学包ORCA和MOPAC合并,创建了动态过程的界面,如肽与膜和膜组件的相互作用。
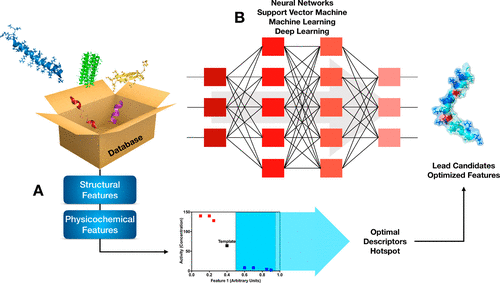
图3. 用于肽设计的高通量框架
图片来源于ACS Nano
自主学习计算方法
神经网络,特别是深度学习,作为一种计算机引导的方法,正在加速AMP的发展(图3B)。与其他方法相比,该方法具有直接预测AMP活性和通过自学习构建序列潜力的显著优势。例如,遗传算法的目标是基于输入序列进化序列,输入序列作为初始数据集,其结果不用于改进算法。
监督ML方法,用于发展抗菌活性预测模型。其采用了两种不同的预测策略:序列大小变化和序列顺序。一旦选择了序列顺序,大小就不能修改,根据每个位置的氨基酸残基频率进行预测。否则,如果序列大小变化是一个选项,序列将被转换为描述符。
AMP的异质性代表了肽设计领域的另一个挑战,预测方法,如遗传算法、ML和深度学习,已被用于补充其他序列搜索方法。另一个可以用来更好地理解和设计AMPs的重要工具是MM,通常与MD相关联。建模可以通过两种技术来识别相似的序列:线程和从头算建模。线程方法使用模板从查询序列中预测三维结构,而从头建模,也被称为从头建模或自由建模,利用设计的能量函数和广义的构象概率来指导构象搜索,预测一个没有先前结构信息的蛋白质结构。这些方法已成功地应用于半胱氨酸保守肽的筛选,包括防御素、环肽和蛇肽,以及抗利什曼肽的鉴定。与MM肽相关的另一个挑战,是将具有不同生物活性的相似结构与功能进行关联。
另外,深度学习技术也可以用来生成AMPs。Müller等人提出了,一种可通过组合设计生成从头生成多肽的循环神经网络,例如,使用不同氨基酸残基进行单突变筛选。该模型捕获了α-螺旋AMP序列的模式,从而从学习的环境中生成肽。该方法的结果是,82%的活性AMPs预测,而65%的随机采样序列与训练集相同的氨基酸分布。
最近FDA批准了磷酸二胺吗啡啉寡核苷酸,可用于治疗杜氏肌营养不良症。Wolfe等人使用ML预测了细胞穿透多肽,作为这些寡核苷酸的有效传递手段。成了64个寡核苷酸-肽缀合物,并将活性数据用于拟合随机决策森林分类器,以预测给定肽的共价结合是否会增强化合物的活性。所有计算预测的阳性序列,在检测中都是活跃的。
混合预测计算机引导方法,可作为AMP预测策略的替代方法。Schneider等人将混合网络和深度学习模型结合起来,创建了一种体系结构,可将多种多样的多维描述符组合在一个自组织图中。将描述符转换为二维图像进行进一步处理;然后,二维图像被用作前馈神经网络的输入层。与缺乏自组织映射的前馈网络分类器相比,所提出的混合模型提高了分类精度和具有更高的预测鲁棒性。
采用计算方法设计AMP的一个缺点是预测—验证步骤与下一轮的优化和AMPs预测无关。因此,需要更多的方法,来更全面地评价肽-膜的相互作用。如上所述,MD模拟是一种强大的方法,在AMPs和生物膜之间的相互作用的原子水平上提供动态信息(相对于静态)。尽管这种方法成本高、耗时长,但已被用于阐明AMPs的简单作用模式,并已被应用于设计通过形成小孔和/或破坏细胞膜发挥作用的肽。
展望与结论
至此,研究者描述了最近的一些进展,这些进展可能使AMP分子按需编程成为可能。经验和计算方法和工具的标准化,在生物学中仍处于起步阶段。结合计算机科学、建模和合成生物学的原理,将允许更系统地探索多肽的作用和活性模式,从而产生新的抗生素。
参考文献
Marcelo D. T. Torres, Jicong Cao, Octavio L. Franco, Timothy K. Lu, and Cesar de la Fuente-Nunez. ACS Nano 2021, 15, 2, 2143-2164. DOI: 10.1021/acsnano.0c09509.