靶点
RNA 聚合酶II
计算方法
QM/MM MD
计算软件
QChem/Amber
作者与单位
香港理工大学黄旭辉教授课题组(http://compbio.ust.hk)
发表杂志
Nature Catalysis
前言
酶催化机制的理论研究一直以来是计算化学、计算生物学的重要方向之一,其中QM/MM MD是当前最为广泛应用且较为认可的计算模拟方法。而计算与实验相结合,往往能更加深入探究复杂的酶催化机制。在这分享香港理工大学黄旭辉教授在2019 Nature Catalysis上一篇关于RNA聚合酶II 催化内裂过程的理论模拟与实验验证相结合的文章。[1]
背景
在基因表达中,为了保证较高的转录保真度,RNA聚合酶(RANPs)能够通过其内在的核酸酶活性来校对新生RNA的转录,这是一种水解裂解反应,可以进一步受到其他转录因子的刺激。[2]而在一些物种中,转录因子的刺激并非必需,如酿酒酵母RNA 聚合酶II (Pol II) (Fig.1a)的内在裂解活性已经足以维持细胞的活力。
RNA聚合酶利用相同的活性位点进行核苷酸聚合和内在的裂解反应(下简称内裂)。在单个核苷酸发生错配时,RNA聚合酶采取单-核苷酸-回溯态(one-nucleotide-backtracked state),在无转录因子参与下通过水解磷酸二酯键发生内裂并移除3’-端的核苷酸(Fig.1b)。研究结果表明,RNA聚合酶内裂采取了与其他酶中(如多种DNA聚合酶)类似的双金属机制。然而对于RNA聚合酶来说,核心问题仍然是确定这个裂解反应所涉及的广义酸碱(general acid/base)问题。
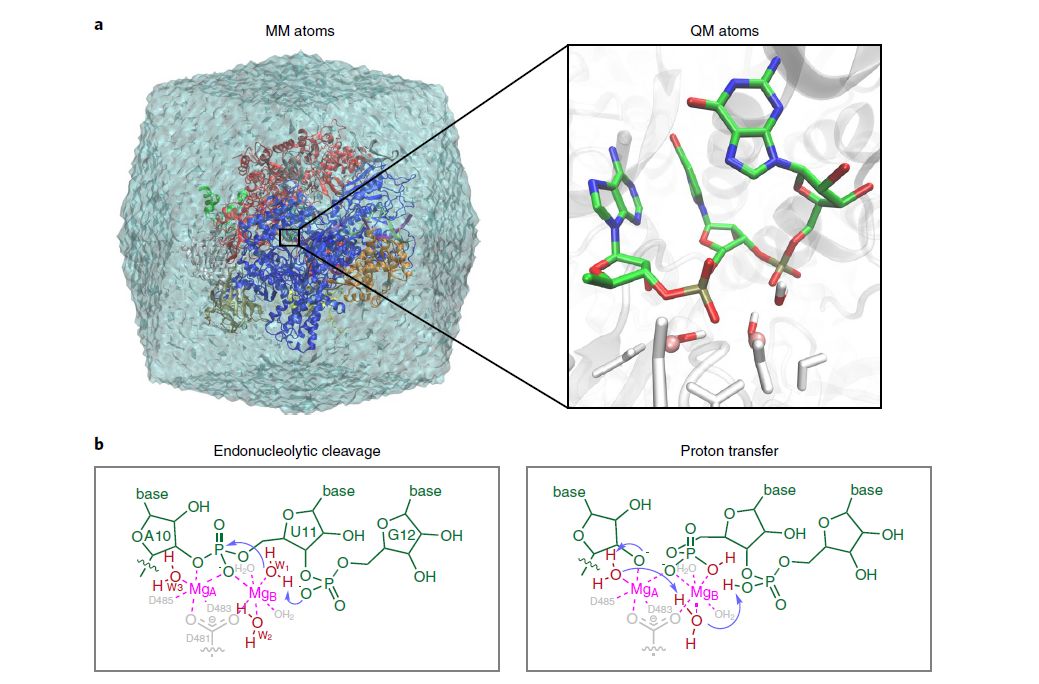
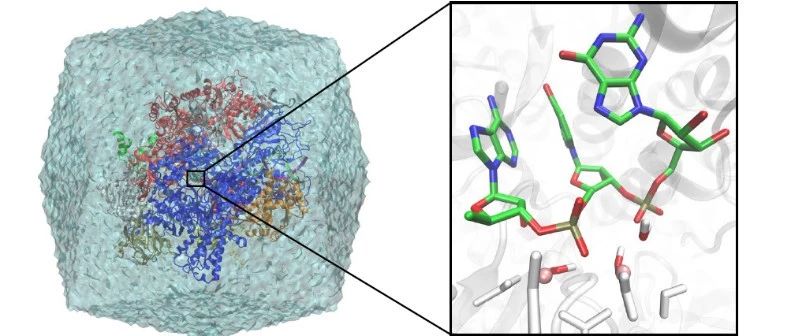
Fig. 1 a, QM/MM-MD模型(PDB:3GTG)及QM区域划分。b, 反应机制示意图, 步骤1(左):核苷酸内切断裂,以水为亲核试剂,端位磷酸O作为广义碱;步骤2(右):水介导的质子转移过程。
图片来源Nature Catalysis
虽然时间分辨x射线实验研究已经成为实时监测酶反应过程的一项强有力的技术,[3]但由于晶体结构分辨率的限制,氢原子无法准确定位,仍然无法完整揭示RNA 聚合酶II (Pol II)中的反应机制。尤其是对于参与激活亲核进攻的广义碱,以及参与质子化O离子离去基团的广义酸的分析研究。之前的研究推测末端核苷酸的碱基或参与进来的氨基酸残基(如His等)可能作为广义碱,[4]而对于广义酸则仍不清楚。考虑到实验研究的局限,计算研究可以作为强大的与实验互补的方法,来揭示反应机制中的细节。[5]
于是研究人员采用从头算量子力学/分子力学的分子动力学模拟(ab initio quantum mechanics/molecular mechanics– molecular dynamics, aiQM/MM–MD)方法,解析Pol II 回溯过程中的内裂机制。研究结果表明,端部错配核苷酸上的磷酸O原子出人意料的充当广义碱,并协助进攻水(attacking water)的去质子化。而暴露的3 ‘ –端氧阴离子在无广义酸的情况下,通过水介导的质子转移进行质子化。上述理论计算预测得到了生化实验的验证。
研究内容
RNA 聚合酶II (Pol II)内裂的协同反应机制
研究人员基于回溯态的Pol II体系进行了aiQM/ MM-MD模拟,体系包含103 QM区原子和372091 MM区原子(Fig. 1),使用方法基组为B3LYP/6-31G*。在典型的aiQM/ MM-MD模拟中,一般采用球边界方法将中心区域封闭在截断的球体中(如Qchem-Tinker)。而该研究采用了新近开发的Qchem-Amber考虑了周期性边界条件,可以更准确地描述远距离静电相互作用。[6]由于体系中含有两个金属Mg2+离子,在QM/ MM-MD模拟前首先仔细验证了初始结构,确认催化所需的重要结构保持得很好,包括两个Mg2+离子之间的距离以及它们的配位。
通过MD模拟得到合理的复合物初始模型后,沿反应坐标进行QM/MM计算,先后获得一维(1D)和二维(2D)势能分布图,然后以伞形采样(Umbrella Sampling)的方式基于QM/ MM MD模拟进行充分的采样,沿二维势能面上最小势能路径进行采样,共收集了115个窗口(每个窗口收集7ps轨迹),利用加权直方图分析方法(weighted histogram analysis method,WHAM)计算平均力势(PMF)即获得反应自由能曲线。同时通过将每个窗口的QM/ MM MD模拟时长加倍来验证QM/MM MD自由能模拟是否收敛。
结果表明,回溯态Pol II中错配核苷酸的磷酸二酯键断裂过程采取协同机制。研究人员通过系统的调查反应中心附近可作为潜在广义碱来激活亲核试剂的基团/原子,发现并不是Pol II中的氨基酸残基而是下游的磷酸O (G12 pro-Rp)作为广义碱来激活MgB-结合的亲核进攻试剂(活化能垒~29 kcal mol−1, 见Fig.2)。反应过程所历经的态分别为反应态RS(自由能0 kcal mol−1),过渡态TS1(29.18 ± 0.58 kcal mol−1),中间体态IS(20.43 ± 0.99 kcal mol−1),过渡态TS2(28.66 ± 0.31 kcal mol−1)和产物态PS(0.77 ± 0.91 kcal mol−1)。
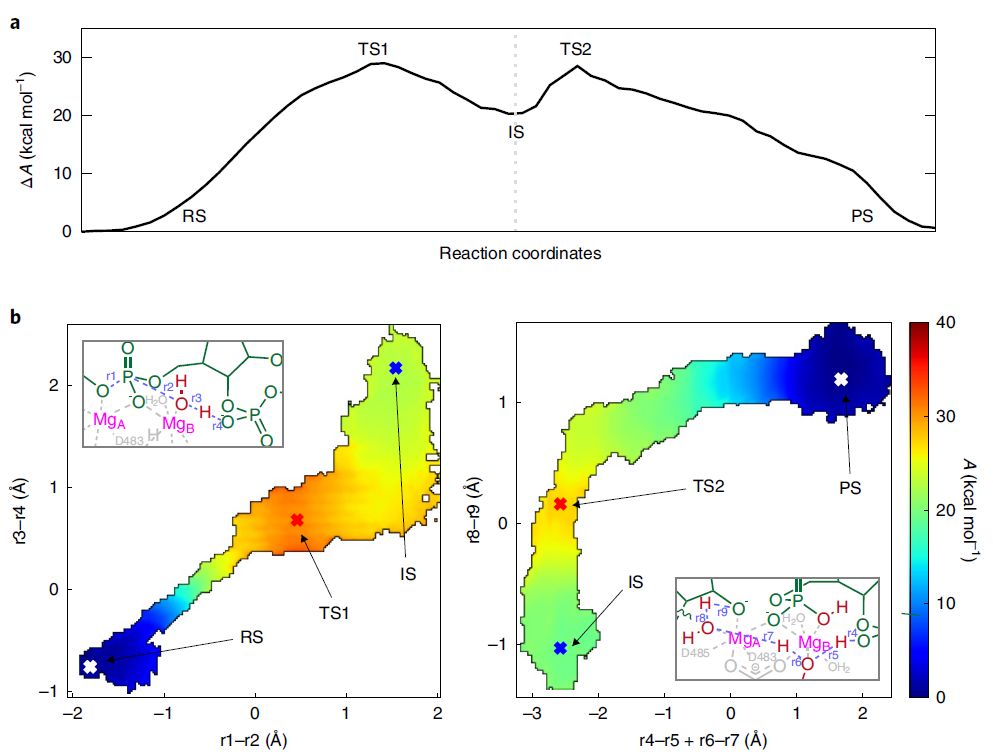
Fig. 2 a,一维自由能曲线。b, 步骤1(左)和步骤2(右)的二维反应坐标及自由能面。
图片来源Nature Catalysis
QM/ MM-MD模拟表明,内部裂解的协同机制与过渡态(TS1)有关,该过渡态包含与亲核进攻试剂和离去基的部分键合(Fig. 3)。这会导致亚稳态IS出现,其中包含一个暴露的A10 (左侧核苷酸)3 ‘-端,并被MgA的配位水进一步质子化。
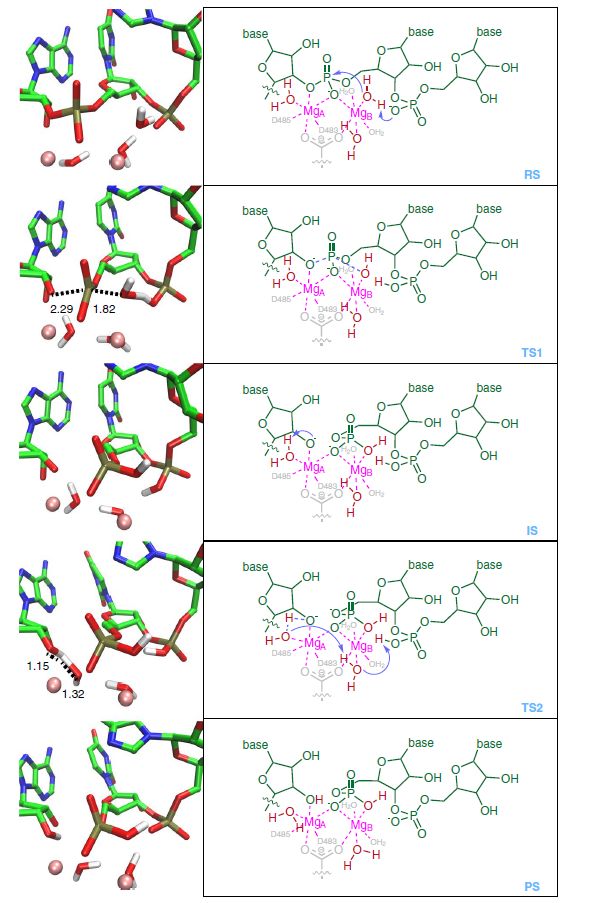
Fig. 3 反应途径中各个态的结构及反应历程示意图。
图片来源Nature Catalysis
反应的最后一步是完成质子转移过程,质子转移将使活性部位的水分子恢复中性。为了验证TS的结构,将TS1和TS2进行弛豫(注:TS结构很不稳定,伞型采样中通过外加限制力才可对不稳定的TS进行采样,此处弛豫是将TS的限制放开,进行QM/MM MD采样。理论上采样可以够捕捉到由TS向两侧局部能量最小态的运动,从而可辅助验证TS和反应途径的合理性),弛豫途径与所确定的机制完全一致:即进攻水被末端磷酸O去质子化。同时还检测了IS的稳定性,其结构包含一个质子化的末端磷酸和一个裂解的磷酸二酯键,这进一步支持了3’-端核苷酸磷酸O接受质子并作为广义碱的能力(Fig. 3)。研究所提出的下游磷酸(G12 pro-Rp) O原子作为内裂过程的广义碱 ,得到了酿酒酵母Pol II和嗜热菌RNAP的单-核苷酸-回溯态晶体结构的支持。这二者结构中都包含扭曲骨架结构(此处指核酸骨架或核酸主链),位于回溯核苷酸和上游核苷酸之间,使前者的pro-Rp O原子能够靠近活性位点(Fig. 4)。
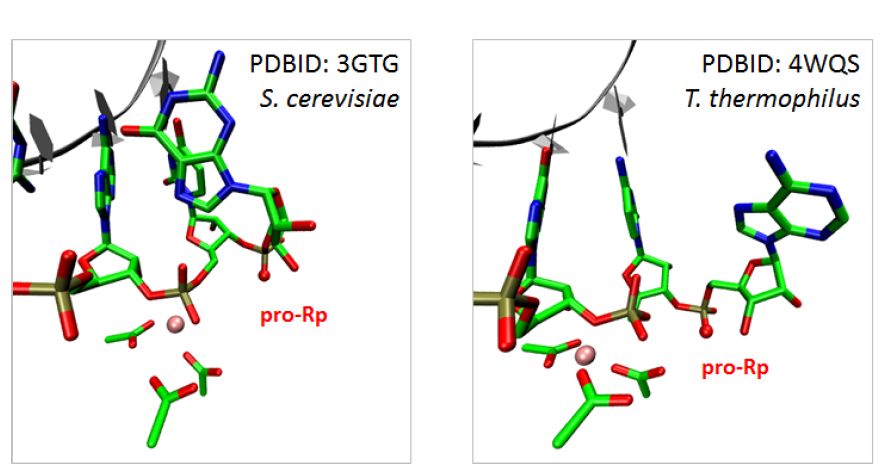
Fig. 4 酿酒酵母Pol II和嗜热菌RNAP的单-核苷酸-回溯态晶体结构。二者都包含扭曲的骨架结构使pro-Rp 原子靠近活性位点。
图片来源Nature Catalysis
3′-RNA核苷酸磷酸O原子作为广义碱(实验验证部分)
上文提到研究所提出的机制中,G12的pro-Rp O原子作为激发进攻水裂解的广义碱。因此G12 pro-Rp O原子与进攻水之间的距离是激活的重要因素。为了直接验证这一机制,后续采用了两种不同的方案来研究pro-Rp O在内裂中的作用。
在第一种方案中,通过将末端G12和U11之间的3 ‘ -5 ‘磷酸二酯键替换为2 ‘ -5 ‘磷酸二酯键,增加了G12的pro-Rp O和攻击水的距离。其结果使得G12非桥接磷酸O与进攻水之间的距离大大增加,从而使其不能再作为广义碱。为了证明这一点,对2 ‘ -5 ‘RNA进行MD模拟,结果显示G12 pro-Rp O与进攻水上氢平均距离为5.47±0.29 A,大于3’ -5的3.44±0.29 A,严重影响催化活性。
为进一步验证上述理论观察,研究人员使用Fig. 5a中描述的骨架进行了内裂实验,RNA的G12呈3 ‘ -5 ‘或2 ‘ -5 ‘连接方向。通过对Pol II的内裂实验分析,可以清楚地看出U11和G12核苷酸两者之间的键合发生了变化,并大大降低了裂解率。在量化裂解速率时,研究人员发现双相离解方程(double-phase dissociation equations)比单相(single-phase)更适合拟合实验数据(Fig. 6)。值得注意的是,2 ‘ -5 ‘连接的裂解速率(kfast)比3 ‘ -5 ‘连接裂解速率(慢14倍以上(0.0091±0.0022 min-1 vs 0.14±0.02 min-1)。此外, 3 ‘ -5 ‘连接骨架的快速相(fast phase, 注:双相法分为快速和慢速相,比例根据实验拟合结果得出)比例(69.5±6.0%)也高于2 ‘ -5 ‘连接支架(32.1 ± 8.6%)。
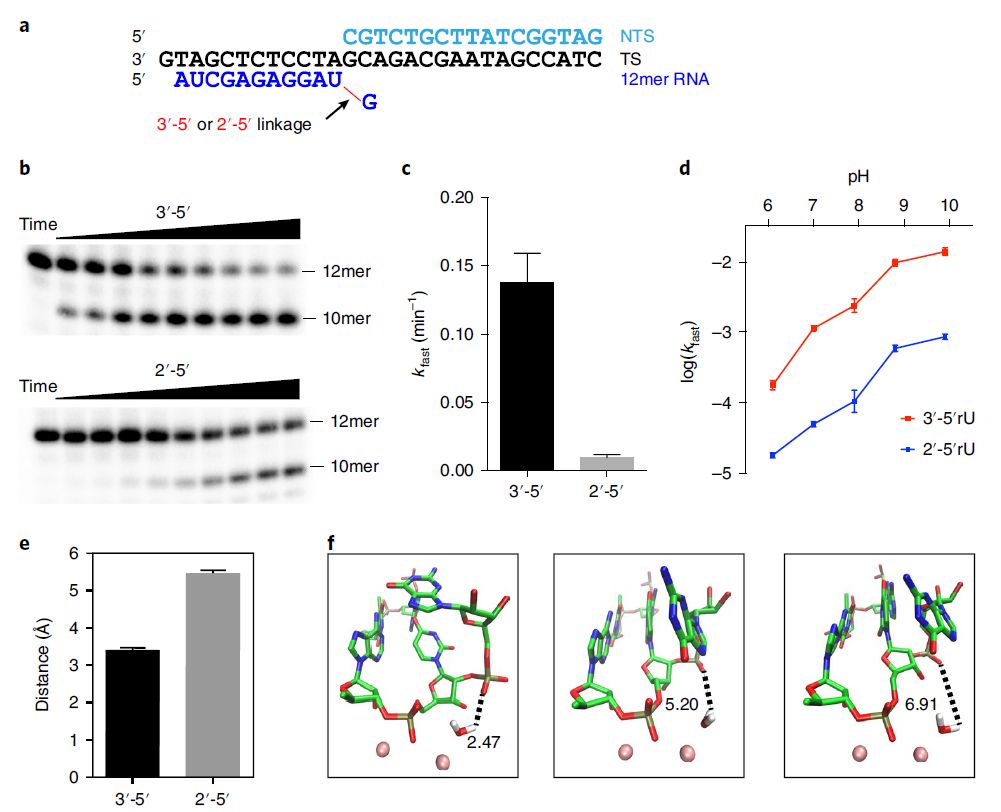
Fig. 5 改变RNA 3 ‘-端的连接降低内裂比率。a,内裂实验所用核酸序列。b,c, 与3’-5’ 连接相比,2’-5’连接将会大大降低内裂速率。d, pH对反应的影响。e, G12 pro-Rp O原子与进攻水之间的平均距离。f, 三个2 ‘ -5 ‘连接骨架的代表性类反应物结构,标注为G12 pro-Rp O原子与进攻水距离。
图片来源Nature Catalysis
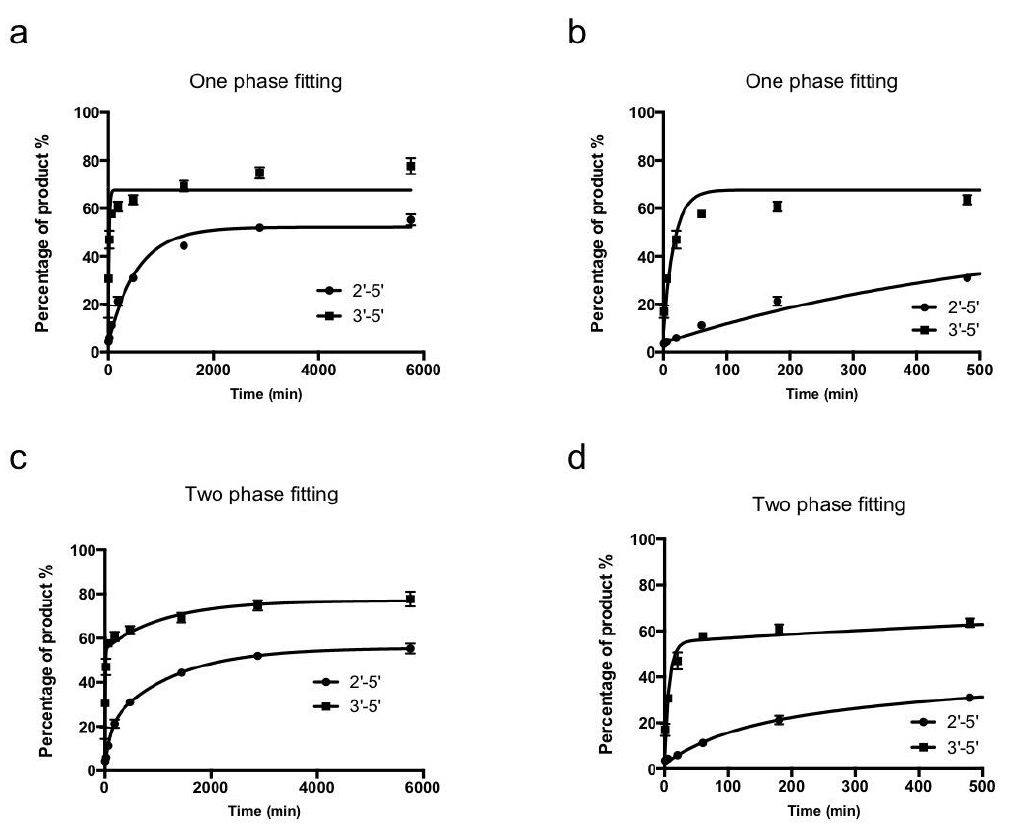
Fig. 6 用两种方法拟合内裂反应产物曲线(单相(a) vs双相(c))。右图为早期时间点的对应放大图。
图片来源Nature Catalysis
这些实验揭示了两个重要的观察结果,与G12在回溯内裂过程中的作用有关。首先,双相拟合结果表明,回溯过程可能同时存在两种不同的内裂机制。
其次, kfast受回溯核苷酸磷酸二酯键连接方式的高度(3’-5’ vs 2’-5’连接)。这一结果明确表明,回溯核苷酸的连接改变会强烈影响其内在的裂解活性。这种改变直接影响了磷酸基的位置,(Fig. 5f),因此,裂解活性降低了14倍,并且快速相比例也降低。这些结果支持理论计算中提出的磷酸盐基团在内裂过程中的促进作用。
为了检验2 ‘ -5 ‘和3 ‘ -5 ‘连接骨架的内裂机制是否相同,研究人员比较了2 ‘ -5 ‘和3 ‘ -5 ‘连接的pH谱。高pH值能通过去质子化进攻水来刺激磷酸二酯水解,因此pH-依赖的裂解曲线可以为反应机制提供重要信息。实验获得的pH谱非常相似(Fig. 5d),这表明是相同的一组原子参与了裂解反应,这个结果与前面获得的结论一致。
为了进一步证实G12 pro-Rp O原子的参与,再采用第二种方案进行实验,在不显著改变活性位点原子结构的情况下,使用S原子来取代这个关键O原子(Fig. 7a)。含有硫代取代物的寡核苷酸会产生两个立体异构体,即(pro-)Rp-S和Sp-S(Fig. 7a)。采用高效液相色谱法进行分离,从混合物中提取Rp-S和Sp-S寡核苷酸,并测定分离组分的裂解速率。在得到的两个峰(peak1和peak2)中,peak2的裂解速率显著降低,而peak1的裂解变化不大(Fig. 5b, c)。这一结果与提出的反应机理是一致的。一方面,立体定向的Rp-S取代了末端磷酸O (Fig. 7a左)会降低去质子化水的能力,造成裂解速率降低,因此实验Rp-S取代对应peak2。另一方面,在Sp-S取代过程中,O仍然是质子的受体,裂解速率受到的影响很小(Fig. 7a右),因此认为Sp-S取代对应于peak1。
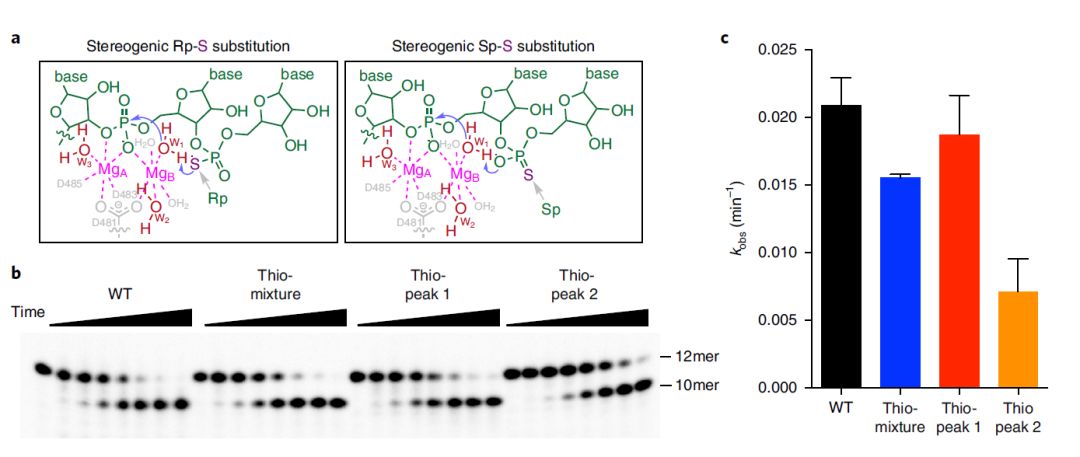
Fig. 7 S取代磷酸O对内裂的影响。a,两种不同的硫取代异构体结构。b,c, 硫代异构体对内裂速率的影响。
图片来源Nature Catalysis
3′-RNA 核苷酸碱基作为可选广义碱的有限作用
之前的研究表明,虽然细菌和真核生物之间存在差异,但回溯核苷酸的碱基可能直接作为广义酸/碱参与内裂过程。为了研究鸟嘌呤(G)碱基是否能作为广义酸/碱参与内裂,实验将鸟嘌呤与取代N7原子鸟嘌呤的酶催化内裂速率进行比较。结果表明二者对内裂速率的影响都非常小。与这些实验相一致,以G12的N7原子为广义碱,进行QM/MM计算的结果显示活化能势垒增加了20 kcal mol-1。对G12的O6原子作为广义碱的可能性同样进行了QM/MM计算,结果显示活化能势垒同样增加了~20 kcal mol-1(Fig. 8)。综上所述,G12的碱基对Pol II内裂的贡献非常小。
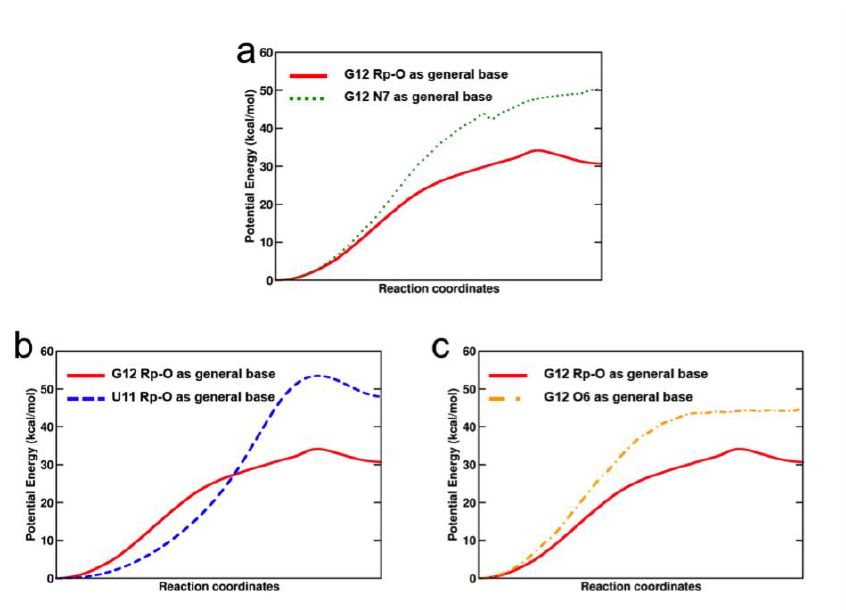
Fig. 8 QM/MM计算排除三个可能作为广义碱的碱基原子。
图片来源Nature Catalysis
Pol II 残基不太可能作为广义酸
考虑到天冬氨酸D485是距离新出现的A10 3’-端O原子最近的蛋白残基,研究者采用质子化的D485进行MD模拟检测D485是否可以作为广义酸,以及质子化的D485在回溯态Pol II的活性位点是否稳定。在所获得的三段MD轨迹中,其中一个呈现了一个扭曲的活性位构型,U11不与MgA配位,而另外两轨迹中质子化的D485保持稳定并指向口袋外侧(Fig. 9a)。为了进一步研究在去除最近的水分子后,D485的质子是否能够被成功的容纳,计算使用五配位MgA重复了MD。结果表明,在位置约束平衡过程中,质子在离活性位点较远的D485的稳定性并没有增加(Fig. 9b)。为了消除源自力场的不稳定因素,在对D485质子进行限制的情况下重复MD模拟。在所有结果中,MgA-MgB的距离在平衡过程中增加到5A附近(Fig. 9c)。因此可以得出结论,对于回溯态的活性位点,质子化的D485不是一个稳定存在的状态,并且不能作为广义酸。所以,结果表明Pol II中的蛋白残基在裂解反应中不直接作为广义酸参与质子转移。
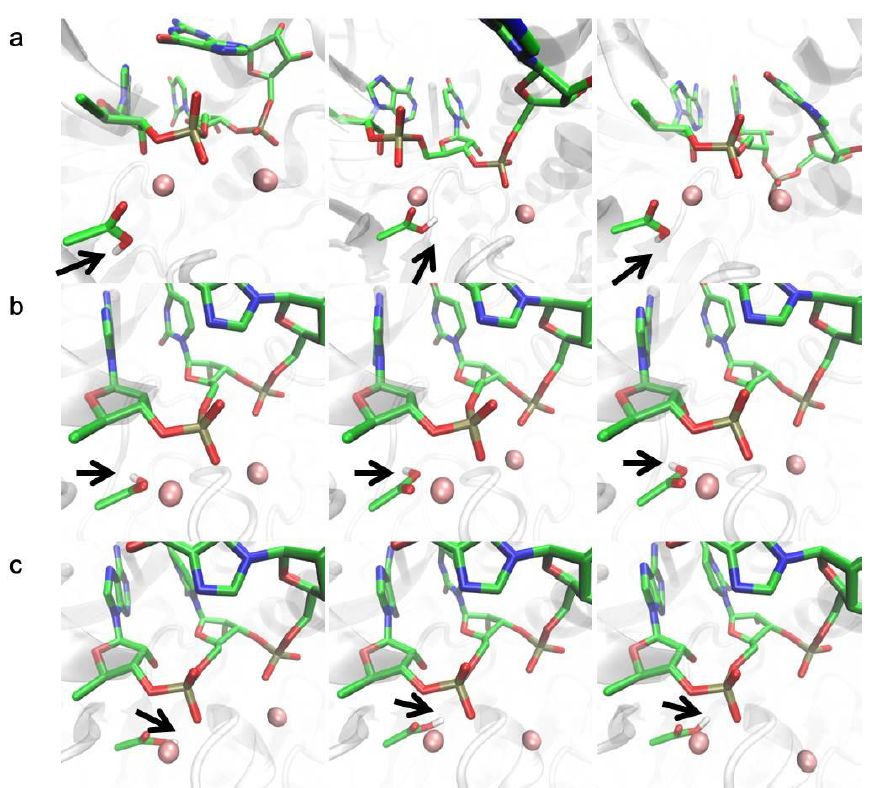
Fig. 9 质子化D485的三组模型MD模拟。a, 6配位MgA; b, 5配位MgA; c, 对5 配位MgA及D485质子进行限制。
图片来源Nature Catalysis
结语
这篇催化机制研究案例文章是非常典型的计算结合实验的工作。RNA聚合酶II内裂机制中,由于不确定参与反应的广义酸/碱,一直存在争议。研究工作在获得可靠的模型,确定QM区原子及选择反应坐标后,通过QM/MM-MD模拟找出了可行的RNA聚合酶II内裂机制,分析了磷酸O原子作为广义碱参与反应的过程,同时将其他的可能性进行逐一排除。而后根据计算结果,巧妙的设计了两个实验方案(改变核酸骨架影响磷酸位置和S代磷酸O原子),给出了有力的实验证据,对理论计算提出的反应机制进行了验证。从中我们可以学习到在对酶反应机制进行研究时,如何瞄准关键科学问题,在一开始构思合理的计算模型,使得计算结果能够阐明催化机制的同时,更好的配合实验或在一定程度上指导实验方案设计。但文中提出的反应机制能垒偏高(29kcal/mol),作者认为这与RNA聚合酶II内裂速率较慢相符(比受转录因子刺激时慢~400倍),并通过进一步计算,利用类似排除法的方式将其他可能反应途径进行排除。不过值得我们注意的是在反应途径能垒偏高时,同样要考虑到初始模型、反应坐标、所用QM方法和QM区选择等影响因素,确认计算的合理性,再进行实验验证。
参考文献
(1) Tse, C. K. M.; Xu, J.; Xu, L.; Sheong, F. K.; Wang, S.; Chow, H. Y.; Gao, X.; Li, X.; Cheung, P. P.-H.; Wang, D.; Zhang, Y.; Huang, X. Intrinsic cleavage of RNA polymerase II adopts a nucleobase-independent mechanism assisted by transcript phosphate. Nature Catalysis 2019, 2, 228-235.
(2) Udo, L.; Winfried, H. Transcriptional fidelity and proofreading in Archaea and implications for the mechanism of TFS-induced RNA cleavage. Mol. Microbiol. 2010, 52, 1133-1143.
(3) Samara, N. L.; Yang, W. Cation trafficking propels RNA hydrolysis. Nature Structural & Molecular Biology 2018, 25, 715-721.
(4) Mishanina, T. V.; Palo, M. Z.; Nayak, D.; Mooney, R. A.; Landick, R. Trigger loop of RNA polymerase is a positional, not acid-base, catalyst for both transcription and proofreading. Proceedings of the National Academy of Sciences of the United States of America 2017, 114, E5103.
(5) Lu, X.; Fang, D.; Ito, S.; Okamoto, Y.; Ovchinnikov, V.; Cui, Q. QM/MM free energy simulations: recent progress and challenges. Mol Simul 2016, 42, 1056-1078.
(6) Zhou, Y.; Wang, S.; Li, Y.; Zhang, Y. Born-Oppenheimer Ab Initio QM/MM Molecular Dynamics Simulations of Enzyme Reactions. Methods Enzymol. 2016, 577, 105-118.
内容来源自QMCLab,唯信计算整理发布