引言
瑞典科研团队通过分子对接对化学暗物质数据库筛选发现GPCR家族的A2A腺苷受体和D4多巴胺受体的活性小分子。
先睹为快
靶点
GPCR家族的A2A腺苷受体和D4多巴胺受体
筛选化合物库
化学暗物质数据库,从PubChem数据库收集非活性的可购买的lead-like分子(39655)
计算方法
分子对接
计算软件
DOCK3.6
计算流程
本文通过分子对接对化学暗物质数据库(39655)筛选发现G蛋白偶联受体(GPCR)的配体。针对与中枢神经系统疾病相关的A2A腺苷受体和D4多巴胺受体的晶体结构进行了分子对接筛选,选择对接打分前500名进行分析,然后针对A2AR和D4R分别选择35和18个化合物进行了实验评估。每个受体均发现了2个具有亚微摩尔级亲和力的配体。该研究为虚拟筛选挖掘暗化学物质数据库提供了一种有效的手段,有助于开发安全性更高的药物。

化学暗物质(Dark Chemical Matter,DCM)
化学暗物质(dark chemical matter,DCM)是指在大量生物活性实验中进行了测试仍未显示出生物活性的一类化合物。传统观念认为这类化合物不具有很好的成药性,不过越来越多研究发现这类分子一旦发现对某一靶点有活性,这类分子将会具有很好的选择性,因此off-target效应将会很弱,后期临床研究中发现毒副作用的概率将会大大降低,因此被认为是药物研发的重要起点。
基于结构的虚拟筛选
作者对PubChem和ZINC数据库中的DCM化合物进行了筛选,构建了共有39655个小分子化合物的DCM数据库。随后采用DOCK3.6对接程序对该数据库中可与A2AAR及D4R结合的化合物进行了虚拟筛选。使用打分函数对每个配合物进行评估,通过预测的结合能对对接的化合物进行排名,并对500个得分最高的分子(对应于筛选数据库的1.3%)进行了人工目测筛选。综合考虑化合物与结合位点的互补性、化学多样性,并考虑了评分功能中未包括的能量项(例如,受体解溶和配体内能),针对A2AAR及D4R分别选择了35和18个化合物购买,随后通过放射性配基结合分析以及生物活性验证进一步筛选最终得到4个活性化合物(A2AAR:2个,D4R:2个)。
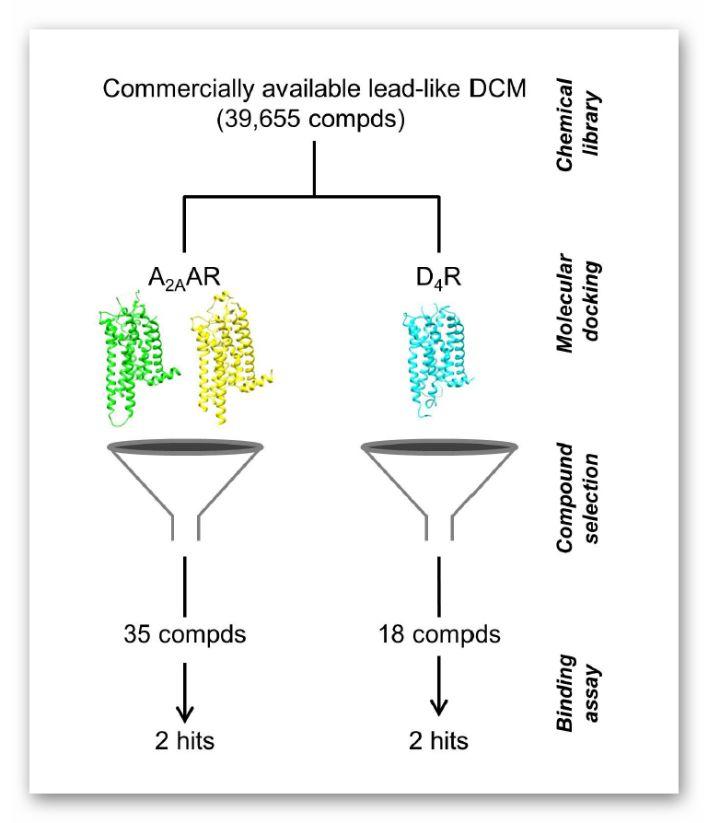
图1. 针对A2AAR和D4R的DCM基于结构的虚拟筛选流程示意图
图片来源JMC
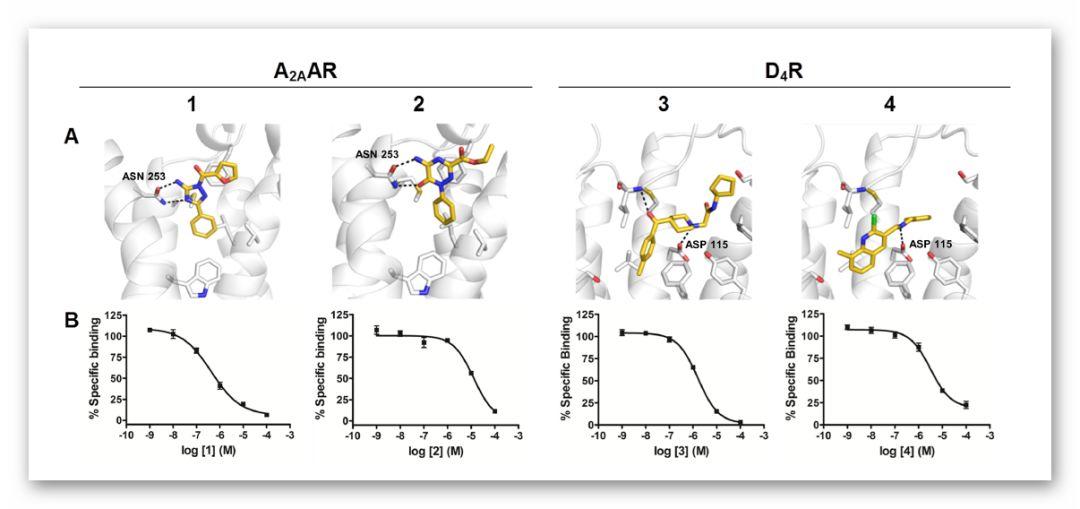
图2.化合物1~4与A2AAR和D4R的结合模式及实验数据
图片来源JMC
靶标选择性评估
作者通过检索PubChem和Uniprot数据库中化合物1~4的生物活性数据和靶标信息,对化合物活性图谱和蛋白质靶标进行了分析,化合物1~4在覆盖了362-527个靶标中均表现为非活性(图3)。为了研究GPCR靶标之间的一般选择性是否也包括其他腺苷和多巴胺受体,作者评估了化合物1~4与A2AAR和D4R不同亚型靶标的结合。结果表明,化合物1和2对A2A 和A1AR具有相似活性,但是在D4R中,化合物3和4相对D3R,具有26和3倍的选择性。该结果表明应用基于结构的虚拟筛选方法对DCM数据库筛选所得小分子具有靶标的高选择性,并可能导致候选药物具有更可靠的安全性。
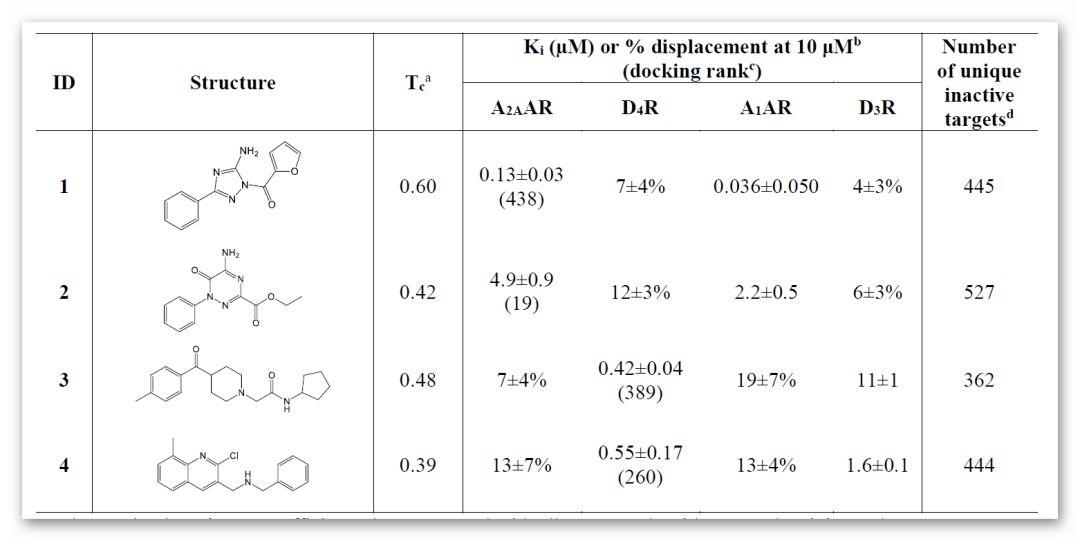
图3. 已发现A2AAR和D4R配体的实验数据和最接近的已知配体的相似性以及在PubChem生物活性数据库中报告的化合物没有活性的独特蛋白质的数量
图片来源JMC
参考文献:
Ballante F, Rudling A, Zeifman A, et al. Docking finds GPCR ligands in dark chemical matter[J]. J. Med. Chem. 2020, 63, 613−620. DOI: 10.1021/acs.jmedchem.9b01560