
引言
在药物发现过程中,准确预测小分子的疏水性是一个重要的问题。细胞和整个人体内部存在许多不同的化学环境。例如,药物必须穿过疏水性细胞膜达到其细胞内靶标,疏水性是药物与蛋白质结合的重要驱动力。分子动力学(MD)仿真可用于计算药物与蛋白质结合、以及溶剂化的小分子的自由能,缺点是计算成本高昂。在药物研发过程中也使用机器学习(ML)和设计经验,但这些方法依赖于实验数据,适用范围有限。我们计算了从水到环己烷转移的15000个小分子的迁移自由能,并作为数据集训练可预测迁移自由能的机器学习模型。研究表明,空间图神经网络模型实现了最高的准确性,其次是3D卷积神经网络,基于化学指纹的浅层模型的准确性远低于前两者。最佳模型与MD计算相比,平均绝对误差约为4 kJ / mol。此外,研究显示采用多任务学习模型可改善预测效果。
研究背景
分子动力学(MD)模拟和机器学习(ML)已广泛用于药物发现中。研究的应用范围涵盖了蛋白质-配体结合,蛋白质-蛋白质相互作用等多个研究领域。MD模拟的优势在于可以模拟原子细节和热力学性质,高昂的计算成本限制了计算的小分子的数量。使用实验数据训练模型并提取分子特征后,ML模型可以准确预测小分子性质。可是训练数据集可能存在稀疏的现象,这限制了模型的适用范围。因此,我们需要构建能够利用MD和ML方法的优势的模型。例如,我们可以用ML来代替昂贵的自由能计算。
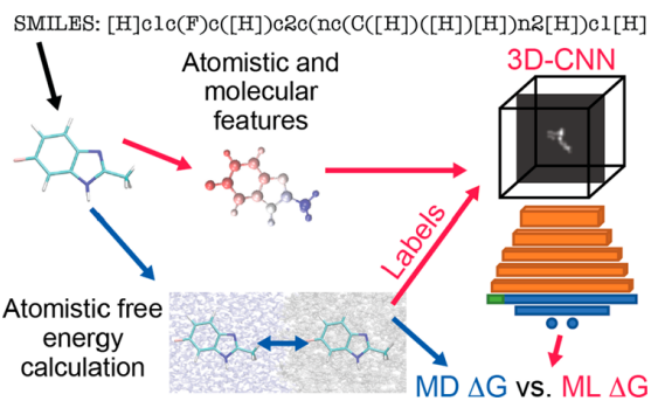
图1. 分子模拟和深度卷积网络的流程图
图片来源于JCIM
疏水性是关键的分子性质,例如药物疏水性是药代动力学建模的重要参数。药物与血液蛋白非特异性结合是药物失效的关键原因,传统药物研究中,使用小分子在有机溶剂和水和有机溶剂的分配系数(logP)评估药物透膜能力。有许多测定logP的实验方案,并且有许多计算疏水性的模型(即使用clogP计算logP)。但是,它们通常采用的是拟合实验数据的方式。自由能是MD计算得出的属性,它与给定状态的概率成正比,并与许多实验可观测值(如浓度)成正比。预测结合时需计算自由能差,必须“采样”(或探索)体系的所有相关构象。因此,预测分子的自由能变化并非易事。MD模拟已被广泛用于研究跨脂质双层中小分子分配与渗透。为了克服MD计算量大的缺点,可以采用简化模型和增强的采样技术,加快计算速度。ML已广泛用于药物研发中。分子的特征表示和获取有关分子样本是机器学习的两个主要挑战。研究表明,小分子3D空间信息作为小分子特征的模型效果优于传统的分子特征的模型。提取MD数据用作预测分子溶剂化的特征可以提升模型预测效果。我们构造了ML模型来预测原子MD的自由能。MD模拟用于计算15000个转移的小分子自由能,以训练模型。MD和ML的示意图是如图1所示。从MD模拟中,我们提取了3D原子特征,用于训练ML模型,以预测从水到界面(ΔGWater‑Interface或ΔGW-I)和水到环己烷(ΔGWater-Cyclohexane或ΔGW-C)的转移的自由能。
结果
1. 分子动力学自由能计算
使用MD模拟构建用于ML预测的数据集,用来计算从水到环己烷的小分子转移的自由能。图2展示了我们用于通过MD计算15000个分子的迁移自由能的过程。对于基础分子,我们首先在整个水-环己烷系统中使用umbrella sampling采样(图2A)。然后,我们使用相对热力学积分(TI)自由能计算,在三种化学环境(水,界面和环己烷)中将基本分子微扰成10个不同的分子(一个官能团不同)(图2B)。图2显示了如何使用umbrella sampling和TI计算转移的相对自由能。其中的垂直箭头表示使用TI计算的环己烷与环己醇的自由能差(图2A)。自由能曲线表明,几乎所有分子在界面处都具有最小自由能(图2C)。因为它们是类似药物的小分子片段,所以大多数是双亲性的。与界面处的情形相反,大约一半的分子更喜欢水,另一半的分子更喜欢环己烷,能量最大+40,最小-40 kJ / mol(图2C)。图2D展示了具有不同迁移自由能的代表性分子。
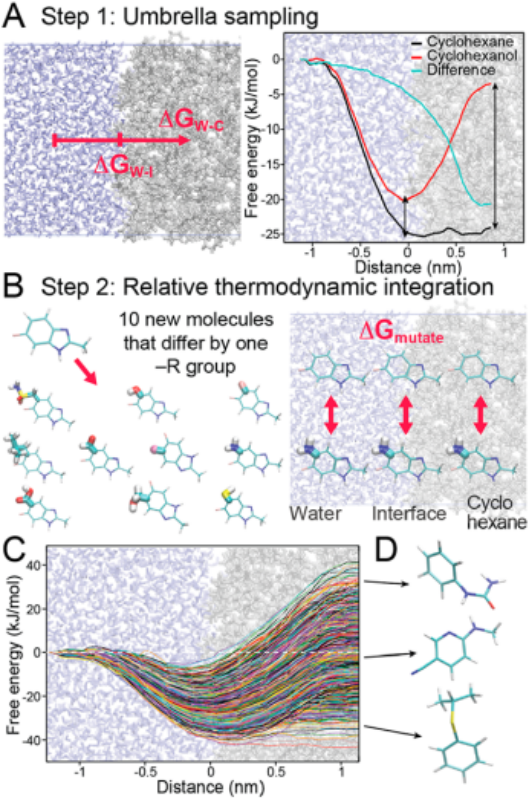
图2. MD计算水、界面、环己烷三个位置分子的自由能
图片来源于JCIM
2. 小分子性质与迁移自由能的关系
为了分析影响分子分配系数的因素,我们比较了分子描述符和自由能。图3中显示了所有15000个分子的ΔGW-C和ΔGW-I的散点图,两者相关性较弱。我们依据模拟轨迹计算了不同化学环境中的分子特性。在图3A-D中,我们绘制了每个分子的自由能的散点图并按照分子性质着色。分子形成的氢键平均数如图3A所示。与环己烷相比,分子更倾向在水中形成更多氢键。在交界面处,分子可以对齐并保持氢键,这种倾向就变得不太明显。我们还绘制了每个分子的回转半径,用来评估分子的大小。图3C是每个分子的碳原子数和自由能的散点图,它的趋势与旋转半径的趋势相似。从图中可以发现,较小的分子更喜欢水上界面。分子的大小对分子在水与环己烷之间的转移影响较小。图3D绘制了平均分子偶极子和自由能的散点图,它与氢键数具有相似的趋势。偶极子低的分子更偏好水,但对界面自由能的影响较小。我们使用RDKit计算每个分子的clogP,并与我们对ΔGW-C和ΔGW-I的预测对比(图3E)。结果表明我们的预测结果与clogP具有中等相关性。对于具有高clogP(非常疏水)的分子,ΔGW-C和ΔGW-I预测都与clogP拟合得很好。与clogP相比,随着分子变得更加亲水(logP越小),与预测值的相关性减弱。这表明对于一个分子,它具有一个clogP,但可以具有两个差别很大的ΔGW-C和ΔGW-I。对于低logP的分子,传统上将这些分子视为疏水的,但是我们的结果证明这些分子仍可被界面吸引。我们绘制ΔGW-C和ΔGW-I的散点图,并依据clogP着色(图3F)显示,clogP与我们两个指标都相关。
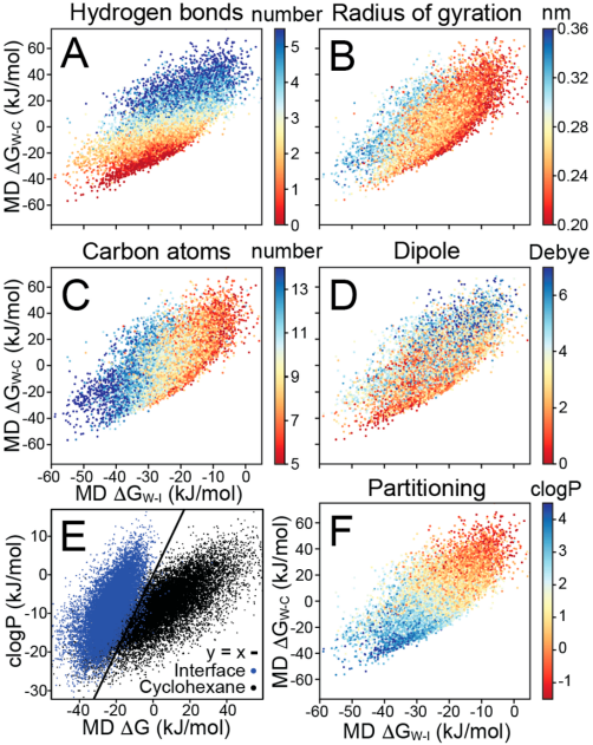
图3. 迁移自由能和分子描述符的关系
图片来源于JCIM
3. 用于预测迁移自由能的机器学习模型
从15000个自由能计算中,我们测试了许多ML方法来预测迁移自由能。对于简单的浅层学习模型,我们选用岭回归的2D Morgan分子指纹进行了测试,还构建了更复杂的3D-CNN和SG-CNN(图4)。我们在模型训练中使用按照骨架分割和与随机分割两种方法划分数据集。表1显示了不同ML模型预测ΔGW-C和ΔGW-I的结果。总体而言,对ΔGW-C的预测具有比ΔGW-I预测更高的相关性,但也具有更高的MAE。这可能是因为ΔGW-C的样本间差异比ΔGW-I大得多,导致更大的误差。加入了MD的特征参与训练后效果有所提升(表2)。总体上看,效果最佳的模型是SG-CNN,其次是3D-CNN,浅层网络的ECFP模型的表现最差。我们还训练了多任务模型,一个模型同时预测分子在环己烷和界面处的迁移自由能。与单独预测环己烷环境的模型,或者单独预测界面环境的模型相比,具有很好的拟合效果,更少的离群点(图5)。
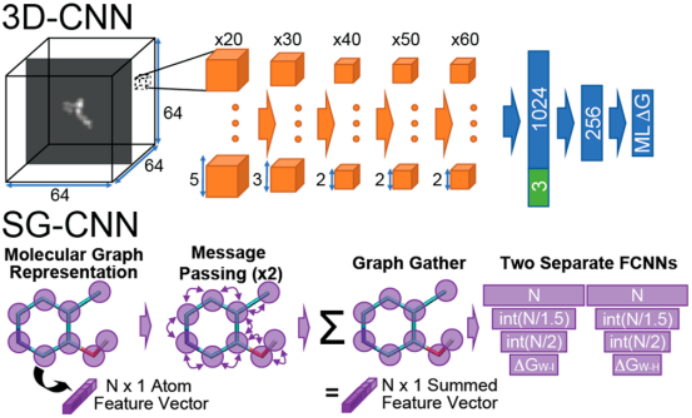
图4. 用来预测自由能的3D-CNN和SC-CNN的网络架构
图片来源于JCIM
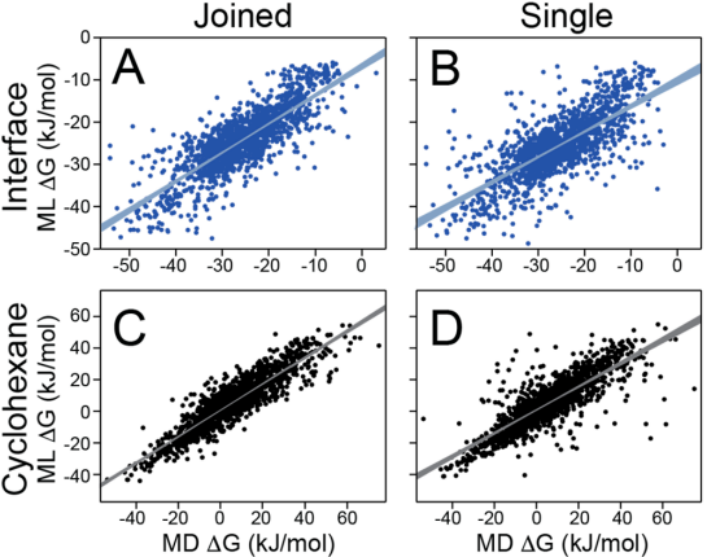
图5. 多任务学习的预测值、单模型的预测值和MD的数据对比。(A)多任务预测的界面处的自由能和MD结果的关系;(B)单模型预测界面处的自由能和MD结果的关系;(C)多任务预测的环己烷中的自由能和MD结果的关系;(D)多任务预测的环己烷中的自由能和MD结果的关系
图片来源于JCIM
表1. 三个模型使用MD的特征预测ΔGW-I和ΔGW-C的结果
表格来源于JCIM
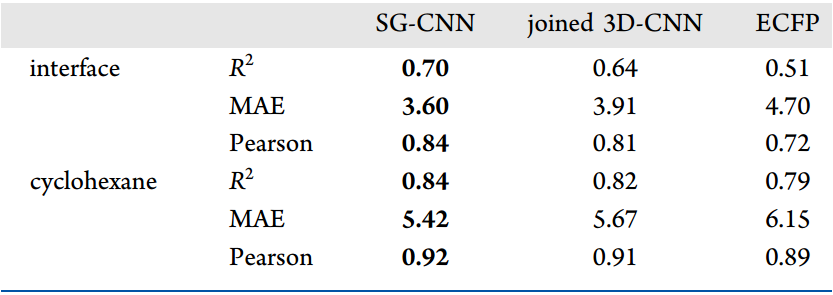
表2. 三个模型不含MD的特征时预测ΔGW-I和ΔGW-C的结果
表格来源于JCIM
4. 机器学习模型对上市药物的预测
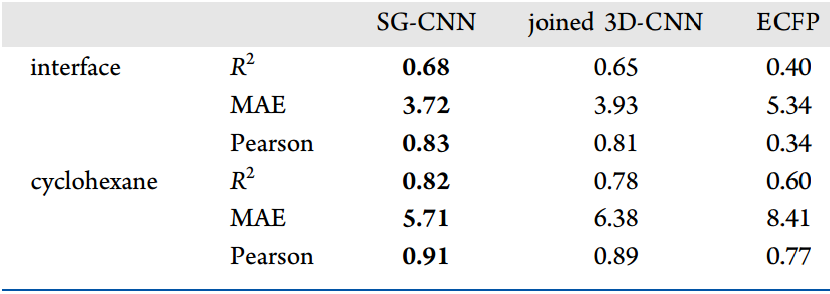
对于模型通用性的测试,我们使用在小分子数据集上训练的ML模型,在批准上市的药物分子的子集做测试。模型能够准确预测药物测试集分子的ΔGW-I(表3和表4)。但是模型对ΔGW-C的预测明显变差。于是我们使用MD派生的特征改进了模型。ECFP方法在上市药物的预测表现要差得多,这表明它无法学习到重要的化学信息,说明模型没有通用性。3D-CNN在上市药物的测试中表现最佳,表明其具有更好的通用性。
表3. 三个模型在上市药物分子子集上的预测结果
表格来源于JCIM
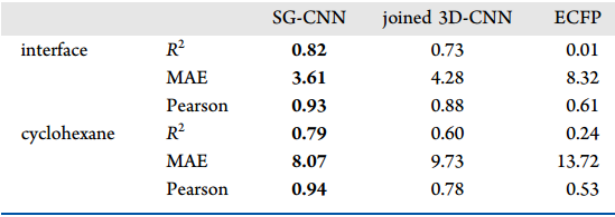
表4. 加入MD派生特征后模型在上市药物分子子集上的预测结果
表格来源于JCIM
讨论
这项工作是为了测试使用ML从MD模拟数据预测复杂的热力学变量的可行性。为此,我们为从水中转移到界面和本体环己烷中的15000个小分子库生成了自由能。接着我们将这些自由能用作标记来训练3D-CNN,结果表明,模型能够预测迁移自由能,模型的精度接近MD的计算误差。总体而言,上市药物数据集umbrella sampling的误差为4.6 kJ / mol。深度学习在界面预测中的MAE稍低,但在环己烷中则差得多。分子动力学模拟和数学模型还表明分子性质可能与一种热力学性质密切相关。在计算资源上,我们首先要计算出自由能的数据,然后使用机器学习模型来预测小分子的自由能。对于小分子,每项采样计算的MD模拟花费约200个CPU小时,计算15000个自由能数据则花费约300万个CPU小时。在GPU上生成样本特征后,大约需要7分钟才能做出15000个预测。平均0.03 s才能预测出一个样本。考虑到CPU与GPU,目前我们还没有尝试优化这两种方法。虽然机器学习模型显然比机器学习模型快几个数量级,但也需要大量预先计算的自由能来进行训练。使用MD模拟数据训练ML模型,预测小分子的迁移自由能。仿真结果揭示了小分子性质对小分子在脂水中分布的影响。几乎所有的小分子都更喜欢水或环己烷的界面。模型选取上,SG-CNN的效果略胜于3D-CNN,两者均明显胜过ECFP模型。添加MD相关数据能改善模型效果。我们的结果和数据集将在分子建模、药物发现和材料科学中发挥重要作用。
参考文献
W. F. Drew Bennett, Stewart He, Camille L. Bilodeau, Derek Jones, Delin Sun, Hyojin Kim, Jonathan E. Allen, Felice C. Lightstone, and Helgi I. Ingólfsson, Predicting Small Molecule Transfer Free Energies by Combining Molecular Dynamics Simulations and Deep Learning, J. Chem. Inf. Model. 2020, 60, 11, 5375-5381. DOI: 10.1021/acs.jcim.0c00318.