Drug Design Data Resource: D3R
药物设计数据资源(drug design data resource,简称为D3R)旨在通过高质量的蛋白质配体复合物数据库和标准化的工作流,以及举办高质量的竞赛,来推进计算机辅助药物技术的发展。D3R竞赛中的基准测试集中包含高质量的结构以及来源于文献的可靠的结合能数据。这些数据为CADD技术提供了验证,校准和开发虚拟筛选(VS)模型的绝佳机会。
最近的D3R Grand Challenge 4(GC4)挑战赛于2018年9月4日至12月4日间举行。此次竞赛涵盖了两种不同的蛋白质靶标:组织蛋白酶S(CatS)和β分泌酶1(BACE)。竞赛由两个部分构成,第一部分具有两个子任务,即阶段1a和阶段1b。在阶段1a中,参与者被要求预测结合模式,对亲和力进行排名并估计BACE配体结合自由能。阶段1a之后,阶段1b给出了蛋白质受体结构,参与者被要求预测20个BACE配体的晶体学结合模式。GC4竞赛的第二部分包含BACE和CatS化合物的亲和力排名和自由能挑战,参与者可以利用已经给出的BACE配体复合物结构。
蓬勃发展的生物大数据集,高性能计算集群(HPCC),使得机器学习成为生物大分子数据分析和预测中的新兴技术,但是基于机器学习的打分函数的准确性在很大程度上取决于它们是否能够捕获蛋白质-配体相互作用中的物理和化学信息。由于3D大分子的自由度数目过多以及原子在不同结构中的变化,在深度学习网络中直接使用三维(3D)生物分子结构计算量会非常巨大。因此,迫切需要开发生物大分子新型表示形式。

MathDL比赛结果
本文介绍了深度学习模型- MathDL,并介绍了其在GC4上的测试性能。这项测试涉及了β-分泌酶1(BACE)的结合模式预测,亲和力排名和自由能预测,组织蛋白酶(CatS)的亲和力排名和自由能估计。作者使用微分几何,代数图和/或代数拓扑,开发出了一套先进的数学工具,可以准确地将高维度的物理/化学相互作用编码为低维度表示形式。
总体而言,MathDL模型在BACE配体的结合模式预测中排名第一。此外,在460种CatS化合物的亲和力排名上,MathDL模型的皮尔森相关性系数打分最高;在39个CatS分子的自由能集上获得了最小的中心均方根误差。
从GC2竞赛起,作者的团队就参加了相关的竞赛。GC4之前,在RMSE方面,作者的结果与其他参与者相比没有竞争力。在GC2和GC3上,作者的平均RMSD值分别为6.03Å和3.78Å。对接方法有进步,但其准确性仍落后于GC3上的顶级论文。在GC4中,作者的对接方案不是像以前挑战中那样依赖于Autodock Vina 和GLIDE 等对接程序,而是由高级数学表示和复杂的深度学习架构驱动的。因此,MathML在结合模式预测任务上取得了很好的成绩。
GC4中所有结合模式预测仅针对BACE配体,并且分为两个阶段,即阶段1a和阶段1b。在阶段1a中,主办方提供了20个配体的SMILES字符串,BACE蛋白的FASTA序列以及用于参考的蛋白结构(PDBID:5ygx,链A)。在阶段1a结束后,阶段1b提供了20种配体以用于复合物结构的结合模式预测。结合模式预测任务有两个评估指标,即在预测的结构和晶体结构之间的所有RMSD值上计算的中值和均值。
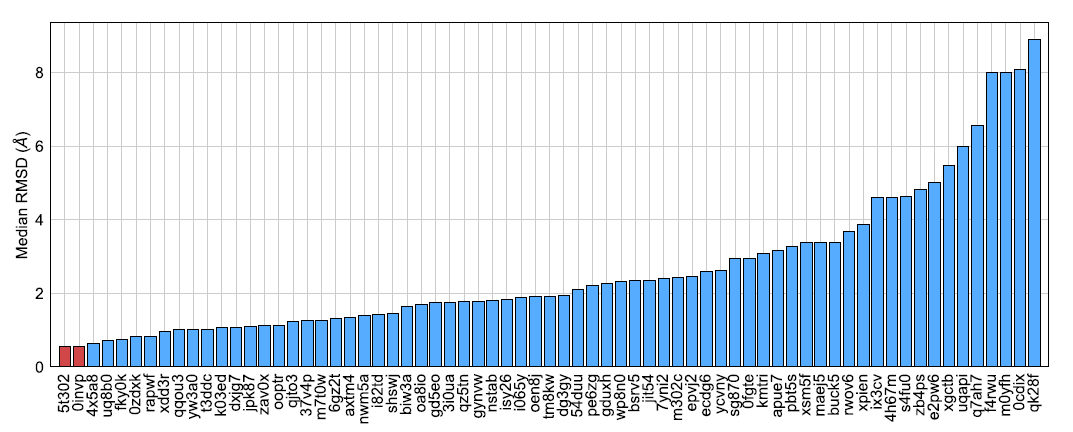
图1. BACE数据集的姿态预测挑战
图片来源Journal of Computer-Aided Molecular Design
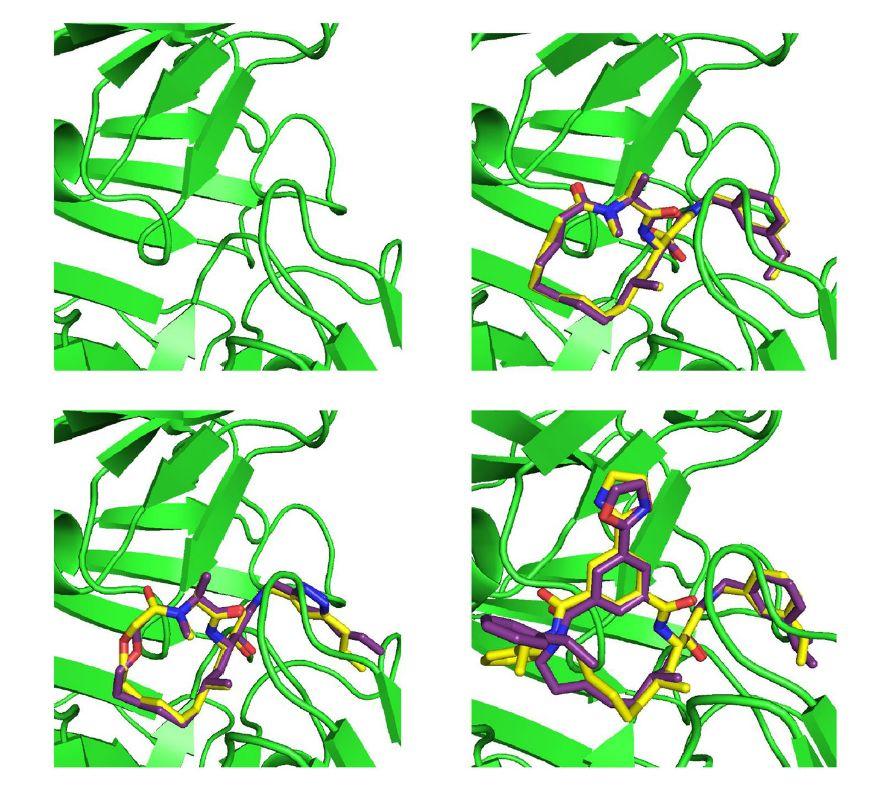
图2. MathGAN 的对接模型
图片来源Journal of Computer-Aided Molecular Design
在BACE亲和力预测中,作者的结果不在前三名中,仅落后于排名前三的两个团队BP2是用于CatS和BACE数据集的结合亲和力预测的最佳模型。BP2具有出色的预测性能,因为它结合了代数拓扑,微分几何和图论特征,涵盖了物理和生物学特性的最重要的方面。DM4是用于CatS结合模式预测的最佳模型,在CatS姿态预测的阶段1B挑战中,其平均RMSD为1.8Å。
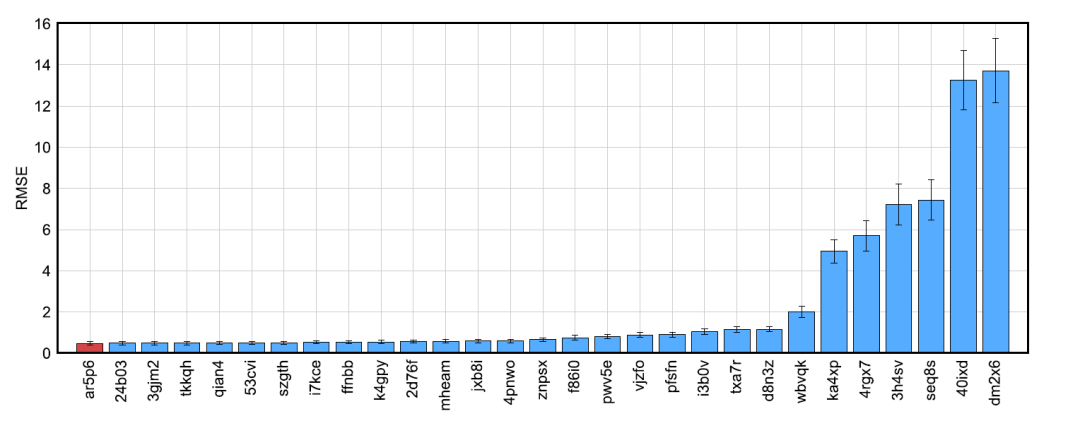
图3. CatS 数据集上的测试得分
图片来源Journal of Computer-Aided Molecular Design
MathDL方法
(1)MathDL能量预测模型:
作者将数学特征与卷积神经网络集成在一起,形成了强大的机器学习预测模型。CNN属于人工神经网络,由许多卷积层和几个全连接层组成,在图像识别和计算机视觉分析中取得了很大的成功。
作者使用的模型为前馈神经网络,主要特征为将当前层中的信息进行线性组合,通过激活函数进行非线性处理,然后发送到下一层。CNN模型的预测能力取决于离散卷积算子在空间维度上的局部交互作用的表征。CNN网络中特征输入的选择产生了结合能预测模型的变体。在GC4中,作者使用了两种不同的模型。第一种,网络中采用了代数拓扑和微分几何特征的组合,此模型命名为BP1。第二种,将代数拓扑,微分几何和代数图表示综合在一起,此模型命名为BP2。
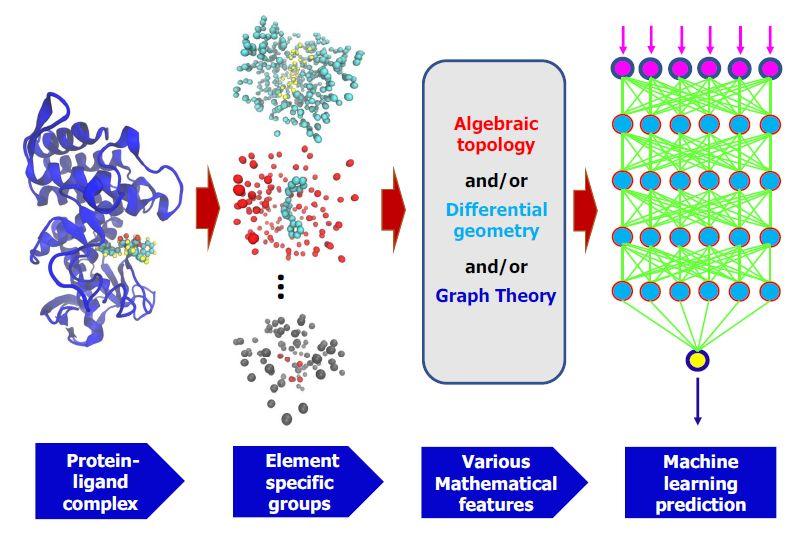
图4. MathDL能量预测模型
图片来源Journal of Computer-Aided Molecular Design
(2)MathDeep对接模型:
作者提出了一种新型的pose生成方案(MGAN),它使用高级数学表示GAN。GAN是一种深度学习模型,它由学习数据分布的生成器G和鉴别器D组成。从D反馈迭代地改进G模型,直到D无法分辨训练集结构信息和D之间的差异。通过不同数学的不同组合,作者得到了几种对接模型。具体来说,如果DL网络中G和D仅使用高等数学中的代数拓扑部分,则将其命名为对接模型DM1;当GAN模型仅包含代数图时,命名为DM2;仅包含微分几何时,命名为 DM3对接模型;DM4模型则由代数拓扑,代数图和微分几何构造综合组成。
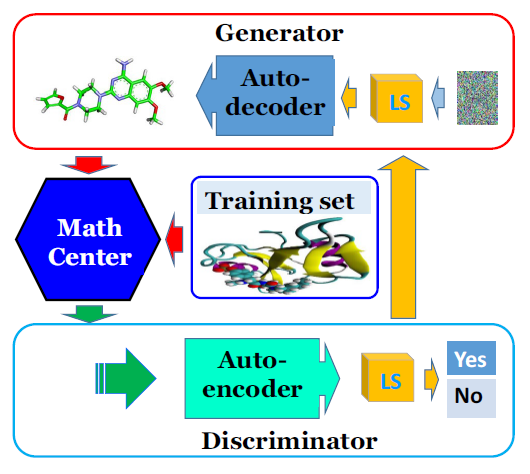
图5:MathDeep对接模型
图片来源Journal of Computer-Aided Molecular Design
总结
本文论述了深度学习模型(MathDL)在GC4竞赛上的表现,包括结合模式预测、亲和力排名和自由能预测。总体而言,MathDL模型在1a阶段的结合模式预测,组织蛋白酶配体的亲和力排名预测和自由能预测中排名第一。与之前的D3R挑战(即D3R GC2和D3R GC3)相比,作者GC4中进行了两项改进。第一个改进是结合模式预测方面,第二个改进是亲和力排名预测方面。MathDL模型的自动化程度非常高的。如果有足够的计算机资源,MathDL模型可以在一周左右的时间内完成所有GC4竞赛任务。
参考文献
Nguyen, D.D., et al., MathDL: mathematical deep learning for D3R Grand Challenge 4. J Comput Aided Mol Des, 2019. DOI: 10.1007/s10822-019-00237-5