引言
蛋白与肽段的相互作用(PpIs)介导了大量的蛋白-蛋白相互作用,阐明蛋白-肽复合物的结构细节对于理解肽疗法和蛋白-肽识别基础的分子机制至关重要。但是,PpIs的高度动态性和瞬时性极大地阻碍了蛋白-肽复合物结构的实验表征,因此,多种蛋白-肽对接方法被开发出来以预测蛋白-肽复合物的结合模式,但对于这些对接程序的优点和局限性缺乏系统性评估。
简介
浙江大学侯廷军团队与中南大学曹东升团队联合构建了基准数据集PepSet,并提出用界面处配体的RMSD(IL_RMSD)来衡量预测蛋白-肽模型的质量,对3个蛋白-蛋白对接程序(ZDOCK, FRODOCK, HawkDock),3个小分子对接程序(GOLD, Surflex-Dock, AutoDock vina)以及8个蛋白-肽对接程序(GalaxyPepDock, MDockPep, HPEPDOCK, CABS-dock, pepATTRACT, DINC, AutoDock CrankPep, HADDOCK)共14个对接程序的性能进行了系统评估。

蛋白-肽复合物基准数据集PepSet
PepSet是从PepBDB中提取的,收集了肽长度在5-20个氨基酸的所有蛋白-肽复合物并根据序列同一性等标准最终得到185个PDB结构。根据结合结构中肽的构象与其理想伸展构象或螺旋构象之间骨架原子的RMSD,将PepSet分为以下三类:

根据该标准,基准数据集的所有复合物中,132个属于简单,28个属于适中,25个属于困难级别。为了对接方便,使用Python库的PeptideBulider手动生成了肽的3个理想初始构象。受体蛋白中的所有非标准氨基酸都被修饰为标准氨基酸。该基准数据集可从http://cadd.zju.edu.cn/pepset下载得到。图1显示了数据集中肽的分布情况。
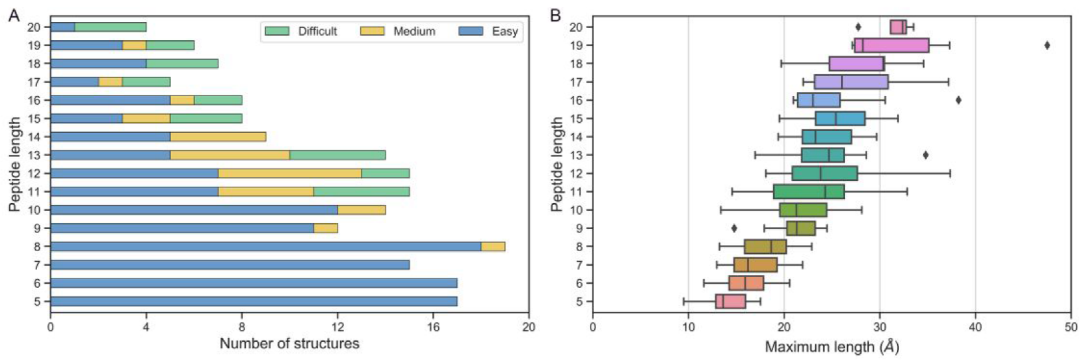
图1. PepSet中复合物按肽长度和难度的分布图.
图片来源:JCTC
评估指标
蛋白-肽结构质量通过界面处配体的RMSD(IL_RMSD)和天然接触分数(fnat)来衡量。IL_RMSD由将距肽段10Å内的蛋白残基进行优化重叠后,基于距蛋白10Å内肽残基的骨架原子计算得到,通过ProFit程序完成RMSD计算。另外,若蛋白和肽中的两个残基的任一重原子在4Å以内,则定义为接触,天然接触分数fnat被用于评估侧链的质量。软件评估标准如下:

在上述标准下,对接成功率被定义为在前N个模型中具有至少一个接近天然构象的预测的案例的百分比。例如,若在前100个预测中找到185个复合物中有74个接近天然构象,则“前100个”级别的成功率为74/185=40%。
基准测试结果
当前的蛋白-肽对接算法可以大致分为基于模板的对接和无模板对接两类,基于模板的对接如GalaxyPepDock使用类似模板的复合物结构来预测蛋白-肽复合物的结合结构。无模板对接根据结合位点是否已知分为全局对接和局部对接,前者会对蛋白的整个表面执行详尽的搜索以捕获和肽的结合位点及模式,后者则是在用户定义的位点(格点盒子)附近搜索。
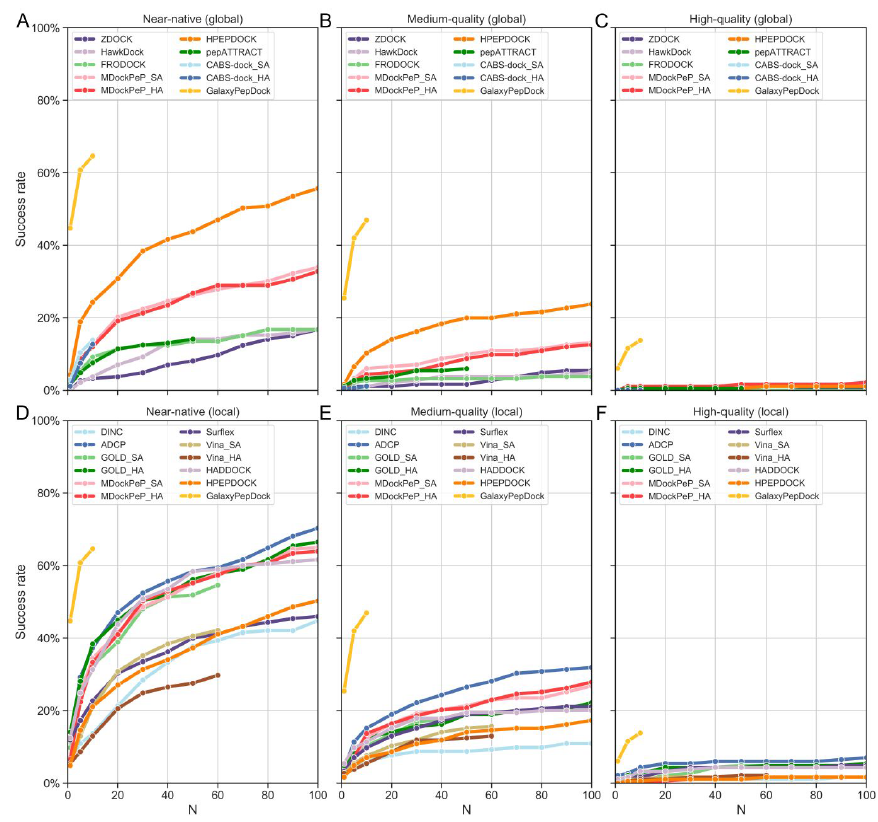
图2. 前N个预测中,全局(A-C)和局部(D-F)对接程序在三种标准下的成功率
图片来源:JCTC
图2显示了在整个数据集上测试全局和局部对接程序的成功率。GalaxyPepDock是基于模板的对接方法,其性能明显优于PepSet数据集上的任何无模板对接方法。对于全局对接,HPEPDOCK表现最好且计算效率更高,在前1、前10和前100级别的成功率分别为4.3%,24.3%和55.7%。其次是CABS-dock_SA,CABS-dock_HA,MDockPeP_SA,MDockPeP_HA,FPODOCK,pepATTRACT,HawkDock和ZDOCK。对于后三种对接程序,每个肽的三个理想构象都通过刚性对接算法对接,因此无法很好地处理肽的柔性。另外,由HPEPDOCK对接的初始肽构象数量多达1000个,这可能是其成功率最高的关键因素之一,也为其他对接程序提高准确性提供了启发性的策略。需要注意的是,所有方法都无法在高质量预测中提供良好的测试结果。
对于局部对接而言,ADCP(AutoDock CrankPep)达到了最佳预测效果,在前1、前10和前100级别的成功率分别为11.9%,37.3%和70.3%。其他对接程序排序为:GOLD_HA> MDockPeP_SA(局部)≈ MDockPeP_HA(局部)> HADDOCK> GOLD_SA> Surflex≈Vina_SA> HPEPDOCK(局部)> DINC> Vina_HA。
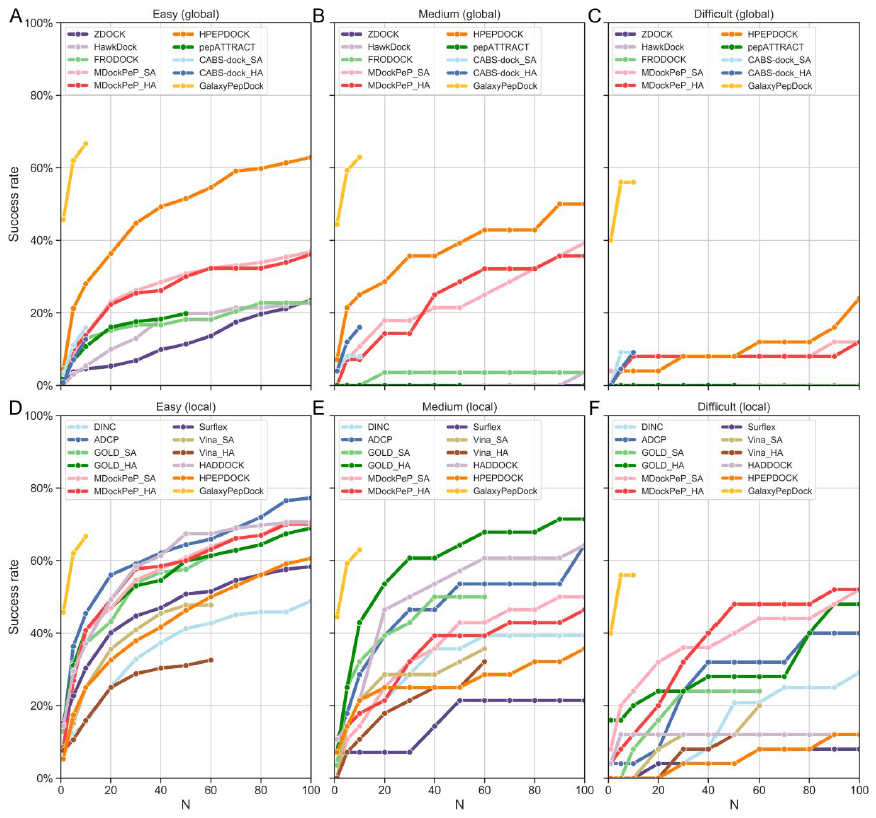
图3. 前N个预测中,全局(A-C)和局部(D-F)对接程序在三类子集上的成功率
图片来源:JCTC
为了研究肽柔性的影响,将基准数据集分为简单,适中和困难三类。对接结果如图3所示。随着肽柔性的增加,对接程序的性能逐渐降低。在全局对接中,HPEPDOCK的性能十分强大,在3个类别中都显示出最佳性能。在本地对接中,ADCP、GOLD_HA和MDockPeP_SA分别在简单、适中和困难子集中获得最佳性能。
总结
本文构建了一个包含185个蛋白-肽复合物的基准数据集PePSet并提出一个新的评估标准IL_RMSD用于评估14种对接程序。总体而言,蛋白-肽对接程序比蛋白-蛋白和蛋白-小分子对接程序具有更好地性能,生成足够多的初始肽构象是改善对接性能的有效策略。全局对接中,HPEPDOCK显示出最佳性能,局部对接中,ADCP在整个数据集上表现最佳。
参考文献
Weng G, Gao J, Wang Z, et al. Comprehensive Evaluation of Fourteen Docking Programs on Protein-Peptide Complexes. J Chem Theory Comput. May 6 2020. https://doi.org/10.1021/acs.jctc.9b01208