引言
基于结构来预测蛋白质与其他生物分子之间的相互作用仍然是生物学中的热点问题。蛋白质结构的surface表征已被长期用于研究蛋白相互作用,并且已成为研究蛋白质-溶剂静电相互作用的首选。几何深度学习是一个新兴领域,它成功的将基于图像的深度神经网络体系结构扩展到几何数据方面(例如蛋白表面)。

基于几何学习的蛋白表面相互作用指纹MaSIF
瑞士联邦理工学院与瑞士生物信息学研究所的B. E. Correia教授在Nature Methods发表了MaSIF,一个基于几何深度学习的框架,即利用几何深度学习来学习蛋白质分子表面中的相互作用指纹。基于几何深度学习的蛋白表面相互作用指纹框架MaSIF(molecular surface interaction fingerprint)已在Github开源,地址是https://github.com/lpdi-epfl/masif。实现流程如下:
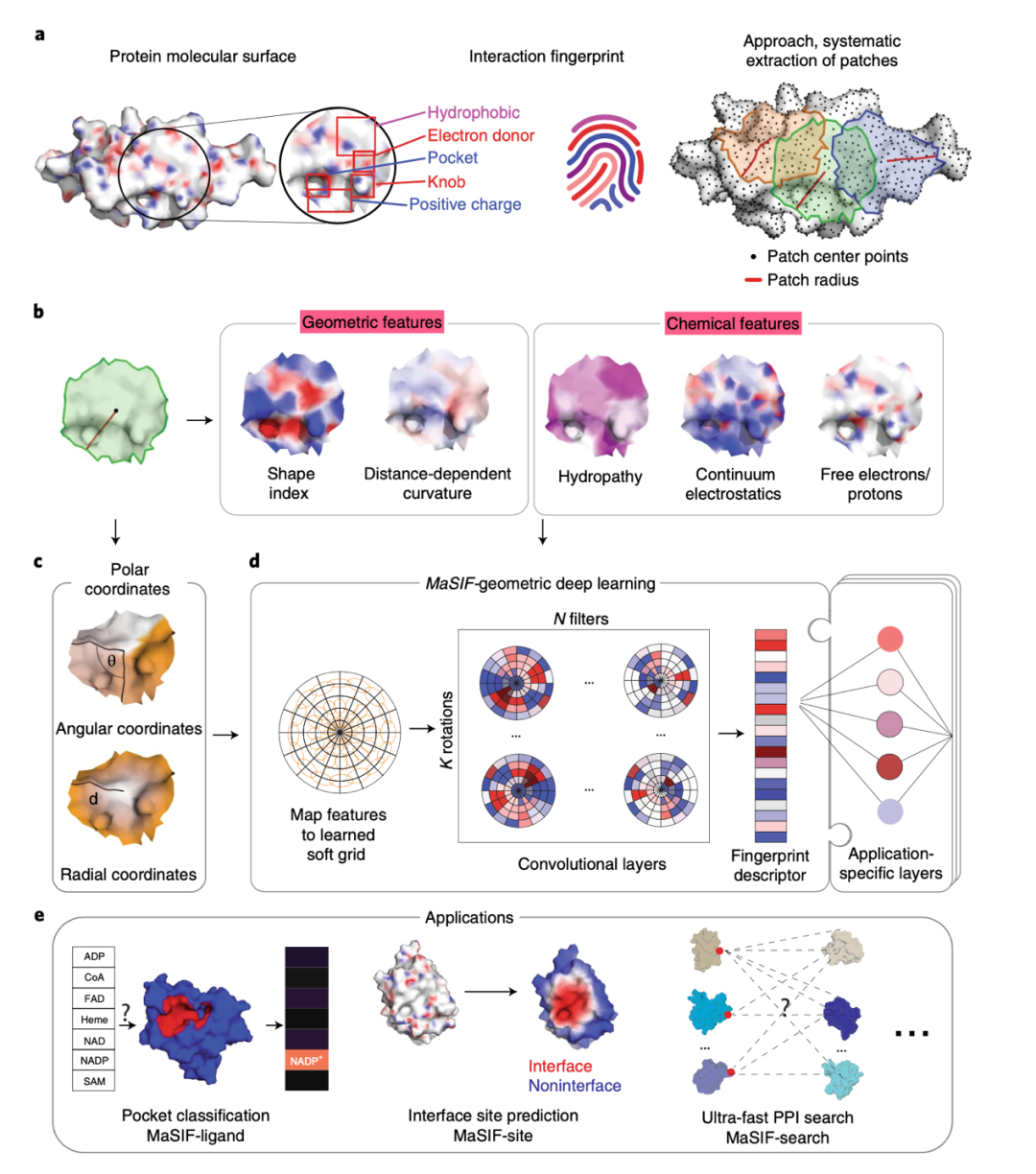
图 1.MaSIF的框架
图片来源:NATURE
首先从一个蛋白结构中,作者将其分子表面进行离散化处理,形成网格,并将几何和化学特征分配给网格中的每个点(顶点)。对于网格每个顶点,提取曲面几何半径为r =9Å或r =12Å的面积作为一个patch。
其次对于patch中的每个顶点,两个几何描述符(形状指数和与距离有关的曲率)和三个化学描述符(亲水指数,连续性静电以及自由电子和质子供体的位置)被计算出用于输入特征。patch内的顶点使用极坐标进行确定。几何特征(形状指数和与距离有关的曲率)和极坐标隐式描述了蛋白表面的几何结构(例如,蛋白表面凹穴的“深度”)。
然后MaSIF使用极坐标对这些输入特征进行空间定位,使用一个几何深度神经网络进行训练。该神经网络由一个或多个按顺序应用的层组成;该体系结构的一个关键组成部分是几何卷积,它将将经典卷积推广到曲面,并对patch进行操作。在极坐标系中,我们构造了一个高斯核系统,高斯核将平均patch的顶点特征,并生成维数固定的输出,该输出与一组滤波器相关。一组卷积层被应用于极坐标网格层的输出。其学习过程包括最小化局部核参数集,针对特定应用进行数据训练。这个框架为patch创建了描述符,这些patch可以在神经网络结构中进一步处理。
MaSIF的应用
(1)MaSIF-ligand,蛋白质口袋中的配体预测。
(2)MaSIF-site,蛋白质-蛋白质相互作用位点预测。
(3)MaSIF-search,超快扫描预测蛋白质-蛋白质复合物。
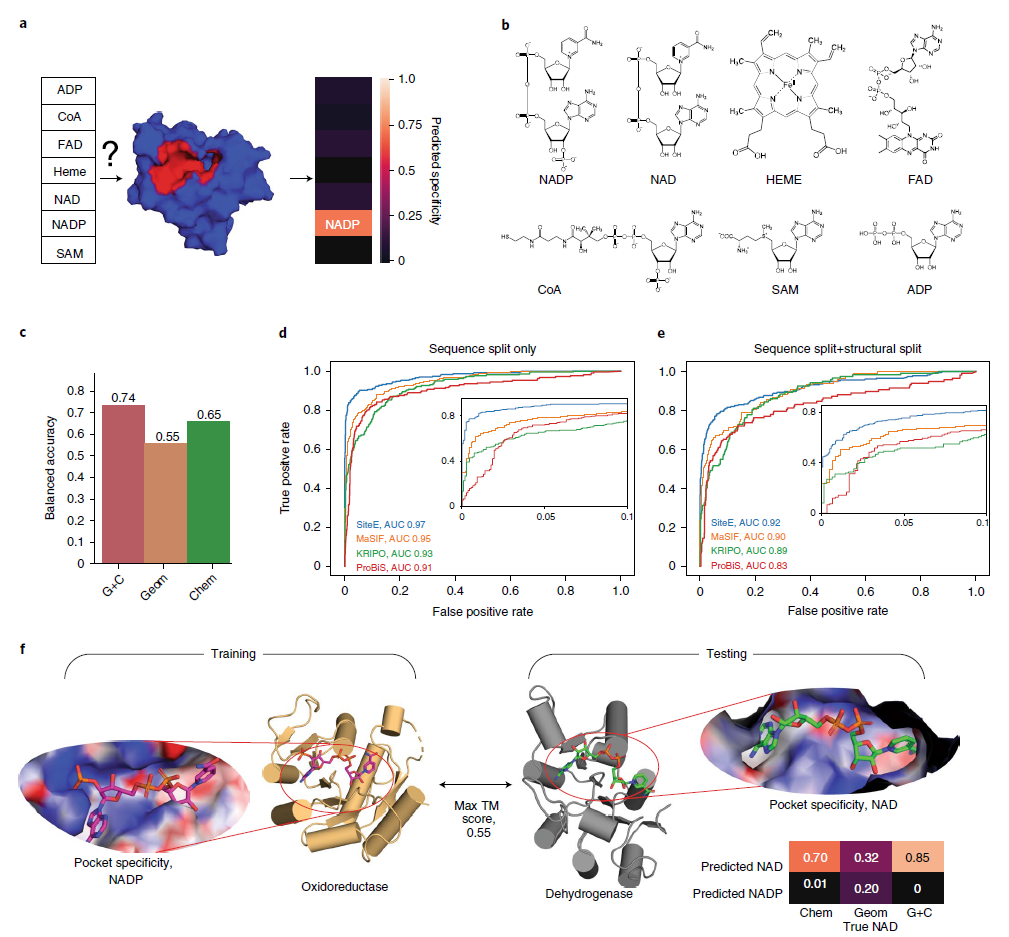
图2. 基于MaSIF-ligand蛋白小分子结合口袋的分类
图片来源:NATURE

图3. 基于MaSIF-site预测蛋白-蛋白相互作用位点
图片来源:NATURE
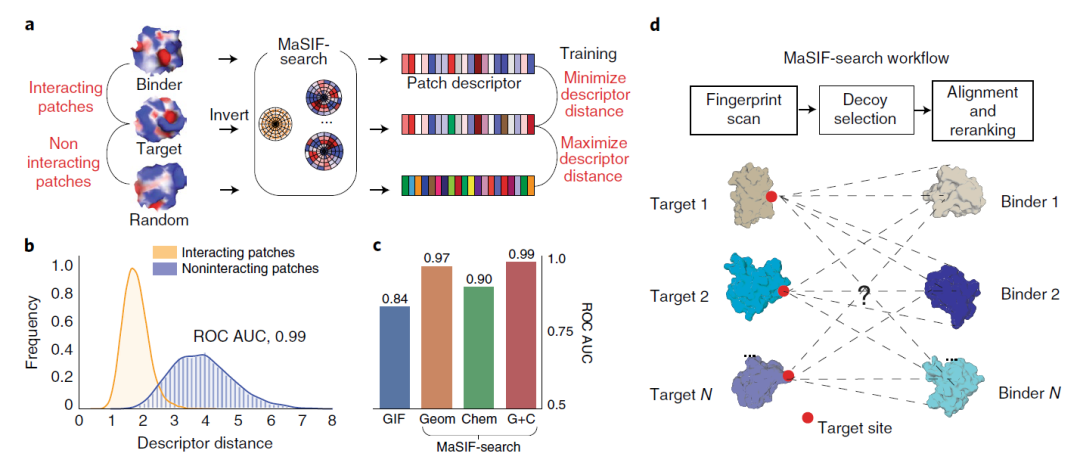
图4. 基于MaSIF-search超快扫描预测蛋白-蛋白复合物
图片来源:NATURE
讨论
分子表面(surface)表征描述了与其他生物分子接触的蛋白质的特征。学习surface指纹的方法可能使人们对蛋白质功能有更完整的了解。可以预见到,MaSIF在蛋白质从头设计领域将大放异彩;另一方面,MaSIF也可用于鉴定蛋白质表面热点,这些“表面热点”可能更易于设计用于治疗的生物制剂(MaSIF-site)。MaSIF-search可以与确定蛋白质结合伴侣的实验方法结合使用。总而言之,MaSIF对蛋白质结构和功能的基础研究以及蛋白质从头设计,以及基于蛋白质的疗法等方面都会产生巨大的影响。
参考文献:
Gainza, P., Sverrisson, F., Monti, F. et al. Deciphering interaction fingerprints from protein molecular surfaces using geometric deep learning. Nat Methods 17, 184–192 (2020).