背景

人类免疫缺陷病毒(HIV-1)逆转录酶(RT)是一种多功能蛋白,包含DNA聚合酶活性和核糖核酸酶H(RNH)活性。RT包含66 kDa亚基(p66)和p66衍生的51 kDa亚基(p51),聚合酶和RNH活性位点均位于p66亚基中。目前虽然有超过10种临床批准的聚合酶抑制剂可供使用,但是这其中确没有RNH抑制剂。鉴于RNH活性对于病毒复制至关重要,因此RNH抑制剂有可能成为抗病毒的重要手段。为了能够合成并测试的几种不同类型的RNH抑制剂,前辈们已经做出了很大的努力,包括通过二价金属离子与RNH活性部位结合的抑制剂。但是,迄今为止,尚且没有任何RNH抑制剂具有实质性的抗病毒活性,并应用于临床。原因在于我们对于RNH抑制剂相互作用的结构信息了解很少,直到最近才公开了与RT或分离的RNH活性部位抑制剂的晶体结构。在所有这些晶体结构中,抑制剂以相似的方式与两个金属离子配位,但是抑制剂与蛋白质之间的疏水或氢键相互作用在在不同结构之间有所不同。蛋白质结构对抑制剂效能的影响尚没有明确的结果。最近,XI等人发表了与羟基异喹啉二酮(HID)型活性位点抑制剂YLC2-155配合使用的RT晶体结构,表明该抑制剂以两种结合方式与RNH结合位点相互作用。对于晶体结构和分子对接的研究结果表明,该抑制剂通过其羟基和羰基螯合了两个Mn2 +离子,而其余化学基团则以两种不同的构型在RNH周围存在,一种朝向Q500,另一种朝向H539。使用分离的RNH结合位点进行的NMR实验显示,当在Mg2 +存在下与YLC2-155结合时,一些RNH残基会产生两组共振。但是,尚未研究分子相互作用的详细机理。除了RNH活性位点抑制剂的螯合特性外,抑制剂与RNH中保守残基的特异性相互作用在抑制活性中也起着重要作用。所以研究RNH抑制剂的重点是要研究这些因素的影响以确定抑制剂相互作用的机制。
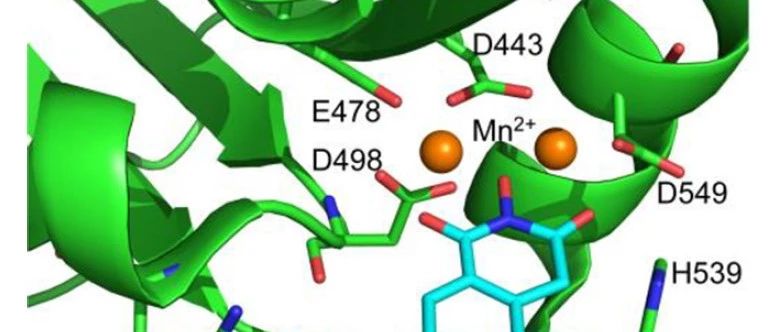
在复合物(5UV5)的晶体结构中观察到的YLC2-155与RNH结合位点的两种结合模式的视图。使用了Mn2 +用于确定晶体结构
图片来自ACS
本文中,XI等人使用NMR光谱,差示扫描荧光法(DSF),分子对接和量子化学计算方法,对包含Mg2 +的RNH结合位点及抑制剂的三元复合物进行了全面的相互作用研究。对于NMR和DSF研究,使用了分离的RNH区域(427至560残基),即通常用于研究活性位点抑制剂与金属的相互作用的部分。Mg2 +可能是HIV-1 RNH活性不可或缺的辅助因子。但是,在生理条件下,由于结合力弱,预计RT中的RNH不会完全被Mg2 +饱和。因此,重要的是要了解抑制剂的结合方式以及是否使无Mg2 +结合态与有Mg2 +结合态之间的平衡向着Mg2 +结合态转移并最终向RNH-Mg2 +-抑制剂三元络合物转移。为了解决这个问题,他们将Mg2 +的浓度从1改变为20 mM,以生成无Mg2 +和结合Mg2 +的RNH的不同群体。他们还改变了20 mM Mg2 +的抑制剂的浓度,以生成三元络合物的不同群体,并通过NMR监测状态变化。NMR实验是在两种不同的条件下进行的。基于HID的活性部位RNH抑制剂YLC2-155和羟基嘧啶二酮(HPD)型活性部位抑制剂ZW566。对以上两种抑制剂进行分子对接和量子力学计算,以研究结合模式,蛋白抑制剂相互作用和螯合特性。两种抑制剂都在两个方向上结合RNH,但是,与YLC2-155相比,ZW566表现出更多的蛋白质相互作用和更强的金属螯合能力。总体而言,XI等人的发现为RNH药物设计提供了宝贵的经验。
结果
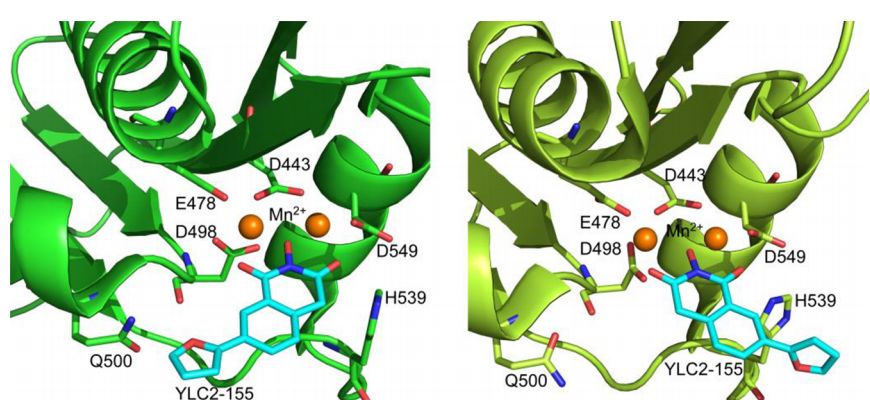
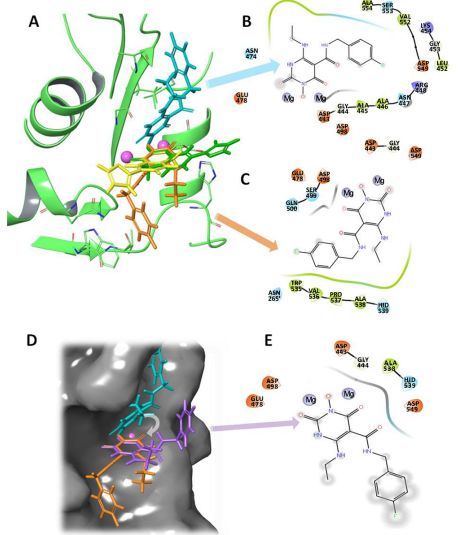
(A)中显示了本研究中使用的RNH活性部位抑制剂的结构。(B)YLC2-155或(C)ZW566结合时显示分裂共振的残基被映射到RNH域(PDB 5UV5)的晶体结构上,并被涂成红色。金属离子显示为球形,金属结合残留物显示为棒状
图片来自ACS
通过构象搜索预测ZW566分子有52个不同共构象。使用灵活的配体采样方案,将所有不同构型的配体对接到RNH的活性中心,对于每个构象,均以特定方式对接。对接后进行分析来获得每个构型的平均结构,并计算RMSD来评估对接方式与平均结构的关系,XI等人得出结论,ZW566出现两种独特的结合模式。结合模式1,从44个对接方式中获得(在对接的44个构象中最低的能量为−8.09 kcal/mol),而从8个不同对接方式中获得的结合模式2(最佳对接能量为−7.36 kcal / mol),在两种结合模式下,ZW566相对于几个活性位点残基的位置是不同的,包括表现出分裂NMR信号的那些残基和位于分裂信号残基附近的那些残基;在结合模式1中,ZW566更接近于Ala445,Ala446和Val552,而结合模式2则使ZW566靠近Val536。此外还在与ZW566相同的对接条件下进行了YLC2-155与RT的分子对接,以直接比较两种抑制剂的结果。为了保证YLC2-155对接方式与先前发布的对接方式相同,显示了两种结合模式。ZW566结合方式1和2与YLC2-155的两种结合方式不同。在两种YLC2-155结合模式下,螯合三联体都位于同一平面上,并且以相似的角度接近金属阳离子。ZW566结合模式2与YLC2-155结合相似,螯合三联体与YLC2-155重叠。如配体相互作用图(LID)所示,在两种结合模式下,与YLC2-155相比,ZW566被大量残基包围。相反,YLC2-155与RNH的相互作用主要在螯合金属离子的羟基异喹啉二酮核心处或附近。为了了解ZW566与这些周围氨基酸残基相互作用的重要程度, 当两种结合模式以相同角度螯合金属阳离子时,我们将ZW566结合模式1与结合模式2的螯合三联体叠加,模拟“ YLC2-155样”结合。此ZW566叠加的LID显示,以类似于YLC2-155的方式结合ZW566会导致氨基酸接触少得多,并且完全将氟化环和氨乙基暴露在溶剂中。
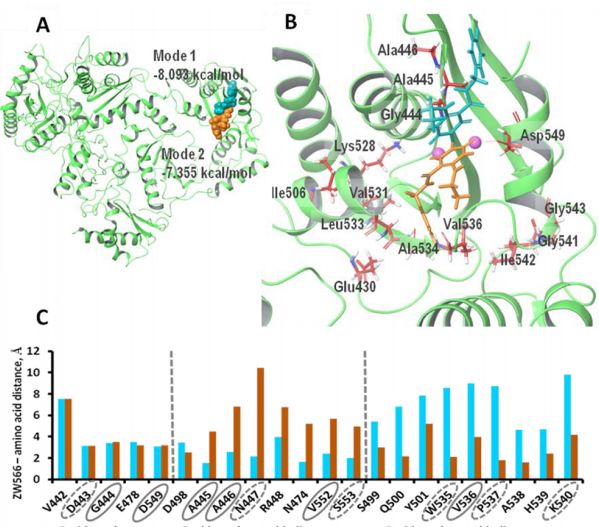
分子对接显示两种结合YLC2-155和ZW566的模式
图片来自ACS
分子对接计算表明与Mg2 和RNH结合的ZW566相互作用更强;XI等人计算了影响对接得分的相互作用能。我们发现,与YLC2-155相比,ZW566的金属结合,疏水相互作用和范德华相互作用均明显更强,这与观察到的对ZW566的亲和力比YLC2-155高一致。与ZW566相比,范德华力较小, 库仑能量相近。
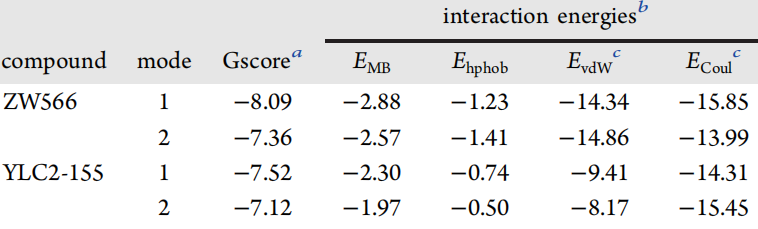
计算的ZW566和YLC2-155最稳定构型的对接分数和其主要成分(以kcal / mol为单位)
图片来自ACS
ZW566的两种不同于YLC2-155的结合模式
图片来自ACS
量子力学计算表明ZW566上的负电荷比YLC2-155小;与RNH区域结合的Mg2 +具有大于1 mM的解离常数,在存在Mg2 +的情况下ZW566和YLC2-155与RNH结合的解离常数分别为1.9±1.7和17.9±7.3μM。这些观察表明,金属-抑制剂的相互作用可以稳定三元络合物。在对接研究中,XI等人研究了每种抑制剂与RNH的Mg2 +结合形式的相互作用。但是,金属-配体相互作用能不能通过对接算法正确估算,对金属-配体配位的准确描述涉及对配体原子上电子密度分布的考虑以及将电子从原子中移出所需的能量。该信息只能从量子力学计算中获得,而量子力学计算则不包括在对接算法中。因此,为了更好地了解ZW566和YLC2-155的金属配位,进行了高精度的量子力学计算,以估算某些电子结构特性,包括部分原子电荷,分子轨道能量和组成以及平均局部电离能。XI等人使用三种不同的方法来计算化合物的原子电荷,以获得与计算方法相对独立的电荷分布模式。从静电的角度来看,当更多负电荷位于配体原子数坐标的金属,金属间的相互作用和配体将更强。结果表明与YLC2-155螯合三元组上的电荷相比这三种原子上的总体负电荷更大,ZW566的螯合氧原子更强,这表明ZW566的金属相互作用比YLC2-155强。他们还对这两种化合物进行了分子轨道分析,以研究电子从配体轨道到金属轨道的离域容易程度。尽管未观察到ZW566和YLC2-155的孤氧对轨道的轨道能量有明显差异,但ZW566的平均计算局部电离能(ALIE)66.86 kcal / mol,比YLC2-155(ALIE = 70.09 kcal / mol)低约3.2 kcal / mol,这表明电子与YLC2-155相比更易于从ZW566脱离。综上所述,所有来自量子力学计算的结果表明ZW566与YLC2-155相比,与金属阳离子的键合性更强。此类观察可能与所观察到的ZW566结合的Mg2 +敏感性高于YLC2-155结合的Mg2 +敏感性一致。
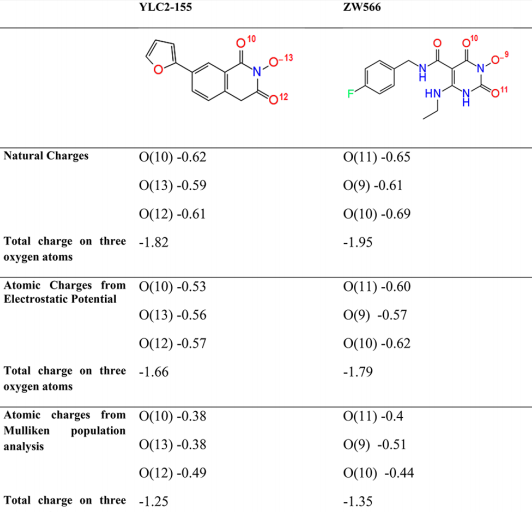
在理论水平B3LYP / 6-311G **下,通过不同电荷估算方法计算的YLC2-155和ZW566原子的部分电荷
总结
逆转录酶RNH对于病毒复制至关重要,迄今为止,还没有有效的RNH抑制剂来抵抗病毒复制。通过分子对接和量子力学计算,本文研究了影响抑制剂相互作用的因素。实验数据表明,ZW566与YLC2-155的RNH结合力更强,这可能是ZW566的较高的金属螯合能力以及与ZW566的更大数量的相互作用造成的。与YLC2-155比较,两种抑制剂都以两种不同的结合方式结合RNH,一种构象比另一种更为有利。另外,当与YLC2-155的类似结合模式相比时,ZW566的两种结合模式之一显示出与周围残基的更多数量的相互作用。因此提高金属螯合强度,更优的蛋白质抑制剂结合模式可能是开发更有效的RNH活性位点抑制剂的重要方向。因此,开发有效的竞争性活性位点RNH抑制剂的策略不仅应考虑金属-抑制剂的配位,还应考虑蛋白质-抑制剂的相互作用和构象选择性。该文章可能为新一代抑制剂的研发提供帮助。
参考文献
(1) Baltimore, D. (1970) RNA-dependent DNA polymerase in virions of RNA tumour viruses. Nature 226, 1209−1211.
(2) Temin, H. M., and Mizutani, S. (1970) RNA-dependent DNA polymerase in virions of Rous sarcoma virus. Nature 226, 1211−1213.
(3) Temin, H. M. (1993) Retrovirus variation and reverse transcription: abnormal strand transfers result in retrovirus genetic variation. Proc. Natl. Acad. Sci. U. S. A. 90, 6900−6903.
(4) Wilhelm, M., and Wilhelm, F. X. (2001) Reverse transcription of retroviruses and LTR retrotransposons. Cell. Mol. Life Sci. 58, 1246−1262.
(5) Eickbush, T. H., and Jamburuthugoda, V. K. (2008) The diversity of retrotransposons and the properties of their reverse transcriptases. Virus Res. 134, 221−234.
(6) Jacobo-Molina, A., and Arnold, E. (1991) HIV reverse transcriptase structure-function relationships. Biochemistry 30,6351−6356.