
引言
通常用于治疗新疾病(例如COVID-19)的药物重定向通常始于对现有药物进行虚拟筛选,然后进行实验验证,但是使用传统的计算方法,实际命中率通常较低。来自中山大学药学院的罗海彬教授研究团队自主发展的基于加速的自由能微扰-绝对结合自由能计算(FEP-ABFE)的虚拟筛选方法可以达到空前的高命中率,在一周内完成对老药数据库的高效筛选,并成功鉴定出15种有效的SARS-CoV-2主要蛋白酶(Mpro)抑制剂,Ki为0.04-3.3μM,特别是抑制活性最好的药物双嘧达莫(dipyridamole)表现出对新冠肺炎较好的临床治疗效果,进一步证明了基于FEP-ABFE的虚拟筛选方法的巨大潜力和可靠性。
背景
由严重急性呼吸系统综合症冠状病毒2(SARS-CoV-2,也称为2019-nCoV)引起的持续的COVID-19大流行已成为全球性危机,最快而有效的方法是将已有的药物重新用于治疗COVID-19。鉴于Mpro在病毒生命周期中的基本功能及其高度的保守性,SARS-CoV-2 Mpro是治疗COVID-19的天然靶标。因此,人们一直在努力尝试使用各种基于药效基团、分子对接和分子模拟的虚拟筛选方法来靶向Mpro的治疗候选药物。但是,使用传统的计算方法进行虚拟筛选的实际命中率一直很低,计算预测的绝大多数的候选分子一般都是假阳性,主要原因之一是因为很难可靠地预测蛋白质-配体分子结合自由能。虚拟筛选的命中率往往取决于虚拟筛选过程中使用的受体-配体结合自由能预测的可靠性和准确性。因此,虚拟筛选工作成功的关键是使用可靠的计算方法来准确预测结合自由能。
加速自由能微扰-绝对结合自由能计算新方法(GA-FEP)
分子间相互作用的自由能扰动(FEP)模拟被认为是一种可靠的方法,能够达到较好的自由能计算精度,但是传统的FEP方法一般仅限于模拟配体的一些细微结构变化以进行相对结合自由能(RBFE)计算。RBFE计算可用于指导有潜力的先导化合物的优化,但不适用于虚拟筛选完全不同的分子结构以识别得到新的靶标药物。与传统的计算方法(例如药效团,分子对接和分子模拟)相比,FEP-ABFE方法的优势在于可以更准确地预测配体与其靶标之间的结合亲和力。但是,先前使用的FEP-ABFE计算非常昂贵且耗时,因此不适合用于虚拟筛选(筛选大量化合物所需的筛选目的)。为了使FEP-ABFE方法对于虚拟筛选切实可行,罗海彬教授团队提出了能准确描述FEP热力学循环中添加束缚步骤ΔU概率分布公式,并将其命名为RED函数。通常FEP约束能量需要12个λ窗口才能够准确计算,而经RED函数分析和拟合后,该方法使用一步微扰(2个λ窗口)即可准确计算约束能量。RED函数极大加速了FEP-ABFE的预测计算速度,使得GA-FEP方法首次能应用于新药的虚拟筛选研究中。
基于GA-FEP的虚拟筛选及实验验证
为了鉴定有效的SARS-CoV-2 Mpro抑制剂,研究团队首先对所有现有药物数据库进行了基于FEP-ABFE的虚拟筛选,然后进行了体外活性测定,如图1所示。
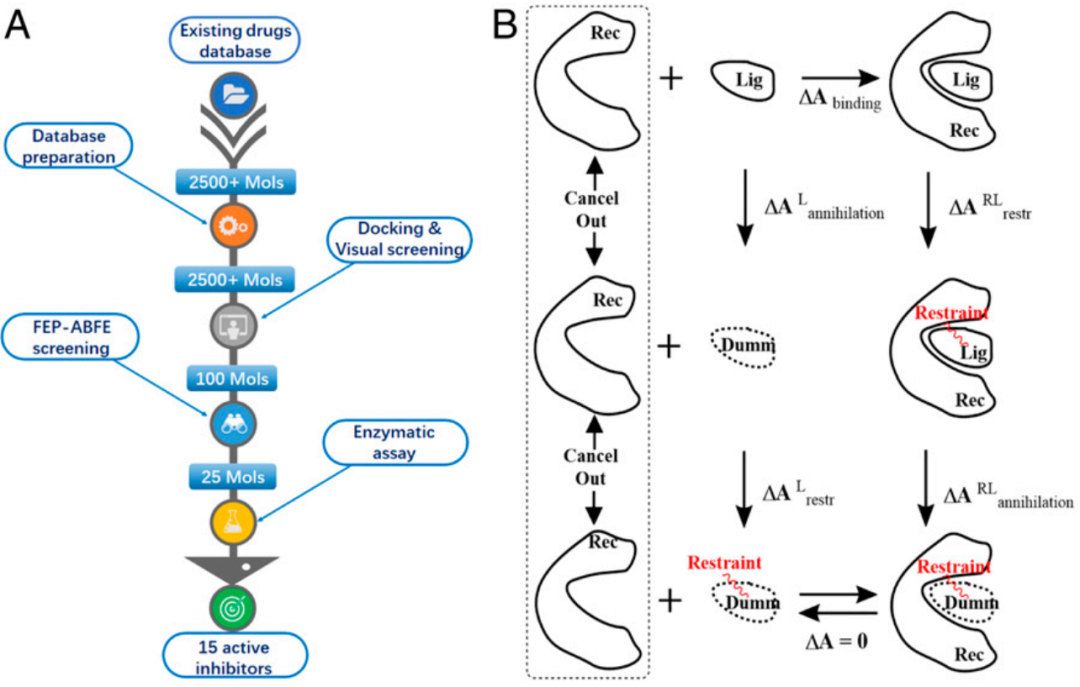
图1 基于FEP-ABFE针对SARS-CoV-2 Mpro的药物重新筛选
图片源自PNAS.
具体而言,将所有现有药物对接到SARS-CoV-2 Mpro的结合位点后,对六个主要残基Cys145,His41,Ser144,His163,Gly143,Gln166和Gln166进行了进一步的FEP-ABFE计算。最终选择了25种药物用于随后的体外实验活性测试。根据体外结果,这25种药物中有16种显示出相当大的抑制SARS-CoV-2 Mpro的潜力,如图2所示。值得一提的是,这些药物与靶标的理论亲合力与实验亲合力差值绝大多数均小于2 kcal/mol,而常用的MM-PBSA和GBSA两种方法得到的差值均大于10 kcal/mol,结果如表1所示。因此,对于蛋白-配体结合自由能预测,FEP-ABFE方法确实比传统的MM-PBSA和MM-GBSA方法都更加准确。
在这16种化合物中,抑制活性最高的化合物为由本方法首次发现的双嘧达莫(Dipyridamole),Ki为40 nM,EC50 为100 nM。该药物已经在后续的临床实验上证实了其对新冠肺炎有较好的治疗效果。
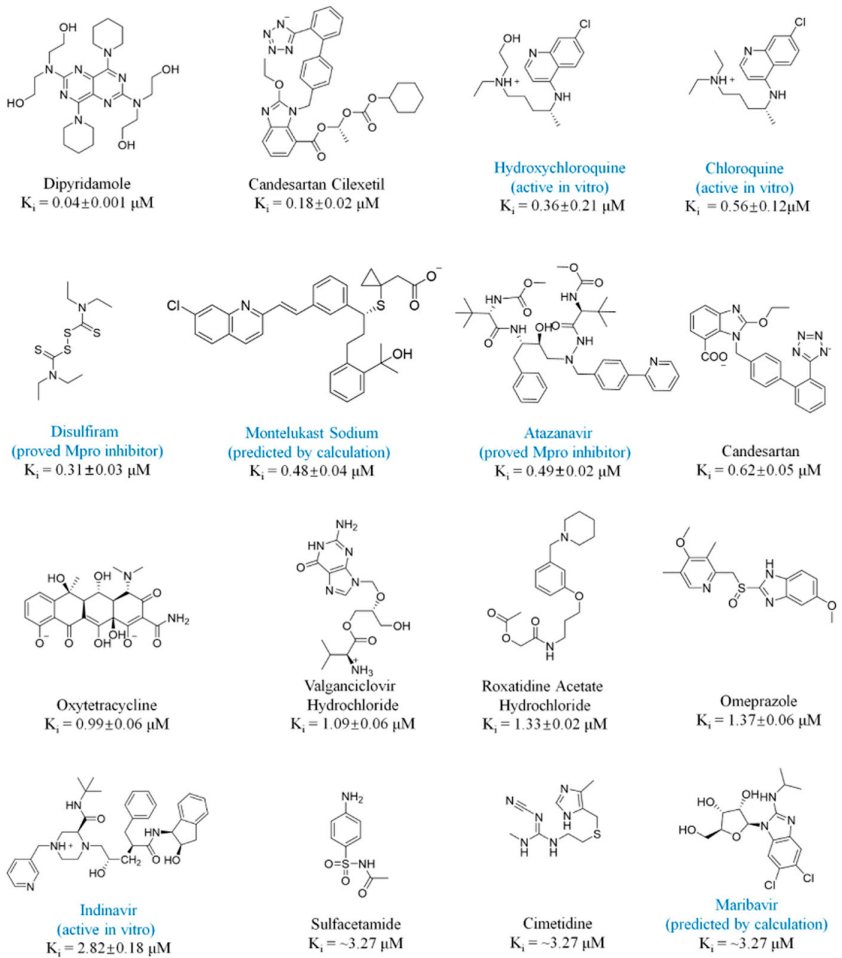
图2 确认的16种SARS-CoV-2 Mpro抑制剂的分子结构和Ki值
图片源自PNAS.
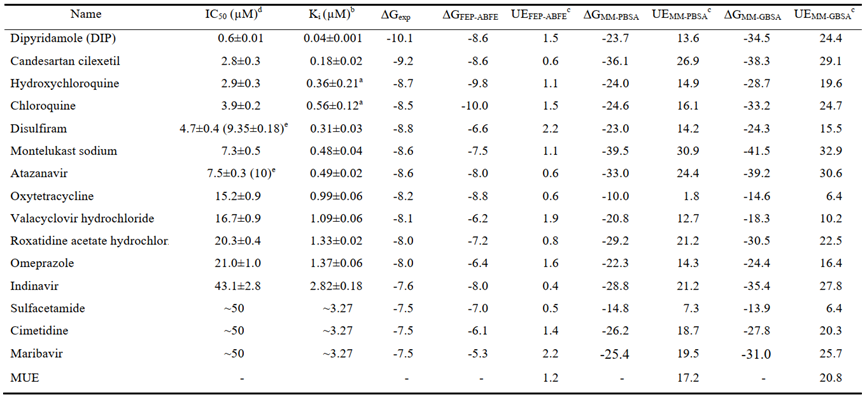
图3 实验确认的SARS-CoV-2 Mpro抑制剂的FEP-ABFE,MM-PBSA和MM-GBSA结合自由能计算结果
图片源自PNAS.
总结
总而言之,本文通过将加速的FEP-ABFE预测用于靶向SARS-CoV-2 Mpro的药物再利用,然后进行实验验证,从现有药物中成功鉴定出总共16种有效的SARS-CoV-2 Mpro抑制剂,其中最有效的是双嘧达莫(Dipyridamole)(Ki = 0.04 µM),目前正在临床研究当中。筛选结果表明通过加速的FEP-ABFE预测方法进行的虚拟筛选已显示出极好的准确性,在Ki = 4μM的阈值下,命中率高达60%。基于FEP-ABFE预测的虚拟筛选方法将在许多其他药物重新利用或发现新的药物工作中将十分有用。
参考文献
Identify potent SARS-CoV-2 main protease inhibitors via accelerated free energy perturbation-based virtual screening of existing drugs.Zhe Li, Xin Li, Yi-You Huang, Yaoxing Wu, Runduo Liu, Lingli Zhou, Yuxi Lin, Deyan Wu, Lei Zhang, Hao Liu, Ximing Xu, Kunqian Yu, Yuxia Zhang, Jun Cui,Chang-Guo Zhan, Xin Wang, and Hai-Bin Luo. PNAS 2020; https://doi.org/10.1073/pnas.2010470117