英国Alchemab Therapeutics股份有限公司的Jinwoo Leem等人提出了来自transformer的抗体特异性双向编码器表示(AntiBERTa),这是一个基于57M人BCR序列(42M重链和15M轻链)预训练的12层transformer模型。在预训练之后,研究人员展示了AntiBERTa嵌入捕获生物相关信息,可推广到一系列应用。作为一个案例研究,研究人员通过微调AntiBERTa来预测抗体结合位,结果在多个指标上优于当前的公共工具。AntiBERTa是已知最深的蛋白质家族特定语言模型,提供了BCRs的丰富表示。AntiBERTa嵌入为多个下游任务做好了准备,可以提高人们对抗体语言的理解。相关的研究成果以“Deciphering the language of antibodies using self-supervised learning”为题发布在国际著名期刊Patterns上。
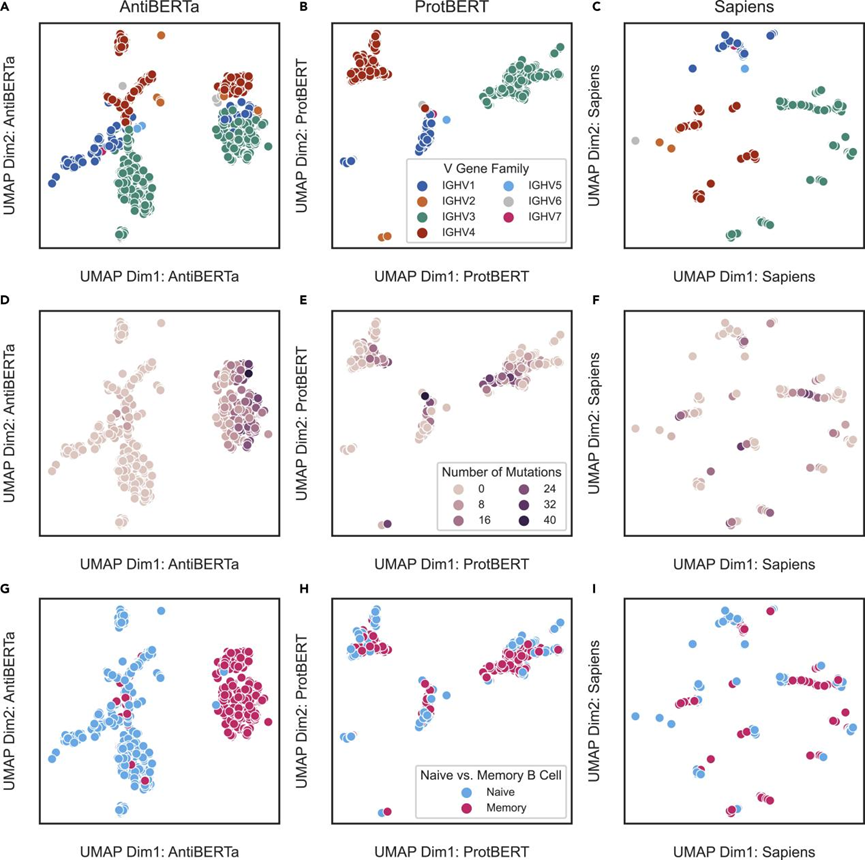
图1. 代表1000个随机选择的BCR重链。图片来自Patterns
AntiBERTa是一个12层transformer模型,在42M未配对的重链和15M未配对的轻链BCR序列上进行了预训练。像其他基于transformer的蛋白质LMs一样,AntiBERTa使用自监督的MLM任务进行预训练。简单地说,输入BCR序列中15%的氨基酸被随机扰动,模型确定了正确的氨基酸取代了这些被屏蔽的位置。这个任务激励了模型对BCR序列的语境理解。
在预训练之后,AntiBERTa输出每个BCR序列的每个残差的分布式向量表示或嵌入。为了使AntiBERTa嵌入可视化,从一个特征良好的公共数据中随机选择1000个BCR重链,然后通过统一流形近似和投影(UMAP)在投影前对长度进行平均。尽管只给出BCR序列而没有其他信息,研究人员发现BCR嵌入根据突变负载和使用的底层BCR V基因片段自然分离(图1)。值得注意的是,来自原生B细胞和记忆B细胞的BCRs也有很好的划分,这表明该模型捕获了重要的功能性信息。
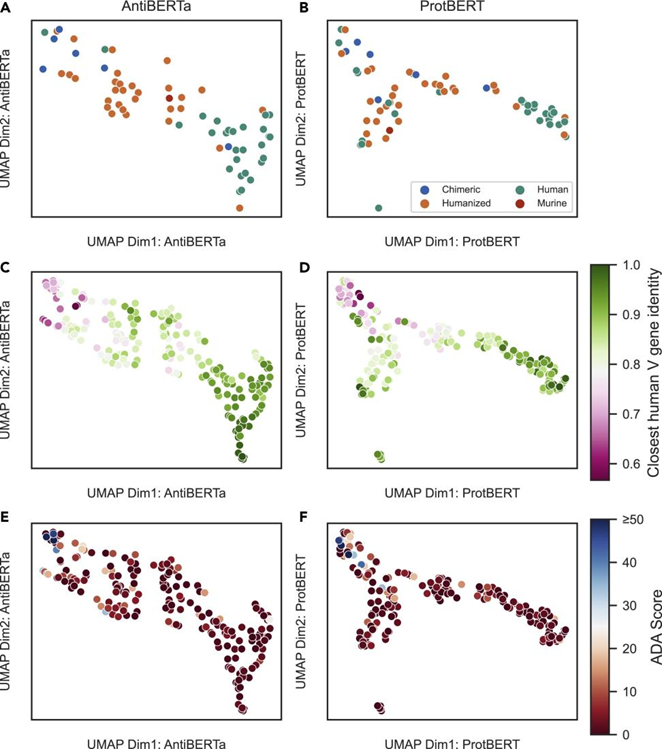
图2. 191种非冗余治疗性抗体的表达。图片来自Patterns
接着,研究人员使用AntiBERTa和ProtBERT将198个特征良好的治疗性抗体的重链嵌入其中。AntiBERTa通常能够根据其来源(即嵌合抗体、人源化抗体、全人源抗体或小鼠抗体)分离治疗性抗体,UMAP即是佐证。这些分离也与序列与它们最接近的人类生殖系V基因的一致性一致(图2A和2B)。ProtBERT能够实现良好的分离,尽管在UMAP中有一个“分叉”。这些抗体也有已知的抗药物抗体(ADA)应答评分;ADA中的分离在很大程度上,与人类种系V基因身份的分离相一致(图2C和2D)。这种嵌入提供了一种潜在的方法来过滤ADA分数高的抗体,并发现更安全的治疗方法。
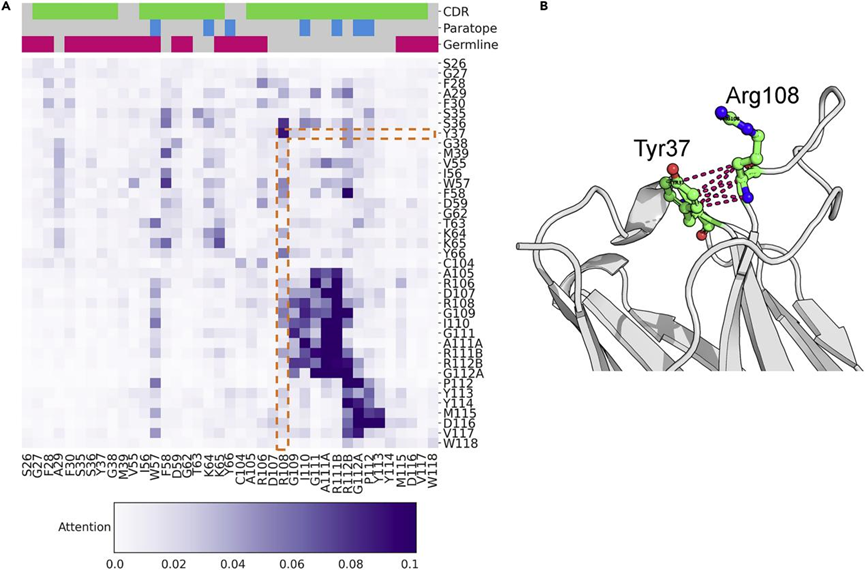
图3. 一种基于网络的机器学习(ML)方法来识别免疫治疗相关的生物标志物。图片来自Patterns
AntiBERTa的12个层中的12个注意头部集中在BCR序列的不同方面(图3)。自注意力得分被用于计算BCR序列中每个氨基酸的最终语境嵌入。通常,AntiBERTa中的自注意力倾向于指向BCR序列的非种系位置或CDR3位置之间。
研究人员发现,与智人相似,具有高自注意力分数的残基对可以揭示长期的结构接触。作为一个案例,研究人员嵌入了aducanumab的重链氨基酸序列,aducanumab是一种最近批准的结合β-淀粉样蛋白的治疗性抗体。AntiBERTa最后一层的第6个注意头部将高度自注意力放在CDR1的Tyr37和CDR3的Arg108之间(图3)。这些位置后来被证实是晶体结构(PDB: 6CO3)内的接触点。
图4. AntiBERTa可以预测形成抛物面的非CDR位置。图片来自Patterns
AntiBERTa可以预测CDR和非CDR位置的抗体结合部位,如ProABC-2。研究人员测试的C1A-C2抗体(PDB: 7KFX)是一种严重急性呼吸综合征冠状病毒2 (SARS-CoV-2)结合剂,AntiBERTa检测到框架中的Tyr66作为抗体结合部位(图4A和4B)。对于研究人员测试的另一种抗体059-152的轻链(PDB: 5XWD), AntiBERTa同样正确地预测了CDRs之外的抗体结合部位(图4C和4D)。AntiBERTa的自注意力通过微调发生变化(图4A和4B),这表明它将自注意力调整为预测互补位的位置。
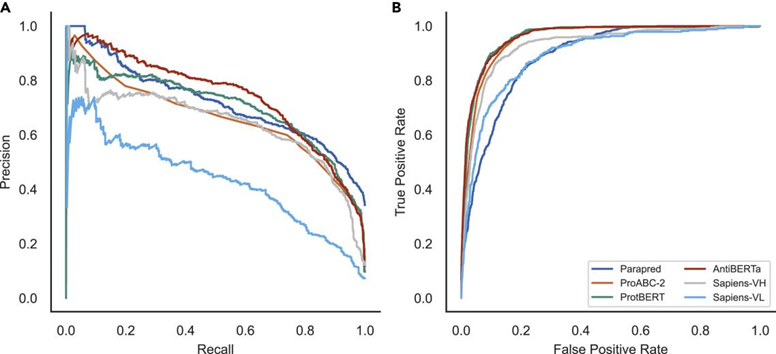
图5. AntiBERTa在抗体结合部位预测方面优于公开可用的工具。图片来自Patterns
研究者的BCR-专用transformer模型在多个指标上优于Parapred、ProABC-2、ProtBERT和Sapiens(图5)。AntiBERTa具有最高的精度,F1, MCC, AUROC和APR,而Parapred具有最高的召回率。虽然AntiBERTa对CDRs及其锚点的召回率高于整个抗体序列,但仍低于Parapred。这可能是由于Parapred被专门训练至少有5个抗体结合部位的抗体,而AntiBERTa只需要两个(一个重链和一个轻链)。
来自AntiBERTa的嵌入反映了BCR的各种生物学意义方面,如突变计数、V基因来源、B细胞起源和免疫原性,尽管在训练前没有这些信息。AntiBERTa理解的一个关键驱动因素是它的多头自注意力机制,它关注BCR序列中结构和功能上的重要残基。鉴于这些功能,研究人员对抗体结合部位预测模型进行了微调,以演示表示的质量,并发现AntiBERTa在多个指标中表现最佳。
AntiBERTa提供了BCR序列的高质量表示,捕捉了BCR的起源、结构和功能的各个方面。来自AntiBERTa的嵌入还提供了BCRs的表示形式,可以通过迁移学习范式用于各种下游任务。
唯信计算提供抗体设计的一站式服务,也欢迎您自助使用我们的WeMol平台完成计算。
WeMol是Wecomput开发的面向生物医学、材料、化学等领域的新一代分子数字智能计算平台。基于流式架构开发,支持低代码定制开发和灵活扩展。核心模块在速度、准确性、效率等方面均超过或媲美主流商业软件。WeMol集成了化学信息学、计算生物学、量子化学、人工智能等计算模块以及小分子和大分子的3D可视化模块,涵盖了生物制剂设计、小分子设计、量子化学、分子模拟等应用需求。平台旨在帮助用户构建一个可积累、可复制、可追溯的计算平台,并可持续、高效地支持计算驱动的创新。
放个链接:wemol.wecomput.com(请使用电脑浏览器访问)
目前Cloud版已正式上线,全功能免费,唯信已开发了数十个计算模块,形成了对Hit->Lead->PCC的全流程赋能的能力,众多独特算法工具(例如构象生成算法AlphaConf,三维形状匹配算法AlphaShape,抗体人源化设计流程AlphaHu,RNA序列优化算法AlphaRNA等)都可在WeMol Cloud中免费试用,详细介绍请见往期文章《分子智能计算平台云端版-WeMol Cloud正式发布》。试用请扫码联系我们~
参考文献
Leem, J., Mitchell, L. S., Farmery, J. H., Barton, J., & Galson, J. D. (2022). Deciphering the language of antibodies using self-supervised learning. Patterns, 100513.
原文链接
https://www.sciencedirect.com/science/article/pii/S2666389922001052#!