抗体是重要的免疫分子,通过与其感染性靶标、抗原结合并触发定向免疫反应来保护宿主。用于诊断和治疗各种疾病的治疗性抗体现已成为现代生物医药的重要组成部分。抗体序列数据只提供了部分免疫库的视图,如何促进抗原结合的相互作用仍依赖于互补决定区(CDR)的6个环的结构,通过实验手段确定结构费时费力,随着计算蛋白质建模的进步和对抗体序列-结构关系的日益了解,对抗体结构进行预测建模已成为业内人士进行抗体设计时的常用手段。
抗体可变片段Fv由重链和轻链 (VH, VL) 的N端结构域组成,虽然VL和VH结构域的结构高度保守,负责抗原识别和结合的CDR环中的5个都可以采用通过序列相似性来预测的规范折叠,但CDR H3环在序列和结构方面差异很大,成为了很多抗体Fv结构预测算法的“阿喀琉斯之踵”。
小编今天介绍的抗体结构预测算法有的来自老牌软件,有的来自最新AI算法,均是业内人士耳熟能详的口碑型选手,可按需取用。
Rosetta始于华盛顿大学David Baker博士的实验室,软件包括一系列用于蛋白质结构的计算建模和分析的算法,RosettaAntibody就是其中一种特异性抗体开发算法,用户可以轻松地根据项目需求使用这个通用的抗体设计框架来定制运行。框架基于严格的生物信息学分析且使用North/Dunbrack CDR来定义。其算法旨在对抗体-抗原复合物的不同序列、结构和结合空间进行采样。它可用于多种项目类型,从重新设计到改进结合亲和力、优化稳定性都可完美胜任。
算法分为三部分:首先用Kabat定义识别CDR,并使用Chothia方案对残基重新进行编号。然后对所有框架和六个CDR中的五个进行模板选择;接着对选定的模板中用同源建模创建初步模型;最后CDRH3从头循环建模完成模型预测,同时优化VH-VL界面。由于RosettaAntibody对于单链抗体的效率较低,因此,对loop定义进行特定调整可以更好地考虑CDR3的特异性。
在一项对抗体建模评估II(AMA-II)中11个未发表抗体结构的基准测试中,RosettaAntibody为其中的4个靶标生成了所有测评工具中表现最好的结构模型,有2个的预测精度低于1A˚ 。所有框架区域和55个非CDR H3环中的42个预测精度低于1A˚。研究还发现将H3的C末端限制为扭结构象将允许更频繁地采样以接近天然构象。
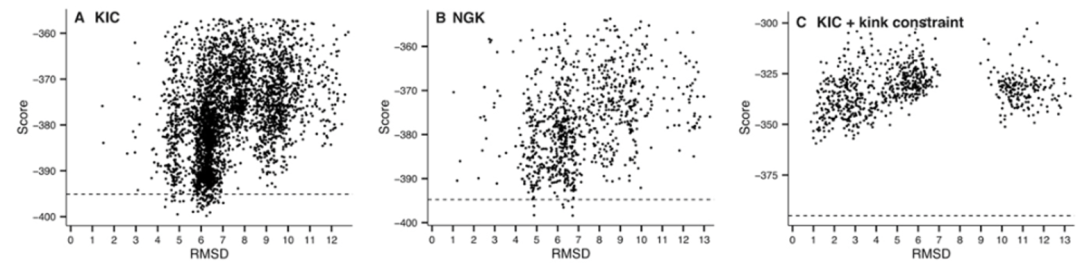
在运用KIC+kink的计算方法后,H3与软件打分的正相关性较高
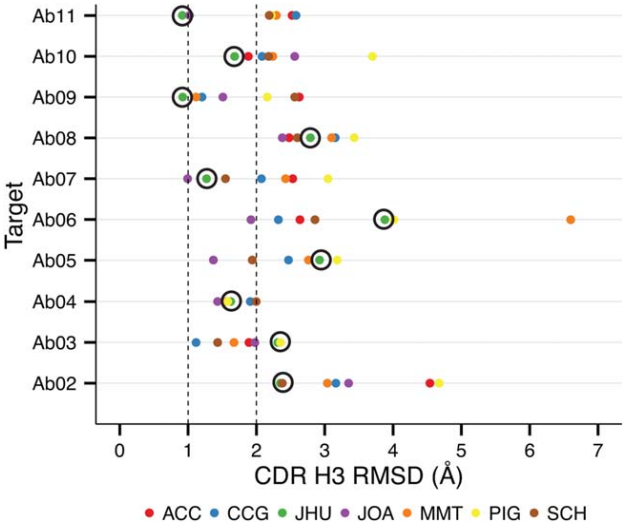
RosettaAntibody(圈起来的绿点)为AMA-II中11个靶标中的4个产生了最好的CDR H2模型【1】
抗体的特异性主要受Fv区中补充决定区CDRs的影响,大多数用于抗体Fv结构预测的方法都采用某种形式的嫁接,H3的预测精度差强人意。约翰·霍普金斯大学的Jeffrey J. Gray教授基于深度学习预测蛋白结构方法的进展开发了DeepAb,性能优于现有的基于嫁接的方法,可用于广泛的抗体预测和设计任务。
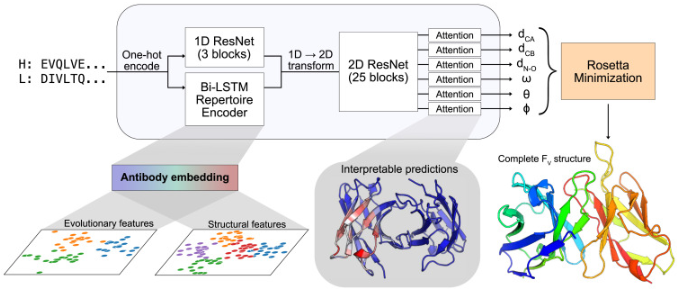
算法由两个主要阶段组成,第一个阶段是一个深度残差卷积网络,用于预测Fv结构,用残差对之间的相对距离和方向表示。网络只需要轻重链序列作为输入,并设计了可解释组件,以提供对模型预测的洞察。结构预测模块最重要的组件是2D ResNet,DeepAb将2D ResNet中的输出通过Attention层转化到6个输出分支,用以表示Fv结构中的关键参数。第二阶段基于fast Rosetta,利用网络的预测来实现结构设计。
与另外三种基于嫁接的抗体特异性结构预测算法RosettaAntibody-G 4、6, RepertoireBuilder 5和 ABodyBuilder相比,DeepAb在重链框架和轻链框架上实现了分别相对于次佳方法14%–18%和16%–17%的平均RMSD改进。在有难度的预测H3环结构任务中,DeepAb平均RMSD为2.33A˚,比次佳方法提高了16%。且随着H3循环长度的增加,所有方法的性能都会下降。只有DeepAb几乎不受此因素影响, 会为每个循环长度生成最准确的模型。
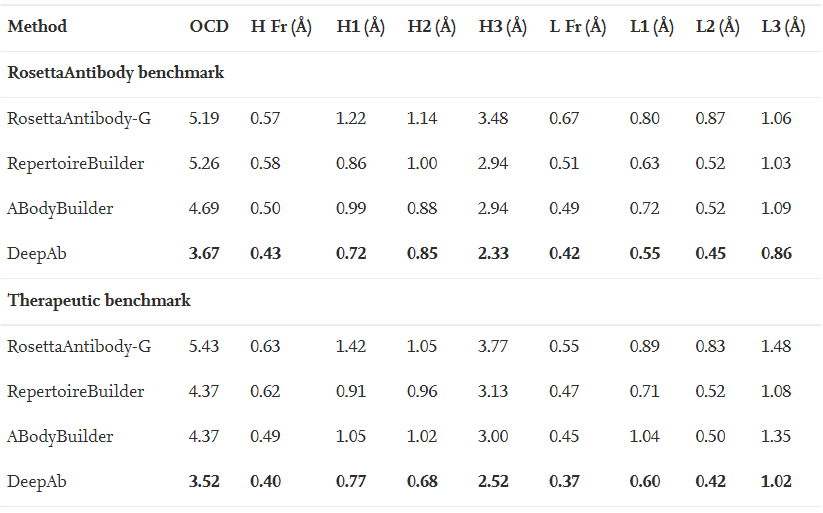
DeepAb在基准数据集上预测Fv域结构的性能【2】
今年4月,约翰·霍普金斯大学发表了可以快速预测抗体结构的IgFold深度学习方法。尽管一些深度学习方法显著提高了包括H3环在内的CDR区的建模精度,但仍存在限制算法实用性的因素。例如预测时间太长、不能直接合并模板数据等,IgFold很好地解决了这类问题。
不同于用Rosetta网络实行抗体设计的DeepAb,IgFold是端到端的预测,算法先从一个预训练的语言模型提取序列表征(该模型在558M自然抗体序列上进行的训练),然后通过图网络直接预测骨架原子坐标。精度媲美AlphaFold2且预测所需时间不到1分钟。在4个被测的抗体结构预测算法中(ABodyBuilder, DeepAb, ABlooper, 和AlphaFold),IgFold预测速度不超过1分钟而且能够预测全原子结构,ABlooper最多一分钟,DeepAb则为10分钟,耗时最久的是AlphaFold,需要大约一个小时。在CDR H3预测准确率层面,IgFold精度好于其他算法,平均RMSD为2.99A˚。IgFold与Alphafold-Multimer使用类似的训练数据集,性能上较为接近。预测纳米抗体时,Igfold对CDR3预测的准确性往往不如Alphafold(平均RMSD为3.85Å),但速度明显更快(Igfold少于30秒,Alphafold则是30分钟)。
除了结构预测外,IgFold还能产生很多靶点的不同构象,这有助于优化现有抗原抗体对接算法的计算速度和准确率。
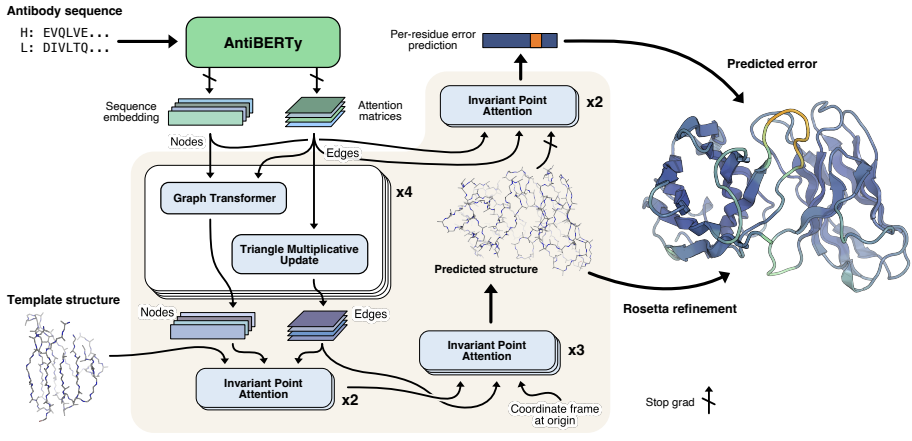

IgFold预测(A)抗体Fv域和(B)纳米抗体结构的准确率【3】
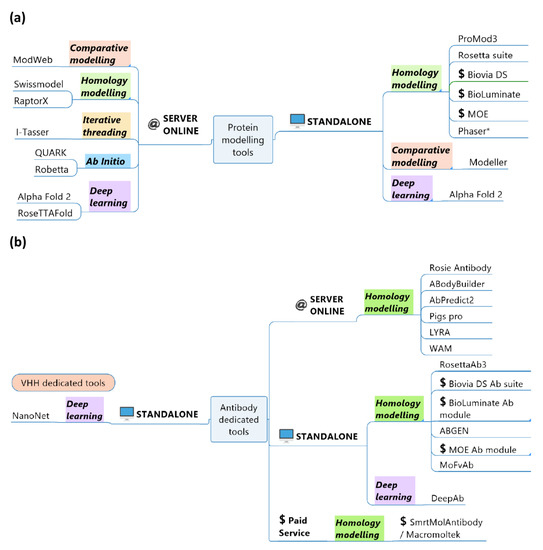
除了以上介绍的三款知名算法,可做抗体结构预测的算法还有AlphaFold2、AbodyBuilder、RaptorX、AbPredict2、RoseTTAFold、NanoNet以及商业软件MOE和薛定谔BIoLuminate等【4】,限于篇幅不展开详述,可自行了解。
上述被业内专业人士广泛使用和验证的算法均已部署在唯信计算自主研发的分子智能计算平台WeMol上(wemol.wecomput.com),只需上传序列即可得到理想的大分子结构,还可在线查看建模结果。无需配置环境,开箱即用,欢迎试用!
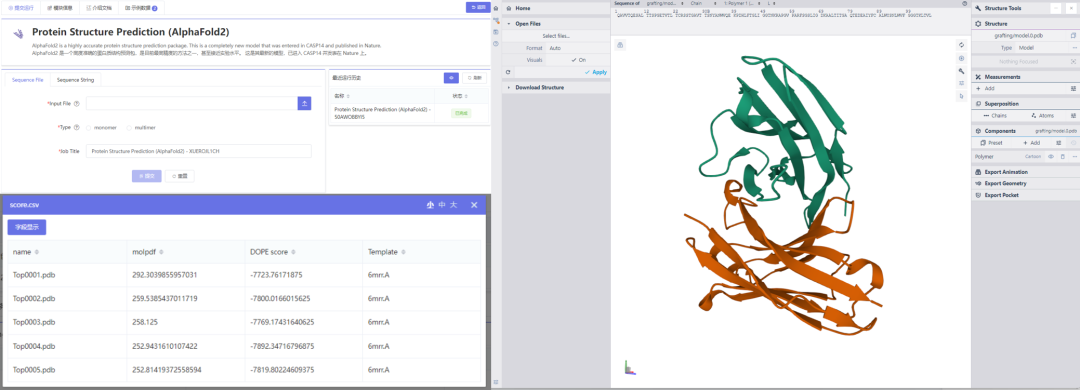
WeMol是Wecomput开发的面向生物医学、材料、化学等领域的新一代分子数字智能计算平台。基于流式架构开发,支持低代码定制开发和灵活扩展。核心模块在速度、准确性、效率等方面均超过或媲美主流商业软件。WeMol集成了化学信息学、计算生物学、量子化学、人工智能等计算模块以及小分子和大分子的3D可视化模块,涵盖了生物药设计、小分子设计、量子化学、分子模拟等应用需求,数十个计算模块可以对Hit->Lead->PCC的全流程进行赋能。平台旨在帮助用户构建一个可积累、可复制、可追溯的计算平台,并可持续、高效地支持计算驱动的创新。
放个链接:wemol.wecomput.com (请使用电脑浏览器访问)
WeMol本地化及云端版本均支持大分子计算、小分子计算、数字化三个解决方案,云端版本WeMol Cloud自8月正式上线后,受到了业界的广泛关注,获得了从入门到专业用户的高度认可。众多独特算法工具(例如构象生成算法AlphaConf,三维形状匹配算法AlphaShape,抗体人源化设计流程AlphaHu,RNA序列优化算法AlphaRNA等)都可在WeMol Cloud中免费试用,详细介绍请见往期文章《分子智能计算平台云端版-WeMol Cloud正式发布》。欢迎扫码联系我们以获得更多支持~
Wecomput一直致力于打造开放、包容、合作的计算生态,除了WeMol现有算法,我们欢迎各位算法开发者或机构将自己的算法部署到WeMol上,获得免费推广及真实用户使用反馈,惠及更多用户,一起在行业层面推动计算技术的广泛应用和发展,加快药物研发进程,让计算创造更多价值。
【1】Weitzner BD, Kuroda D, Marze N, et al. Blind prediction performance of RosettaAntibody 3.0: grafting, relaxation, kinematic loop modeling, and full CDR optimization. Proteins 2014;82:1611-1623.
【2】Ruffolo JA, Sulam J, Gray JJ. Antibody structure prediction using interpretable deep learning. Patterns (N Y) 2022;3:100406.
【3】Jeffrey AR, Lee-Shin Chub, Jeffrey JG, et al. Fast, accurate antibody structure prediction from deep learning on massive set of natural antibodies.Biophysical 2022;121:155a-156a.
【4】Vishwakarma P, Vattekatte AM, Shinada N, et al. VHH Structural Modelling Approaches: A Critical Review. Int J Mol Sci 2022;23.