
引言
耐药性通过药物靶点的突变威胁到许多重要的治疗方法。人们对于突变尤其是远离活性位点的突变的组合,其改变药物结合以赋予耐药性的分子机制了解甚少。本文开发了一种机器学习策略,该策略将平行分子动力学模拟与实验效价相结合,以确定潜在的耐药性保守机制。通过从分子模拟中提取物理特征进行分析,然后将其集成到一个一致且可解释的弹性网络模型中。为了严格测试该策略,作者使用了具有多种突变的HIV-1蛋白酶突变体,其结合药物darunavir的效价范围从皮摩尔到微摩尔级别。通过特征降维产生了具有四个特定特征的模型,该模型预测训练和测试集在整个效价范围内的抑制剂结合自由能与实验值的误差在1 kcal/mol以内。
背景介绍
耐药性在传染性疾病和肿瘤中无处不在,其阻碍了药物治疗的成功,并严重影响了人类健康。通常,耐药性是由突变引起的,这些突变会破坏药物与靶标的相互作用,进而在存在药物的情况下保留靶标的功能。由于失去药物-靶标相互作用而产生的耐药性在病毒和低复杂性生物中十分普遍,这些生物本身不具有编码辅助耐药性基因的基因组能力,但会出现在所有快速发展的药物靶标中。与通常的假设相反,远端突变(远离活性位点)不仅可以补偿原本有害的活性位点突变,而且可以直接赋予耐药性,原因在于远端突变可以通过蛋白内相互作用网络传播的动态变化来改变药物与靶标之间的相互作用。与之相一致,NMR研究表明,耐药性突变体显示出蛋白质动力学的显著变化。因此,在理解和预测耐药性时,不仅需要考虑酶抑制剂系统的结构,而且还应考虑其分子动力学。
对药物产生耐药性的分子机制可能很复杂,因此很难预测。尽管已知酶抑制剂的动力学对耐药性尤其是由于远端突变导致的耐药性至关重要,但其中涉及的分子机制尚不清楚。我们可以通过给定突变体的特定的动态变化和对酶-抑制剂系统进行全面分析而获得的保守机制来赋予耐药性。通过鉴别这种保守的机制将能够预测耐药性的原因,并设计出更强大的抑制剂来避免耐药性。研究最多的理解耐药性的系统是HIV-1,这是一种快速发展的病毒,其治疗历史上一直受到药物耐药性的严重阻碍。众多可利用的耐药性突变体和大量实验数据使HIV-1蛋白酶成为一种阐明耐药性分子机制的理想模型系统。
方法
抑制剂结合的缺失是结构和构象动力学改变的结果。为了量化这些变化并使它们与实验酶抑制作用相关联,作者计算了蛋白质-抑制剂以及蛋白质-蛋白质相互作用和分子动力学的描述子,主要包括:范德华相互作、氢键、扭转角熵和蛋白Cα的RMSF。对于涉及至少一个突变蛋白质残基的每个分子特征,构建回归模型并将其与竞争的无效模型进行比较。
结果1. 数据集收集
作者选择了由28种HIV-1蛋白酶突变体组成的集合,以覆盖超过6个数量级的darunavir(DRV)的敏感性和耐药性。每个突变体最多包含24个突变,在酶中的99个残基中有48个被取代(图1)。突变的空间分布(图1b)突出显示了HIV-1蛋白酶的显著基因组可塑性。只有几个位点具有≥3个取代(图1a),包括多态性残基63和89,以及三个与耐药相关的位点10、54和82。这些耐药突异体中的大多数取代都位于抑制剂结合位点的远端。根据氨基酸序列,蛋白酶突变体聚类成三个主要亚组(图1c),其中聚类3包括从DRV和DRV类似物的病毒传代实验获得的所有突变体,聚类2包括具有少于五个氨基酸取代的突变体,而聚类1包括从临床分离物中获得的所有蛋白酶突变体。
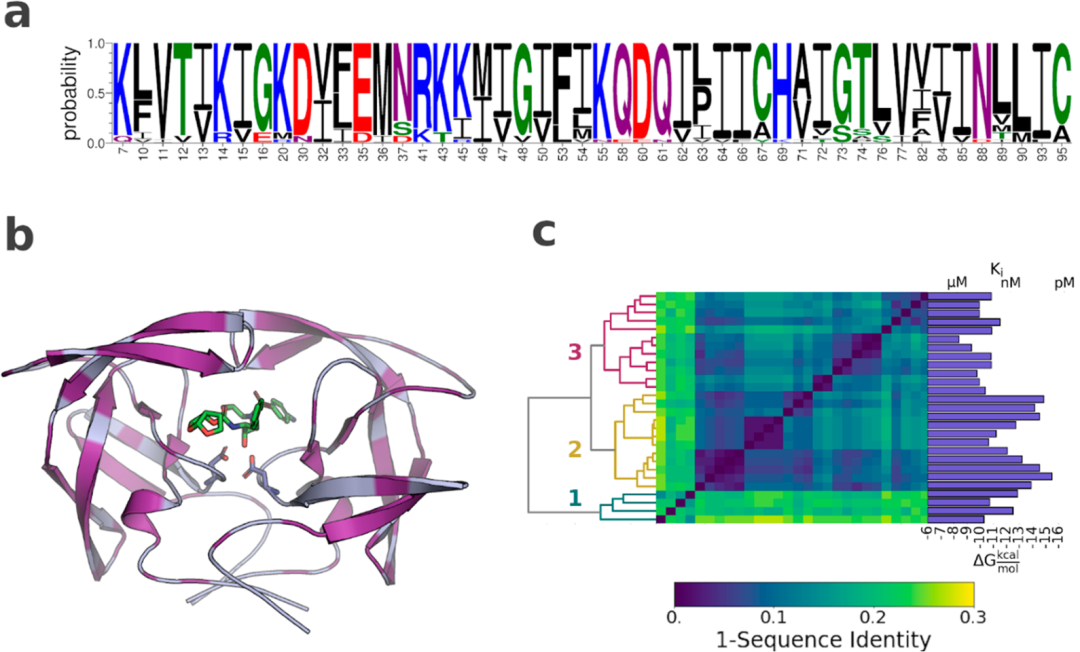
图1. HIV-1蛋白酶突变体中的氨基酸序列突变和分布
图片来源于JCTC
结果2. 特征提取
弹性净回归(elastic net regression)模型被用于确定2858个特征中哪个与观察到的效价最相关。该模型适合于三分之二的训练数据,而三分之一则留给模型评估。为了确保所得系数的收敛,此过程在数据集的随机排列中重复了100次。排序后,平均绝对系数呈现出指数衰减,曲线的拐点位于0.02(图2a)。所选特征中只有9个系数的系数大于0.02,这被认为与效价最相关。根据它们之间的相互关系,可以将这9个选定特征分为两个不同的类,这表明它们相互依存的复杂性(图2b),同时映射到蛋白酶-DRV复合物的结构上如图2c。作者先前已经提出蛋白酶的疏水核心的重排,作为远端突变改变抑制剂相互作用以赋予耐药性的潜在机制。上述结果不仅证实了这些假设,而且所选特征还表明了与此类变化相关联的关键相互作用,这些相互作用转可以化为效价的改变。
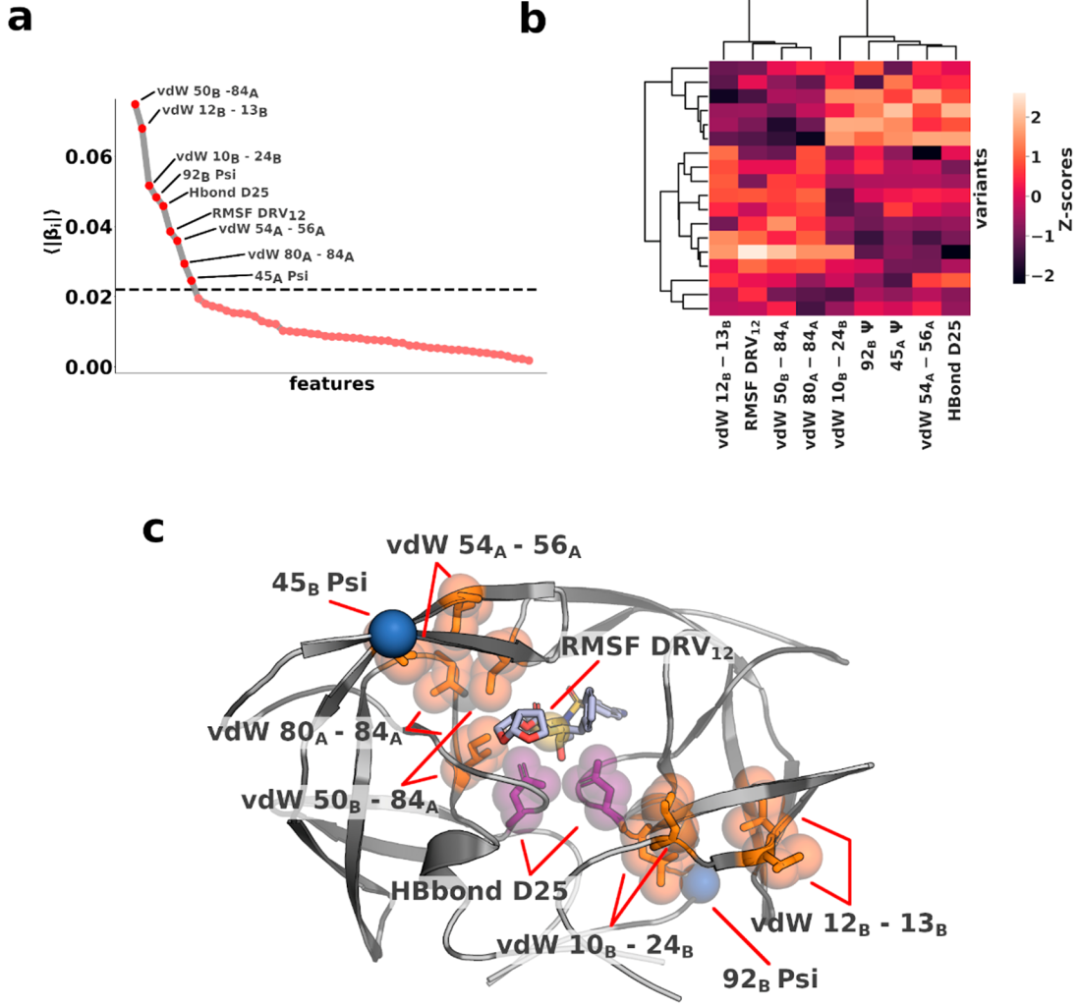
图2. 代表抑制剂效价的分子特征
图片来源于JCTC
结果3. 效价预测模型
接着确定了使用以最少可能的参数确定的九个特定特征预测效价的最佳模型。对这九种特征的所有511种可能组合进行了评估,以收敛于最终模型。该模型仅包含四个特征(vdW 12B–13B,vdW 10B–24B,vdW 50B–84A和HBond D25)。使用5倍交叉验证,训练集上具有这四个特征的模型的均方根误差(RMSE)为0.9 kcal/mol,Pearson相关性为0.9,Spearman相关性为0.8(图3)。接下来,在独立的数据集上测试模型,RMSE为1.3 kcal/mol,相关系数降至0.6和0.5(图3)。测试集上的性能获得了模型的预测能力和通用性,预测的效价变化与实验值非常吻合。由于使用完整的训练集选择了参数,并且模型可以将预测能力转化为原始数据,因此该级别的准确性可以确保模型的物理合理性。
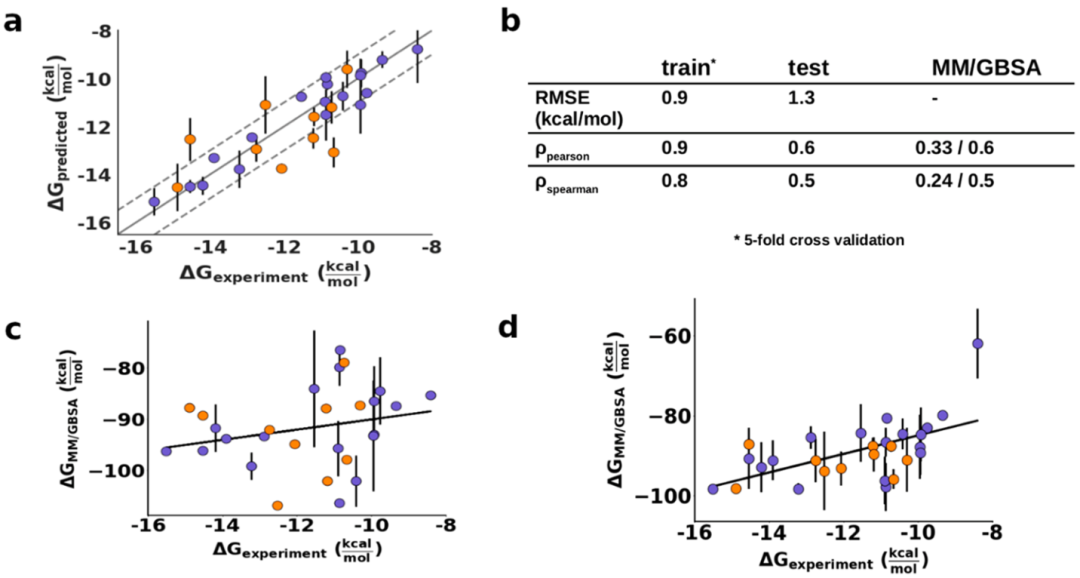
图3. 预测DRV效价的回归模型的评估
图片来源于JCTC
结果4. 效价的分子指标
作者详细研究了预测模型中使用的四个特定特征,这些特征表明了耐药性分子机制中涉及的物理性质和相互作用。回归模型中特征的权重在表1中提供。这些特征中显示出与效价最强关系的特征是远端蛋白酶残基12B和13B之间的vdW相互作用。在训练和测试集中,残基12和13之间的短程相互作用的减少与DRV结合亲和力的降低密切相关。由于这两个残基彼此相邻,因此相互作用的丧失可以通过突变或相对侧链方向的变化来解释。残基13是其他(非B型)HIV亚型中常见的多态性突变,未经治疗的病毒人群中存在13V可以降低耐药性的遗传障碍。总的来说,作者确定了选择性和特异性的相互作用,这些相互作用指示了酶的疏水性核心(vdW 10B–24B和vdW 12B–13B)、活性位点的变化(vdW 50B–84A)以及与结合抑制剂的相互作用(HBond D25),所有这些分子指标可以看作是效价丧失的决定因素。
表1. 回归模型中特征的权重
表格来源于JCTC
总结
阐明耐药性的分子机制对于设计能够避免耐药性并保持针对快速发展的疾病靶标的效价的抑制剂的设计至关重要。本文开发了一种结合分子动力学和机器学习的策略,该策略从分子动力学模拟中计算出大量的相互作用,描述了分子内和分子间的相互作用以及蛋白质和配体动力学的描述子,进而通过四个易于解释的物理特征生成了效价预测模型。在针对HIV-1蛋白酶的研究中,作者证明了远端和活性位点突变的组合如何影响酶的动力学,这种相关性不仅是氨基酸序列变化的指示,而且还反映了酶抑制剂复合物的分子相互作用和动力学变化。作者通过机器学习进行的综合分析确定了发生耐药的保守机制。将机器学习与分子动力学以及实验数据相结合,以识别决定效价的关键因素,将有可能成为药物设计中的一种强大策略。
工具下载地址
https://github.com/SchifferLab/ROBUST
参考文献
Florian Leidner, Nese Kurt Yilmaz, and Celia A. Schiffer, Deciphering Complex Mechanisms of Resistance and Loss of Potency through Coupled Molecular Dynamics and Machine Learning, J. Chem. Theory Comput., 2021, 17, 4, 2054-2064. DOI: 10.1021/acs.jctc.0c01244.