
ProteinTools不同于只含单一应用的蛋白结构分析软件,它以web服务形式提供了四种程序:疏水簇识别、氢键网络识别、盐桥识别和接触图识别。
背景介绍
蛋白质的三维结构对其生物学功能来说至关重要,因此能准确对蛋白结构进行表征或预测意义重大。蛋白是在各种非共价相互作用(如氢键、范德华力、疏水和离子相互作用)的驱动下折叠成天然结构的。因此,要在分子水平上理解蛋白质的特征和功能,就必须对这些相互作用进行描述。现有结构生物学中的大多数计算工作都集中在蛋白质结构预测上,一些用于结构分析的工具只提供了对单个结构属性的分析,并且研究者通常需要适应不同编程语言编写的软件包。本文介绍了拜罗伊特大学团队开发的一款名为ProteinTools 的web服务。相比于其他的工具包,ProteinTools无需繁琐的编程,仅需要通过输入PDB编号或者上传蛋白结构即可快速获得四种不同结构属性分析结果。本文以Di-III_14结构域为例展示了ProteinTools中四种应用程序的应用。
ProteinTools web服务器地址
https://proteintools.uni-bayreuth.de
网站首页:

使用手册
Di-III_14是含有74个氨基酸长度的蛋白,其结构中含有四条β-折叠片和两个排列在β折叠片的一边的α螺旋。β折叠的顺序是1243,其中4号链与其他的β片呈反向平行。研究人员观察到,虽然Di-III_14蛋白在毫秒级的时间内以两种状态展开,但其在高浓度尿素中却可以维持折叠几秒钟,这在天然蛋白质中是一种不常见的特征。实验揭示了许多在慢时间尺度内相互转换的高能状态,这种状态与大型静电网络和疏水簇的形成具有结构相关性。本文作者使用ProteinTools来显示这些属性的计算。
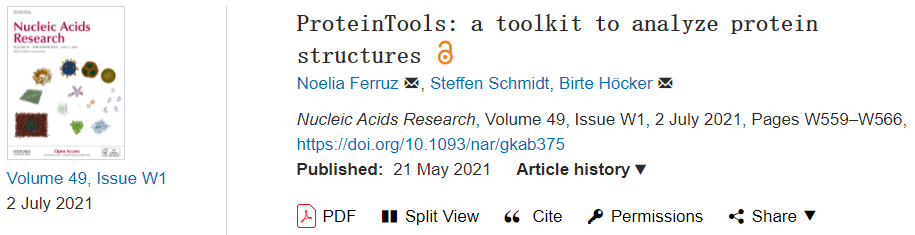
Di-III_14蛋白包含一个大的疏水簇
对Di-III_14进行的氢交换(HDX) NMR分析结果显示了在慢时间尺度上构象状态之间的变换过程,这与氯化胍变性中显示的快速展开过程存在显著差异。虽然这两种过程都倾向于提供天然蛋白质中可比较的评估,但在蛋白质设计过程中进行的稳定性优化可以引入多种相互作用以稳定紧密排列的内部,导致在天然蛋白质中观察不到的复杂行为。作者将强保护的主链酰胺氢(NHs)映射到结构上,发现它们对应于一个被极性侧链包围的大型疏水核。疏水簇的分析显示,单个簇包含14个残基,总面积为1654.0Å2。该簇跨越所有二级结构元素的残基,大多数氨基酸属于β链。每个残基的面积是44.7 Å2,残基之间有37个相互作用。为了解这些参数是否与一种紧凑的IF3-样蛋白相对应,作者比较了Di-III_14的疏水簇与天然IF3样蛋白的疏水簇。随后,作者从SCOPe(40)下载了所有结构域。SCOPe是一个根据蛋白质的拓扑结构和进化关系进行分类蛋白结构的数据库。IF3样蛋白SCOPe标识符为d.68。提取d.68折叠的全部蛋白后,作者剔除了序列长度超过150个氨基酸的蛋白,得到43个结构。使用ProteinTools对这些结构进行可视化显示,每个结构的平均的疏水簇的数量为2.2,位于螺旋和折叠链之间的疏水簇的面积在所有情况中是最大的。这些蛋白的疏水簇的平均面积是1957.5 Å2,略大于Di-III_14 (1654.0 Å2),但在标准偏差(±1078.9 Å2)之内。平均残基量为14.1,与Di-III_14的分析结果一致。用于比较,图lb展示了一个具有代表性的IF3-like蛋白,其含有三个疏水簇,最大的疏水簇面积为1978 Å2。根据这些结果,不能得出Di-III_14的疏水簇与IF3-like天然蛋白的疏水簇有显著差异的结论。
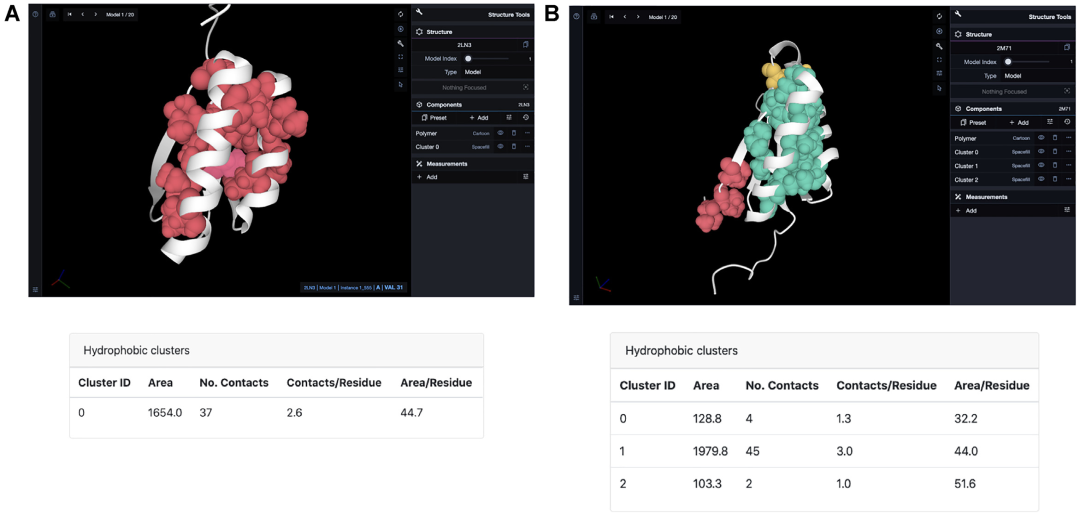
图1. Di-III_14和某IF3-样蛋白的疏水簇分析。图片来源于:NAR
氢键网络
Basak等人观察到两个静电网络,一个跨越αl和α2的表面,另一个包含四个盐桥连接两个内部β链,β2和β4。为了总结这些发现,作者计算了Di-III_14的氢键网络(https://proteintools.unibavreuth.de/bonds/structure/21n3),ProteinTools通过观察2.5 Å距离和120°角内的氮和氧供体及受体,来计算侧链之间的氢键网络。Di-III_14包含8个氢键网络(图2)。最大的一个与Basak等的描述类似,贯穿β1、β2和β4,包含6个残基(氢键网络4,图2,浅绿色)。这些残基分别是Thr6, Glu30, Glu32。Gln64, Arg69和Arg71。另外两个网络加强了内部链的相互作用:网络5(蓝色,Asp34和Lys67)和网络7(暗黄色,Asp28和Ser 75)。
在作者的分析中,观察到螺旋中总共有四个氢键网络,其中三个主要跨越α2。网络3(橙色)由残基Glu 45、Glu49和Lys61组成,将α2和β3连接在一起。类似地,网络0(红色)通过β1中的Ser9与α1中的Asn11和Glu4将β链和螺旋连接起来。另外两个网络对应于网络2(黄色),完全包含在α2 (残基Asp50和Lys 54)和网络1中(深绿色),通过Lys12和Glu43连接αl和α2。
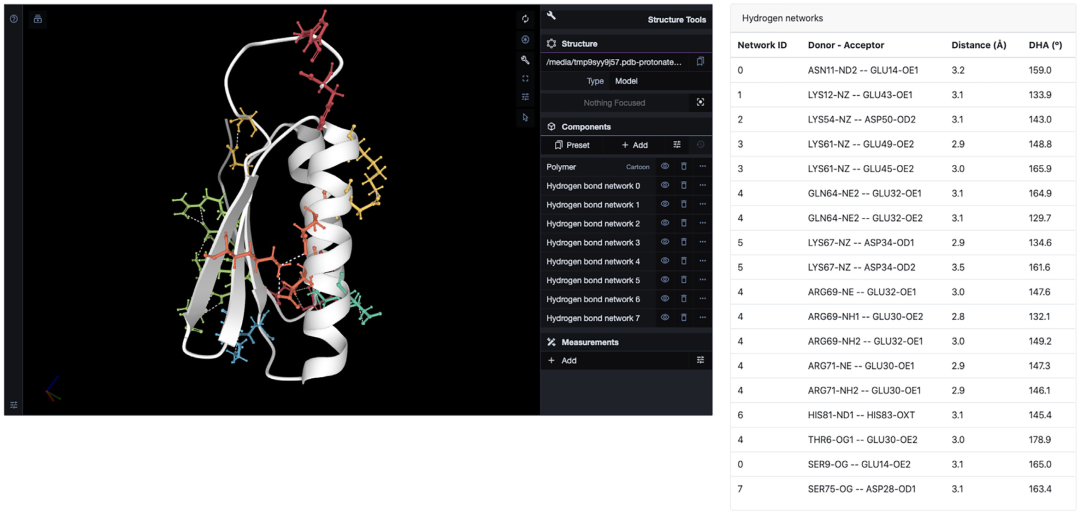
图2. Di-III_14的氢键网络分析。图片来源于:NAR
盐桥
ProteinTool的盐桥应用能够在蛋白质中找到盐桥网络并计算电荷分离参数。作者在https://proteintools.uni-bayreuth.de/salt structure/21n3上计算了Di-11I_14的盐桥(图3)。发现Di-III_14有6个盐桥网络。最大的一个是盐桥4(突出显示),由氢键网络4中的许多残基组成:Glu30、Glu32、Arg69和Arg71;此外还有盐桥3 (Lys67和Asp34),跨越内部的β片。盐桥2连接β4和α2 (Lys61, Glu45和Glu49),而盐桥0则连接αl和α2 (Lys12, Glu13,Glu40和Glu43)。最后,盐桥1通过其残基为Asp50、Lys53和Lys54跨越α2的一半。研究结果与Basak等人的结果基本保持一致,但由于作者更严格地将残基对之间的距离限制在4Å以内,使得结果与Basak等人的有一些差异。
Basak等人认为,不同寻常的大量带电侧链的组成将Di-III_14的折叠机制与天然蛋白质区分开。他们绘制了嗜热Sulfolobus solfataricus几乎整个蛋白质组的带电残基(FCR)与κ的比值,并观察到Di-III_14出现在不同的区域。ProteinTools也能够计算这些参数,FCR为0.35、k为0.25,结果与Basak等人的一致。
图3. Di-III_14的盐桥网络。图片来源于:NAR
接触图
蛋白质接触图展示了所有可能的氨基酸对之间的距离,并提供旋转和翻译固定的蛋白质结构的简写。它们在机器学习方法中得到了广泛的应用,并可以用于重建三维结构或进行蛋白质相似性分析。因此,接触图的快速计算对于各种用途都是有用的。本文作者以Di-III_14为例,计算了其接触图(图4)。
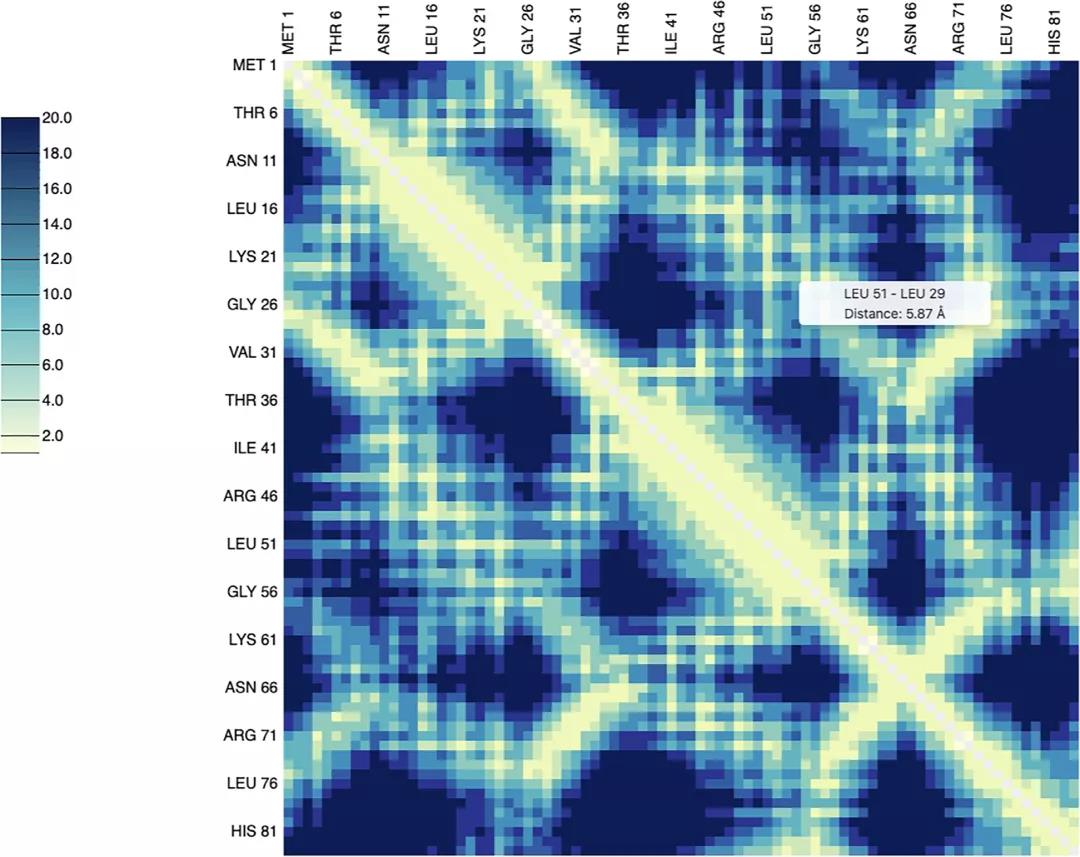
图4. Di-III 14的接触图。图片来源于:NAR
讨论
Web应用程序在过去几年变得特别有用:它们无需安装和学习特定的程序。ProteinTools作为一个模块化工具包来分析蛋白质结构。到目前为止,共实现了四种非常重要的分析工具:疏水簇、氢键网络、盐桥和接触图。这是全球第一款用于计算疏水簇和氢键网络的web服务器。该工具包的模块化特性将使添加其他应用程序到ProteinTools变得容易。作者设想在不久的将来集成一个应用程序,用于突变体的生成和评估其△△G°,以及空腔的计算。
参考文献
Ferruz N, Schmidt S, Hocker B. ProteinTools: a toolkit to analyze protein structures. Nucleic Acids Res 2021;49:W559-W566.