

来自香港科技大学的研究者们综述了针对马尔可夫状态模型(MSMs)在研究蛋白功能动力学中的挑战及代表性的基于机器学习的解决方案。
概述
马尔可夫状态模型(MSMs)在蛋白折叠领域中应用广泛,但在关注局部构象变化的蛋白功能动力学过程中应用较少。功能动力学主要关注蛋白局部的、缓慢的、层级的、特定的构象变化,与蛋白折叠关注蛋白整体的、显著的构象变化有显著不同。目前MSMs用于功能动力学研究有两大挑战:1. 选择合适的结构特征子集(相对于整个蛋白全部结构特征来说)描述这一过程;2. 对构象变化的物理机制的理解能力。作者针对这两大挑战分别介绍了目前最新的基于机器学习的方法,并辅之以具体的案例。
MSMs介绍与其在研究功能性构象变化中的挑战
MSMs通过较多的短时动力学来预测长时动力学,具体来讲,MSMs把构象空间分为许多亚稳态,亚稳态之间变化较慢而内部变化较快,从而使得亚稳态之间的转化具有马尔可夫性质,进而可以如下描述长时动力学:

其中Δ t为时间间隔,P(n Δ t)为状态参量(表示当前时刻亚稳态构象分布),T为状态转移概率矩阵。
MSM建模的关键难点之一在于正确的选择动力学亚稳态,也即选择一组能够合适地描述动力学特征的蛋白结构特征。将这些结构特征降维以后即可得到构象变化过程的广义坐标(Collective Varibales,CVs),继而将短时动力学的结果聚类到亚稳态上。然而,合适的、与功能有关的局部构象变化的结构特征是很难挑选的。此前的挑选过程通常依赖于研究者对研究体系的先验理解,而机器学习方法大大降低了这一过程对经验的依赖。
MSMs的另一难点在于对构象变化的物理机制上的理解。由于MD模拟的限制,MSMs通常会产生数以百计的亚稳态,以使得这一过程符合假设的马尔科夫性质。
功能性构象变化的MSMs建模流程
图1展示了作者推荐的建模流程。在这个流程中,连接已知状态的初始路径通过例如TMD等的方法得到,并进一步使用String method等方法进行优化。之后,根据优化后的路径上的构象进行进一步的更广泛的动力学模拟。根据模拟结果,选取结构特征对功能性构象变化进行描述,这里作者推荐使用Spectral-oASIS等方法自动化的选取一组合适的特征。再之后,例如TICA等降维算法将被用来寻找CVs。通过CVs将动力学轨迹聚类到微状态,聚类方法有多种如Kmeans等。此时建立微状态MSM并使用Chapman-Kolmogorov test进行模型验证,验证式1是否成立,即动力学模拟得到的状态转移过程是否满足该式。另一种Chapman-Kolmogorov test的实现方式是比较MSMs模型得到体系处在某状态的概率与动力学模拟的结果是否一致。
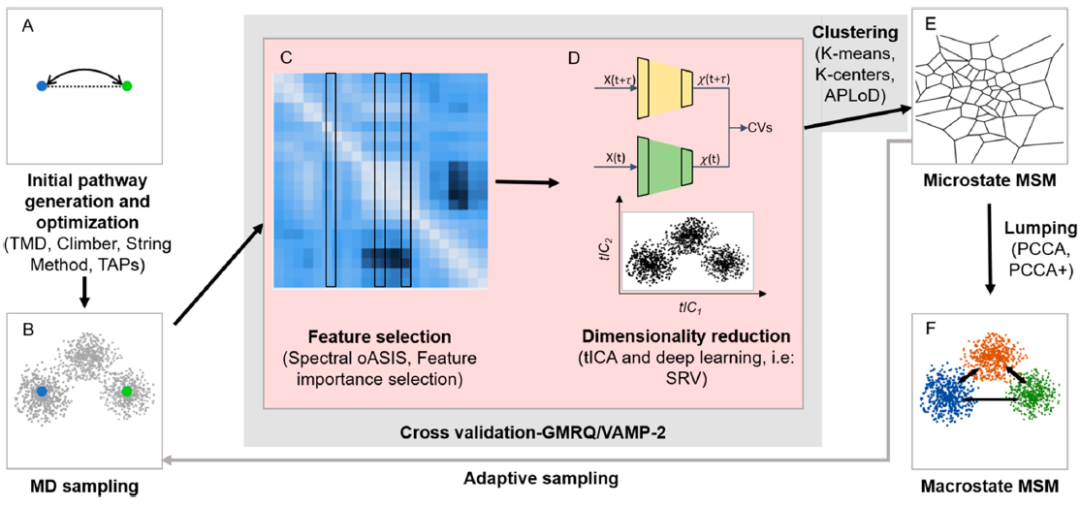
图1. 推荐的MSM建模流程。来源:JACS Au
作者推荐使用交叉检验来避免过拟合及挑选最优参数(如,特征集、CVs的数量、微状态的数量)。交叉检验中可以使用GMRQ、VAMP-2打分等来衡量模型参数的优劣。基于变分原理的这两个打分函数倾向于给出较慢的动力学变化的模型。特别地,GMRQ可以使用状态转移矩阵的本征值计算得到,而VAMP-2可以用时间分割的输入特征的协方差矩阵得到。
如果构象采样不足以建立有效的微状态MSM的话,可以进行适应性采样,并同时重复之前的步骤,直到建立一个有效的模型。
最后,根据是否能快速互变,微状态将会被合并为少数的亚稳定的宏状态。这一步骤可以使用kinetic lumping算法,得到的宏状态MSM将会对理解生物学过程的机制非常有益。尽管如此,想要建立一个马尔科夫的宏状态MSM仍然非常困难,因为状态的时间间隔难以超过动力学模拟的轨迹长度,也即状态之间很难具有完美的马尔科夫性质。此时,qMSMs会非常有用。
近期,MSMs成功地被用于解释一些蛋白的功能性构象变换中。如图2所示的2个例子,分别解释了DNA糖基化酶无法沿DNA双螺旋移动检测DNA损伤的过程,和细菌表面的反向运输蛋白NarK进行NO3-/NO2-的交换的过程与两个精氨酸的靠近有关。
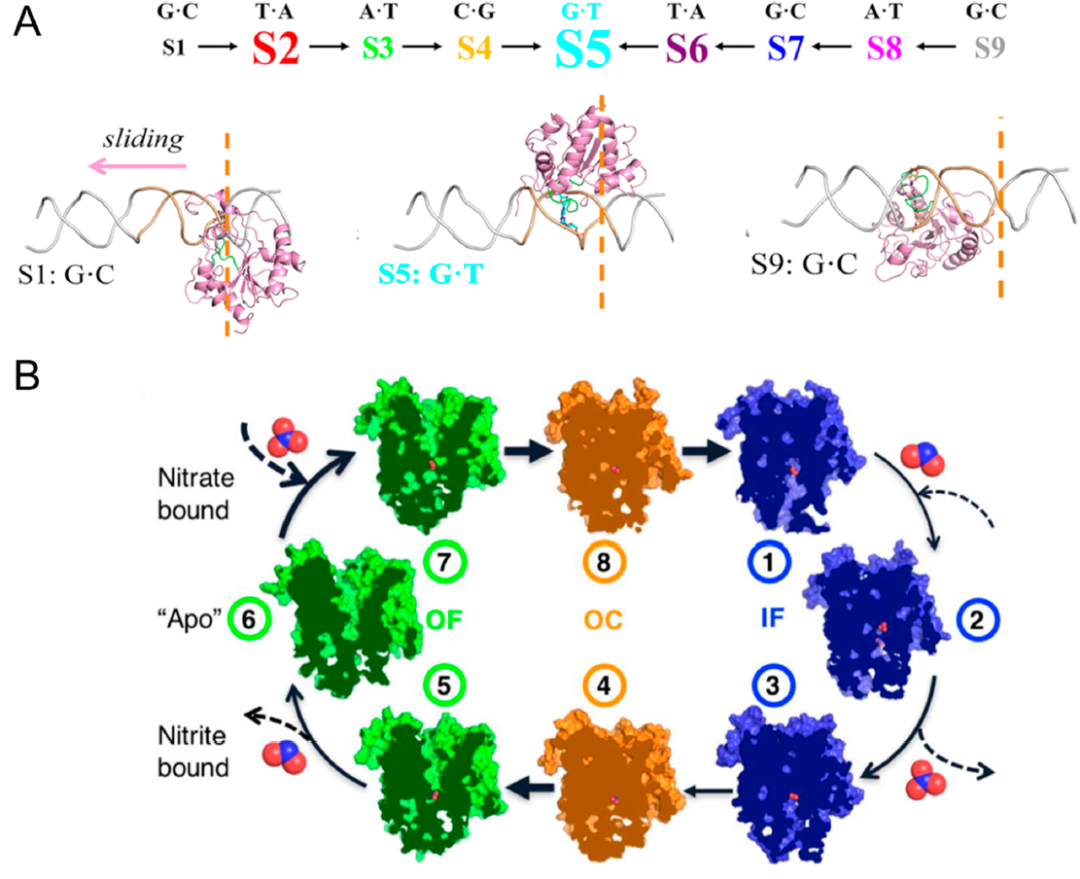
图2. 两个MSMs研究的例子。来源:JACS Au
自动化特征选择与降维
如前所述,有效的选择描述与功能有关的局部构象变换的结构特征是极具挑战性的。这一部分作者介绍了一些新的工具可以自动化的完成特征选择和降维。
自动化特征选择
Spectral-oASIS是特别有用的一种方法。该方法基于Nyström matrix operation theory,仅使用一小部分输入特征就可以重建整个输入的协方差矩阵(图3 AB)。图3 C则展示了一个例子,Spectral-oASIS将25000个特征减少5倍后仍可以精确的描述所关注的构象变化——Trp215的翻转。Sparse-TICA与Spectral-oASIS相似,都是在通过尽可能近似协方差矩阵的关键本征值的情况下来挑选特征子集;但它们的不同之处在于前者使用正则化方法而不是Nyström重建方法。第二种方法则是Stock及其同事发展的,作者称之为“特征重要性选择”。该方法通过使用特征对体系状态(亚稳态的索引)的解释能力进行特征排序,自动选择最高的一批(图3 D)。具体来说,该方法使用输入特征和动力学构象的索引(作为标签)训练决策树,树根部的一批特征则为最重要的特征(图3 E)。第三个方法AMINO是一种具有较大潜力的方法,目前尚未在MSM建模中使用。通过基于共同信息的聚类,Tiwary使用AMINO将428个特征减少到了8个,用于计算配体亲和力。
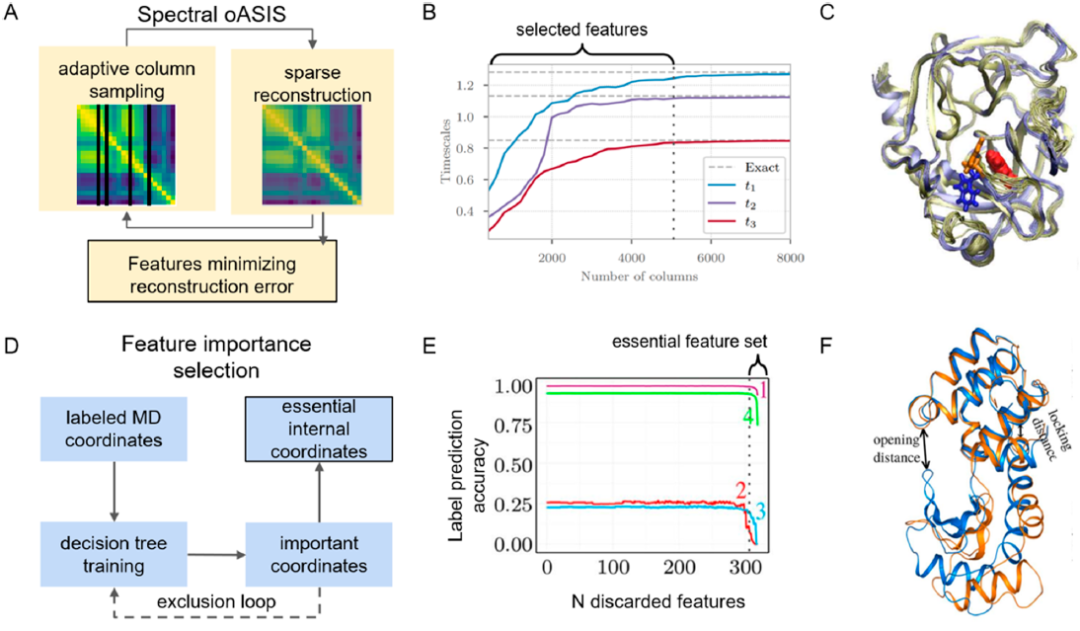
图3. 特征选择。来源:JACS Au
TICA降维算法
TICA是MSM建模中最常用的降维算法,该方法基于对协方差矩阵的特征分解。关键本征向量是对整个体系的线性近似,称之为TICs。作者推荐在Spectral-oASIS或者其他前述方法的基础上使用TICA。
基于深度学习的特征选择与降维
VAMPNets是最早的一批用于MSM的架构(图4)。VAMPNets的一般实现不要求平衡态数据因此不保证系统严格平衡。为了应用在平衡态采样中,VAMPNets的一个变种SRV被提出,通过将协方差矩阵转为对称矩阵强制保证平衡。这些类VAMPNets的深度学习算法用于降维输出少数的CVs进行MSM建模。图4 BCD的例子则展示了一个TICA未能捕捉而SRV可以捕捉到一种模式,同时SRV在交叉检验中更稳定。理论上,基于深度学习的方法可以同时完成特征选择和降维组合为CVs的任务,但作者觉得深度学习也不一定总是万能的,作者建议应用VAMPNets时也可以使用事先选好的特征。
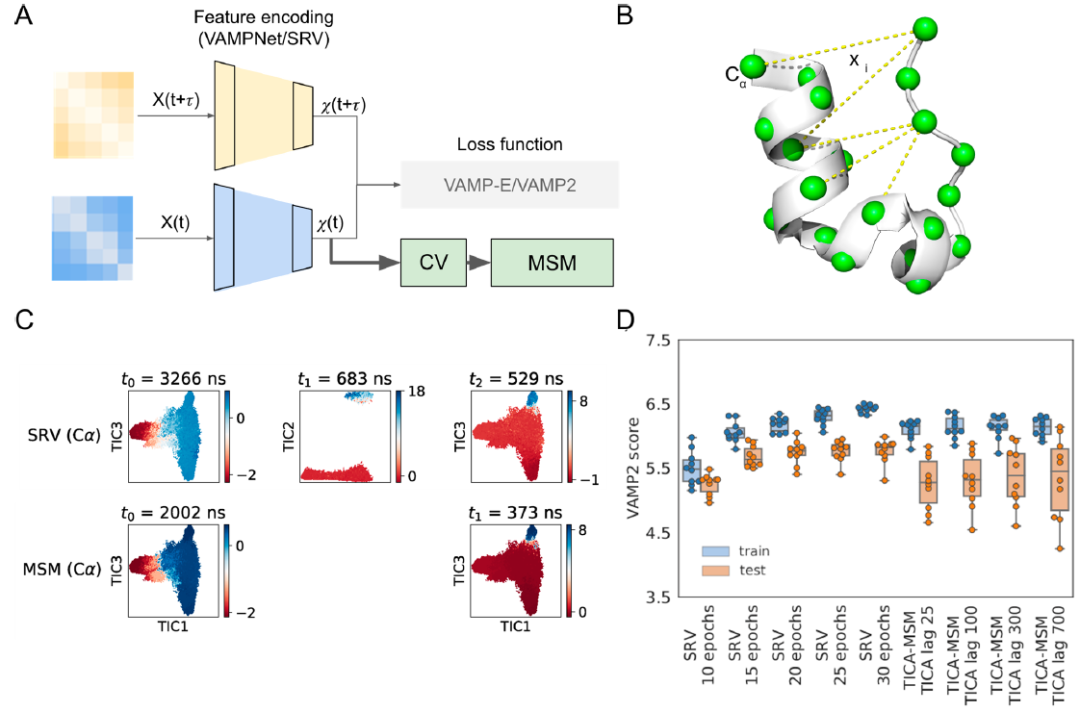
图4. VAMPNets结合交叉验证表现优于TICA。来源:JACS Au
在马尔可夫模型之外:考虑动力学过程中的记忆
如前文所述,MSMs如果只使用很少的状态则难以保证状态间具有马尔可夫性质。对此,作者基于GME方程发展了qMSM方法,引入了显式的记忆核来捕捉状态间的联系:
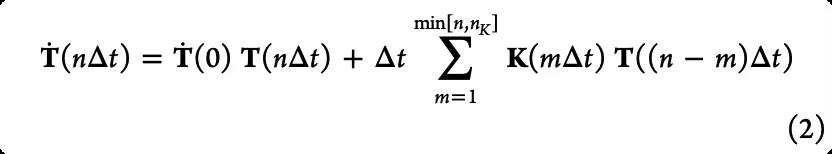
其中记忆核K可以迭代的从状态转移矩阵T和T的导数得到。与MSM不同的是,qMSM使用转移张量K而不是状态转移矩阵T来模拟动力学过程(图5 AB)。图5 CDE展示了一个例子。在RNAP转录复合物中RNAP clamp的打开与转录的启动密切相关,作者展示了qMSM在这一过程的模拟中优于MSM。另外,Tiwary等人近期发展了一种基于长短记忆(LSTM)的算法用来处理动力学过程中的记忆问题,作者期待这类方法此后更好的表现。
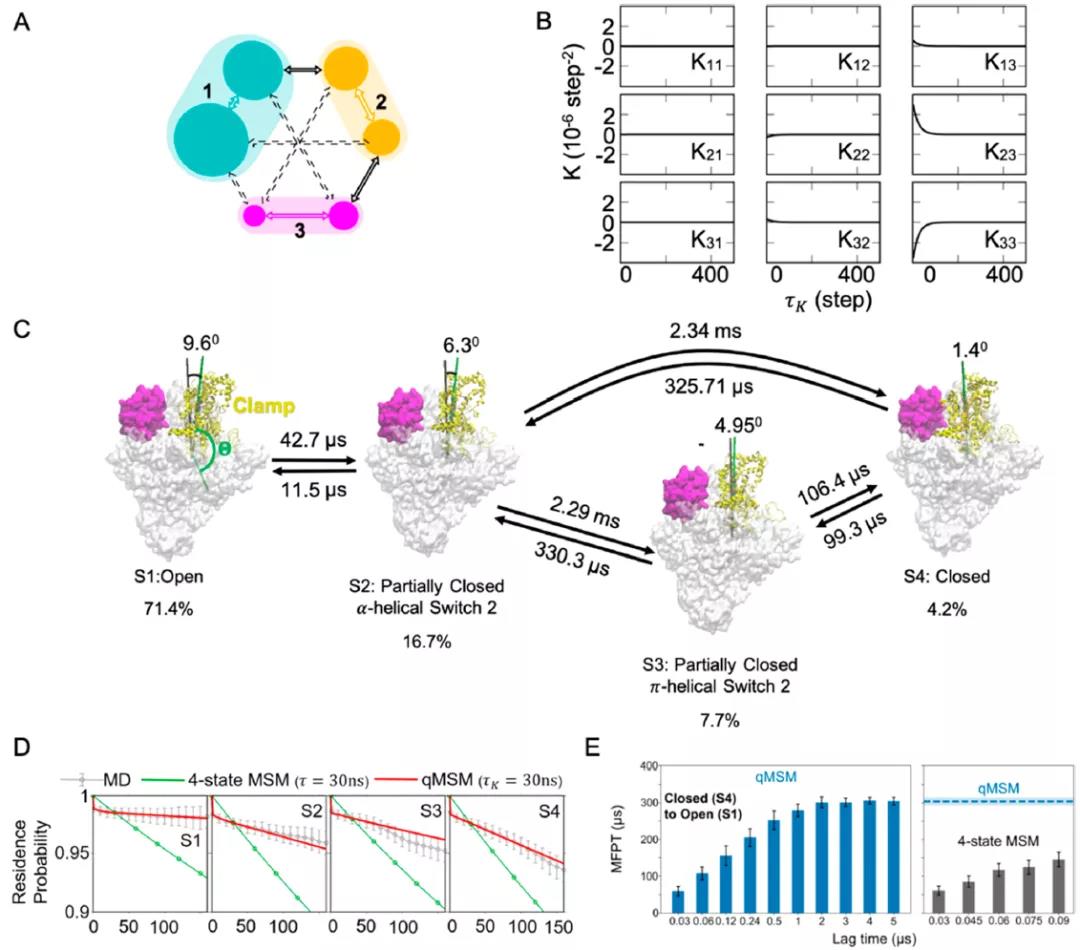
图5. qMSM方法。来源:JACS Au
结论和展望
本文集中介绍了使用MSMs进行复杂生物分子的功能性构象变化研究。针对这类研究中的两大挑战,作者分别介绍了最新的针对性的解决方法。作者强调,本文介绍的大部分特征选择与降维算法都是基于变分原理的,但是找到的过程并不一定与研究目的相关。qMSM,隐式马尔可夫模型和core-set MSMs都可以针对小量的状态进行MSM模拟但仍有可能无法精确解释生物学机理。研究者对研究体系的理解与经验对于更好地发展自动化MSN建模是至关重要的。
参考文献
Konovalov, Kirill A., et al. “Markov State Models to Study the Functional Dynamics of Proteins in the Wake of Machine Learning.” JACS Au (2021).