
摘要
近日,厦门大学、湖南大学和德睿智药(MindRank AI)团队在国际生物信息学重要期刊《Briefings in Bioinformatics》(Impact factor: 8.990 )联合发表了“ A spatial-temporal gated attention module for molecular property prediction based on molecular geometry”。该工作提出了一种基于分子几何学的新型深度神经网络结构,Drug3D-Net,它是一种基于网格的三维卷积神经网络,并具有spatial-temporal gated attention模块。该模型可用于准确预测分子性质以及生物活性。
背景
方法
01
模型概述
Drug3D-Net模型结构如图1所示,主要由三部分组成:
-
Xgrid:3D grid描述符作为模型输入;
-
堆叠的3D CNN和Spatial-temporal gated attention layer;
-
得到分子的3D grid特征表示并用于各种预测任务;
-
对于SMILES数据集中的样本,我们基于原子间的相互作用势能,将3D分子构象离散化成3D grid,再将3D grid输入Drug3D-Net。
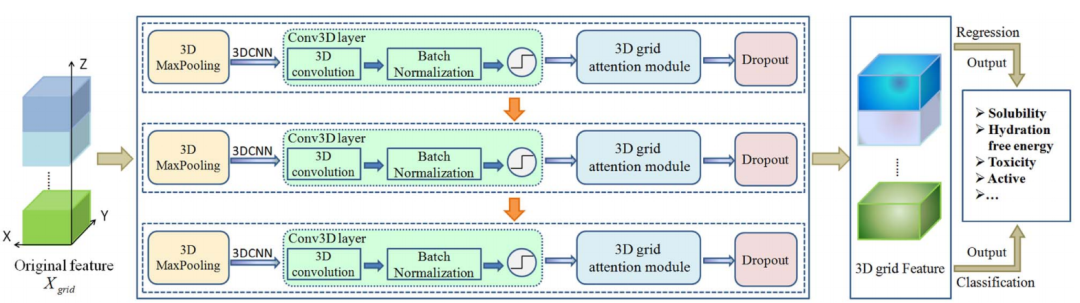
图1 Drug3D-Net模型架构图
如图1,我们搭建了3层带有3D grid attention模块的3D CNN。模型将学习到基于空间结构的3D特征的分子表征,并用于下游预测任务。
02
构建spatial-temporal gated attention模块
Spatial-temporal gated attention模块的详细设计如图2所示。3D grid attention由空间注意力、通道注意力和门控机制构成。空间注意模块通过3D CNN提取3D分子特征并获得空间注意力分数。通道注意力模块通过全连接层获得不同通道上(原子类型)的注意力分数,并采用门控机制来获得全局3D grid attention。
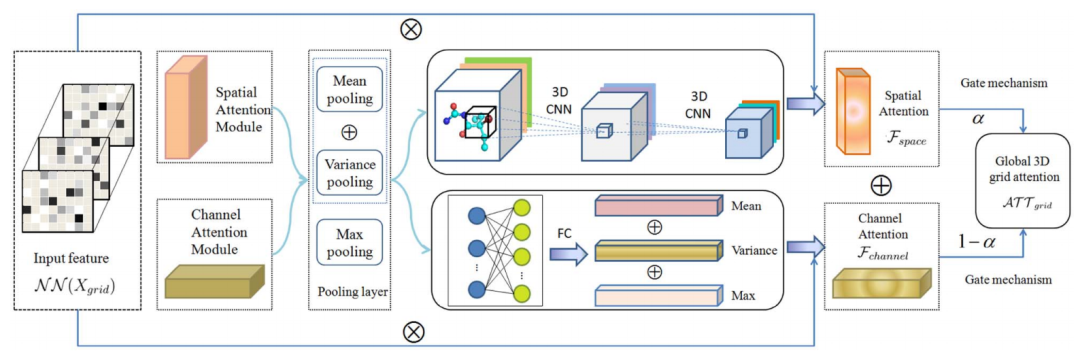
图2 spatial-temporal gated attention模块示意图
数据及其处理
论文中使用的数据集主要有四个,分别为:ESOL,FreeSolv,Tox21以及HIV。
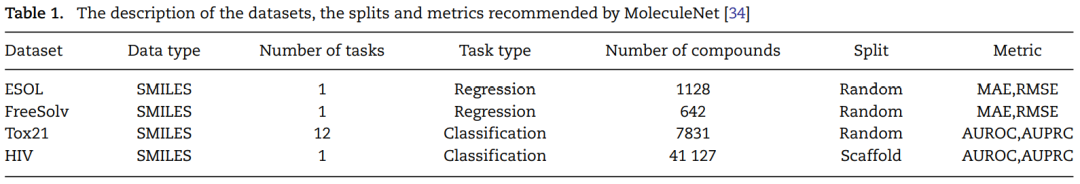
ESOL数据集包含了1128个药物分子的SMILES字符串及其对应的水溶性,分子重量等相关属性信息;
FreeSolv数据集提供了642个小分子在水中的水化自由能;
Tox21数据集记录了7381个药物分子的毒性信息;
HIV数据集收集了超过40000种化合物对HIV病毒复制的抑制作用信息。
其中,ESOL和FreeSolv数据集用于回归任务,Tox21和HIV数据集用于分类任务。
在处理完每个数据集上的数据不平衡问题后,作者通过RDkit包根据分子的拓扑结构信息计算conformers,最终将SMILES字符串转化成48x48x48x10的3D grid表示,这里的10表示10个通道,每个通道上包含一种原子的分布。
实验结果
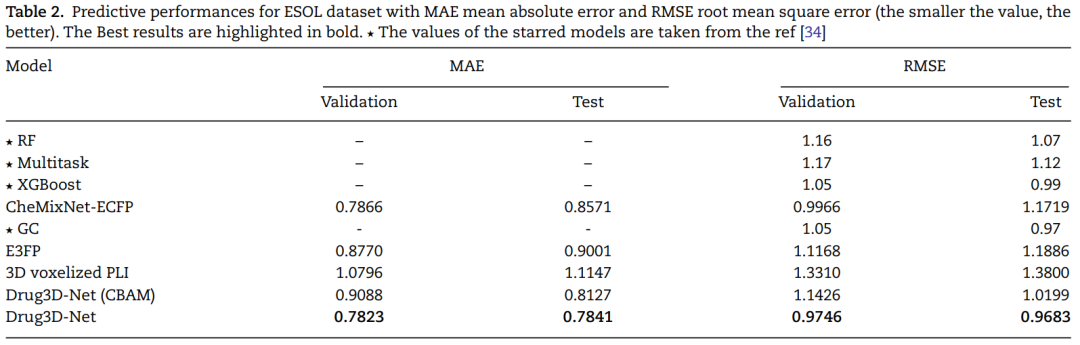
从表2,表3可以看到,在回归任务上,作者提出的Drug3D-Net模型可获得较好的性能。在ESOL数据集上,Drug3D-Net性能大幅优于随机森林(RF),XGBoost这类机器学习算法,以及基于2D分子指纹ECFP的CheMixNet模型、基于3D指纹的E3FP以及基于3D体素的Protein-ligand 相互作用模型。在将CBAM模块替换成作者所提出的3D grid attention模块后,模型效果确实得到了提升,说明相比于CBAM,后者确实能更好地整合空间上的注意力。
在FreeSolv数据集上,Drug3D-Net达到的回归效果也仅次于Graph convolutional Model,作者认为这是由于FreeSolv数据集的数据量过小,不能提供更丰富的结构信息所导致的。但相比于其它几个baselines,这里的差距是微不足道的。
从表4和表5中可以看到,在分类任务中,Drug3D-Net也达到了非常出色的效果,超过了一众机器学习算法以及其它基于图和3D的分子表示方法。作者认为,优越的性能一方面来自于3D grid attention机制,另一方面则是因为作者做了保证3D旋转不变性的数据增强操作。
讨论
在本篇文章中,作者提出了基于spatial-temporal gated attention模块的3D CNN模型来提取3D grid的特征,并将其整合到“端到端”的Drug3D-Net模型当中。在多个对比实验中,作者所提出的模型在回归和分类任务上都达到了state-of-the-art的效果。与CBAM模块的对比实验结果,进一步证明了作者提出的3D grid attention机制能更好地整合空间上的注意力信息。