
引言
蛋白质工程(Protein Engineer)具有巨大的学术和工业潜力。然而,在浩瀚的蛋白质序列空间中进行搜索的能力限制了我们的设计,但是蛋白质功能序列空间却非常有限。在寻找功能序列时,工程师们必须警惕“你得到你筛选的”这一普遍存在的格言,这一格言告诫人们不要使用可能与最终设计目标不完全一致的功能分析来过度优化蛋白质序列。并且通过早期高通量(>104个变异体)实验确定的最佳候选者,在较高保真度的后期分析的验证中往往会失败。此外,许多种类的蛋白质根本不存在高通量分析,因此无法进行筛选和定向进化。基于此,蛋白质工程受到严重的限制。本文介绍了一个机器学习的范例,可以使用24个突变功能性分析突变体来构建一个精准的虚拟度适应空间,以便通过虚拟定向进化来筛选千万级别的序列。通过来自于自然蛋白序列空间中的信息,作者的模型可以学习一个潜在的‘非自然’展示空间,减少搜索非功能性序列空间。随后的low-N监督学习可以帮助提高活性。总之,此模型可以在不牺牲高通量的前提下,高效利用资源进行高保真性分析,并有助于蛋白工程加速进入发酵罐等阶段。
结果
1. Low-N 蛋白工程的一个范例
为了满足有监督的深度学习(通常数量级应该大于106)的巨大数据需求,机器学习指导蛋白质设计方法必须收集高通量的实验数据或完全放弃深度学习。作者利用UniRep中对功能蛋白序列的现有知识,来减少数据需求,实现 low-N设计。作者使用了UniRep,一个在大型未标记蛋白质序列数据集上训练的深度学习模型。UniRep仅从序列开始,就学会了提取蛋白质的基本特征,包括生物物理、结构和进化信息,形成一个完整的统计摘要。
2. 步骤
对于给定靶点的low-N工程化设计,作者的步骤包含以下几步:
(1) UniRep在超过2000万个原始氨基酸序列上进行全局无监督预训练,以提取功能蛋白的一般特征;
(2) UniRep对目标蛋白相关序列的无监督微调(evotining)以了解目标家族的不同特征;
(3) 野生型(WT)靶蛋白的Low-N个随机突变体的功能表征,以训练使用eUniRep表示作为输入的简单监督模型;
(4) 基于蒙特卡罗得马尔可夫链来进行虚拟定向进化;
(5) 预测改进之后的功能序列的实验表征。
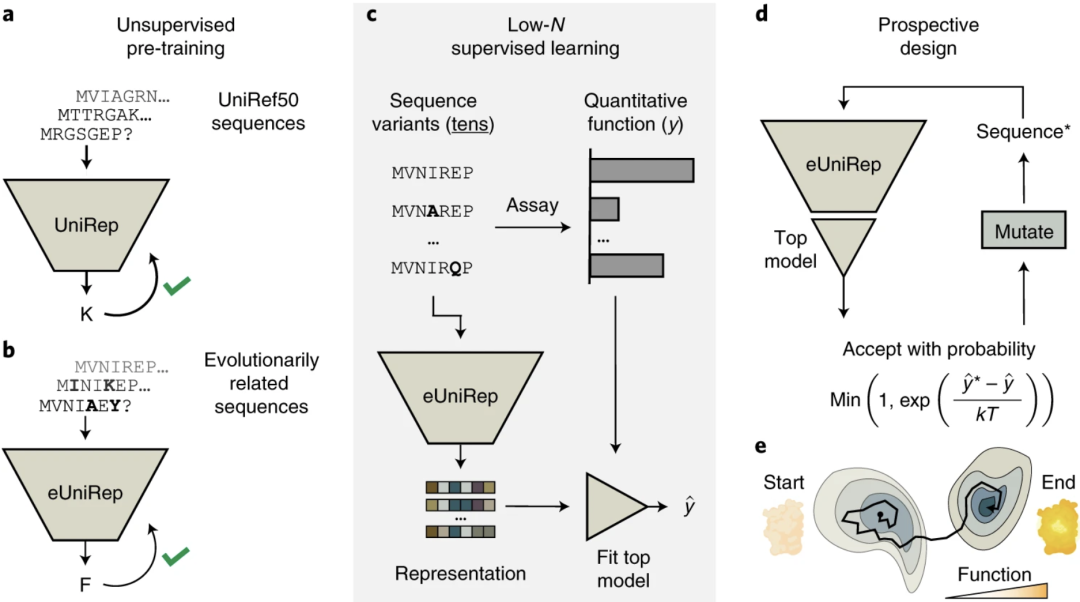
图1. 给定靶点的low-N工程化设计步骤
图片来源于Nature Methods
作者首先尝试对原始avGFP的荧光强度进行Low-N优化。设计过程包括了从易错PCR中随机取样的N = 24或N = 96的突变体,代表序列空间,训练出了一个顶层模型,然后对其进行虚拟定向进化来产生300个优化设计,信任半径为15个突变。总共产生12000个序列设计。
4. TEM-1 β-lactamase的Low-N工程化设计
接下来作者将其推广到 TEM-1 β-lactamase,用其来优化蛋白功能,使用单个突变体作为训练数据。作者设计了一个81个氨基酸,四个螺旋(但是不包括催化丝氨酸S70的中心螺旋),并设计提出了七个突变信任半径。在GFP中,作者为每个Ntrain和表示模型生成了300个设计,并将这个过程复制了5次。Low-N工程对于酶生物催化剂的设计帮助巨大。
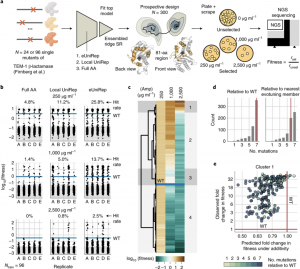
图2. TEM-1 β-lactamase的Low-N工程化设计
图片来源于Nature Methods
讨论
本研究为 low-N 蛋白工程提供了一个可推广的范例。通过从全局和局部序列空间中提取信息,随后重复地利用N = 24来训练突变体和进行虚拟筛选,形成1000多个新的设计(大于WT)。这是迄今为止机器学习引导蛋白质功能优化中最好的案例。作者仅以24个avGFP的随机突变体作为训练数据,设计了新的荧光蛋白(FPs),可以与高通量、高保真蛋白质工程的产物sfGFP相媲美。
代码下载
UniRep: https://github.com/churchlab/UniRep
参考文献
Biswas, S., Khimulya, G., Alley, E. C., Esvelt, K. M., & Church, G. M., Low-N protein engineering with data-efficient deep learning. Nature Methods, 2021, 18, 389–396. DOI: 10.1038/s41592-021-01100-y.