引言
自然界除了作为生命基石的20种原型氨基酸外,还有多种表现出不同生理功能的非典型氨基酸(ncAAs),它们同样可广泛用作生物活性产物的中间体。一项对FDA批准的药物的研究分析表明,在最畅销的200种药物中,大约12%至少含有一种ncAA构成成分。然而,尽管ncAAs具有广阔的应用前景,但由于需要严格的立体选择性和官能团相容性,其合成仍然是一个挑战。因此,对于ncAAs来说,简单、可持续、具有成本效益和可扩展的流程,是非常之必要的。手性ncAAs,可以通过碳-碳双键的氢胺化反应得到,这是合成化学中最热门的反应之一。此外,不对称C-N加成在一定程度上可以很容易合成ncAAs,而且理论收率接近100%,虽然这个过程通常要求繁琐的保护,解除保护步骤和昂贵的催化剂或手性助剂。在这种情况下,自然界通过使用氨裂解酶,来催化可逆的C-N键的裂解和形成,从而规避了上述缺点,比如图1所示天冬氨酸酶(AspB)和苯丙氨酸解氨酶(PAL)。
在这篇文章中,作者在基于机制计算的重设计策略下,重新设计并重新构建了天冬氨酸酶(AspB,一种高度特异性的氨裂解酶)的活性位点,生成了一种用于C-N键形成的多功能氢胺化生物催化剂(具有亲核试剂和亲电试剂交叉兼容性),并在制备脂肪族、芳香族和非天然带电氨基酸物质中展示了其合成价值。
先睹为快
来自中国科学院微生物研究所的吴边等研究者,成功地重新设计了芽孢杆菌天冬氨酸酶(AspB)的活性位点。研究者试图解决长期以来的挑战,即通过执行计算协议,允许酶在功能上有比较大的跳跃,沿适应度遍历非活性序列空间,从而创建出具有非原生底物交叉兼容性的酶。
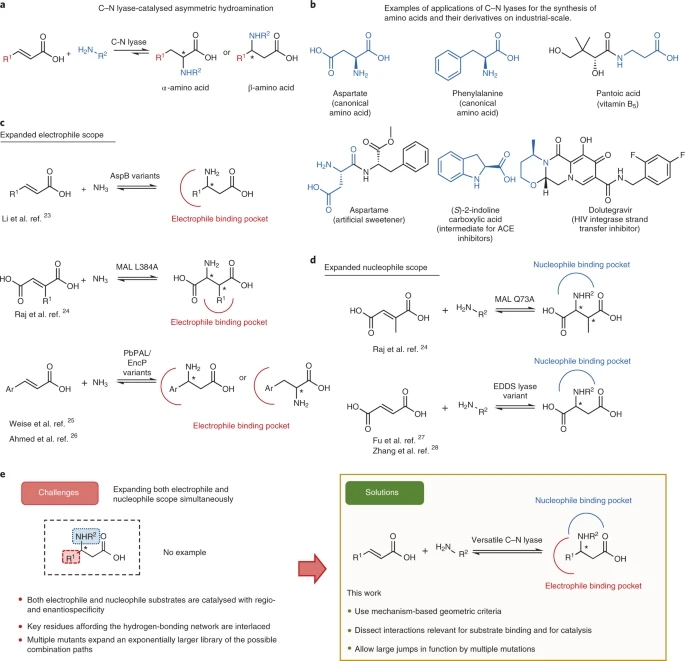
图1. 通过氢胺化反应合成ncAAs的生物合成策略
图片来源于Nature Catalysis.
AspB的计算重设计
AspB的反应机制的第一步是由SS Loop上的Ser318,夺取底物Asp的Cβ原子上的质子,形成的烯酸酯阴离子中间体的羧酸基通过残基Thr101、Ser140、Thr141和Ser319形成的氢键网络来稳定,同时氨基通过Thr101、Asn142和His188侧链的氢键作用固定在亲核口袋中。原来的α-羧酸结合口袋由残基Thr187、Met321、Lys324和Asn326组成,这些残基在之前的研究中被取代,以靶向不同的亲电试剂。初步的胺基不对称氢胺化实验表明,Asn142是一个不重要的残基,而Thr101和His188则是关键性的残基(Thr101和His188被取代为丙氨酸后,引起了催化活性的显著降低)。此外,从结构上看,残基Ala99、Leu358和Glu362创造了一个受阻环境,以阻止较大的亲核试剂进入其理想的反应姿态。
基于以上发现,研究者使用Rosetta酶设计,扩展了AspB的亲核谱。具体来说,在保持酶的整体折叠性的情况下,Ala99、Asn142、Leu358和Glu362被突变为体积较小的残基。通过分子动力学(MD)模拟,生成了一系列不同的起始点,用于Rosetta酶设计。随后,为了提高设计库的质量和减少筛选工作量,对Rosetta酶设计获得的结果进行了计算筛选,以增加由多个独立MD模拟取样的构象空间。此外,酶-底物复合物在β-羧酸氢键相互作用网络中花费的时间的比例,是用几何标准的近攻构象(NACs)来定量的,这是接近过渡态结构的构象。因此,最后根据总能量得分、约束惩罚得分和NAC频率,选择少量设计进行实验表征(具体的活性口袋重设计流程如图2所示)。
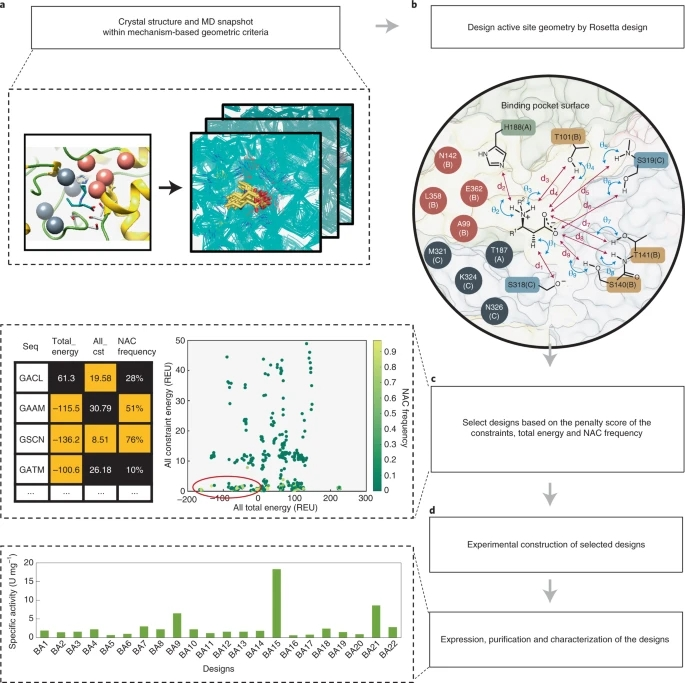
图2. AspB的计算活性位点的重设计流程图
图片来源于Nature Catalysis.
亲核试剂与亲电试剂矩阵的共轭加成
研究者首先评价了巴豆酸(1)与胺衍生物的不对称氢胺化反应。作为计算设计的基准反应,研究者选择了能够为后期多样化提供特殊机会的底物(烯丙胺,h)或具有相对较大取代基的底物(环丙胺,j)。将Ala99、Asn142、Leu358和Glu362同时替换为较小的残基(A99替换为G, N142替换为GAVSTC, L358替换为GAVIMSTCDNH, E362替换为GAVLIMSTCDNH),经由电脑模拟建立了B19酶的突变体库。对1h的22种设计和1j的16种设计进行实验验证,鉴定出37个突变体,其中BA15设计(含A99G-N142S-T187C-M231I-K324L-N326A-L358V-E362M突变体)对1h和1j合成的特异性活性最高,在2 h内获得了很好的转化率(>99%),对照实验表明,在没有酶的情况下,胺不与巴豆酸发生反应。如图3所示,大多数胺被BA15有效地转化为,具有>96%转化率的光学纯产物。对于强亲核试剂乙二胺(m),可发生弱自发反应,产物的对映选择性下降至90% e.e。此外,进一步地结构分析表明,A99G-N142S-L358V-E362M突变,会产生更大的胺结合口袋,在保留范德华相互作用的同时,允许更大的胺基团以不同的方向结合(图3)。因此,除了简单氨外,还可以接受带有带电或大取代基的非天然胺。更重要的是,考虑到亲核口袋的构象变化与亲电子口袋的不冲突,这就意味着亲核口袋和亲电子口袋可直接结合。
为了评价亲核口袋和设计的亲电子口袋的相容性,研究者将重新设计的亲核结合口袋引入了AspB野生型(AA15设计包含A99G-N142S-L358V-E362M突变)、工程酶P1 (PA15设计包含A99G-N142S-T187C-M231I-K324L-N326C-L358V-E362M突变)和F29 (FA15设计包含A99G-N142S-L358V-E362M突变)A99G-N142S-T187C-M231V-K324I-N326C-L358V-E362M突变)中,来催化多种亲核供体到亲电子受体的共轭加成。如预期的那样,含乙基的不饱和氨基酸与评价的胺,进行了有效的氢胺化反应,不仅得到了相应的产物,而且它们的转化率(>94%)和对映选择性(>99% e.e.,2m除外)也都不错。对于芳香底物,由于其溶解性低,必须使用较低的浓度。取代基相对较小的胺的转化率较低。尽管如此,肉桂酸被证明是甲氧胺的有效偶联伙伴,使产品转化率达到97%。带有吸电子基团/供电子基团的芳香丙烯酸酯也提供了理想的产品,具有令人满意的转化率,这表明重新设计的酶,可以耐受各种通常在药物制剂中遇到的功能化基团。

图3. 计算重设计的AspB的催化底物范围
图片来源于Nature Catalysis.
讨论
在现有的酶工程中,理论和计算方法已经证明,其可以成功地提高人类识别酶的卓越性能起源的能力。通过探索巨大序列空间中的一小部分,已经有一些成功的研究案例,聚焦于重塑活性位点,以适应单个底物。然而,很多生物催化剂,如C-N裂解酶,亚胺还原酶和醛缩酶,却是催化多底物的交叉偶联。因此,问题是,是否合理的计算模型可以适用于更具有挑战性的任务,其中酶的操作不再局限于一小组残基,而是扩大到处理集体突变同步排列的整个活性位点。在这里,研究者通过改变极其特殊的酶AspB的底物识别模式,来解决上述的挑战。这一目标是通过基于机制的计算协议实现的,通过分析与结合和催化相关的邻近相互作用,从结构分析和MD模拟中检索基本的催化几何准则,并且允许在序列空间中有较大的跳跃,同时容纳多个同时发生的突变和底物之间的相互作用。这一策略的显著优势,在于尽量减少实验,同时保持对突变间协同作用的探索。改造后的酶拥有多达8个突变分别涉及空间相邻的位置以及活性位点。显然,如果没有计算的帮助,这种酶活性位点的大规模修饰,要么通过艰巨的理性的检查,要么通过极其劳动密集型的实验分子进化。
展望与结论
综上所述,研究者提出了一种通用的可构造C-N裂解酶的平台,研究者预计,这种有效的生物催化剂优化平台的进一步发展,可能会打开新的机会,使结构多样的构建块通过C-N连接,用于ncAAs及其衍生物的合成。因此它不仅在合成化学和药物化学中提供了用途,而且为未来合成生物学的发展奠定了分子基础。
参考文献
Cui, Y., Wang, Y., Tian, W., Bu Y., Li T., Cui, X., Zhu, T., Li, R., and Wu, B., Development of A Versatile and Efficient C–N Lyase Platform for Asymmetric Hydroamination via Computational Enzyme Redesign, Nature Catalysis, 2021, 4, 364-373. DOI: 10.1038/s41929-021-00604-2.