先睹为快
靶点
AmpC和D4多巴胺受体
筛选化合物库
ZINC以及自定义分子枚举库
计算方法
ECFP4分子指纹,分子骨架,分子对接(Docking)
计算软件
分子相似度:https://github.com/docking-org/ChemInfTools
分子骨架:https://www.molinspiration.com/
分子对接:DOCK
单位与作者
Brian K. Shoichet, UCSF
课题组网站:http://bkslab.org
John J. Irwin, UCSF
课题组网站:http://irwinlab.compbio.ucsf.edu
Bryan L. Roth, UNC at Chapel Hill
课题组网站:http://pdspdb.unc.edu/rothlab
杂志
Nature
简介
Nature文章,今年2月份发表,是迄今为止规模最大的一次虚拟筛选,分子库数目达到了亿级别。两个蛋白靶点为AmpC和D4多巴胺受体,AmpC的抑制剂被优化到了77nM,而D4多巴胺受体抑制剂研究中发现了30多种亚微摩尔级别的抑制效果,最好的一个达到了pM级别。
化学空间的扩展
Bohacek及其同事提出,有超过10的63次方种类药分子。显而易见的是,潜在类药分子的数量要比实体药物库数量高许多个级别。扩大化学空间,作者的化学分子库来源于两个方面一个是ZINC,一个是分子枚举库。分子枚举库是为了克服化学可及性而创造出来的虚拟分子库。作者使用了130个化学反应以及来源于Enamine的70000个Buliding blocks来构建分子枚举库。
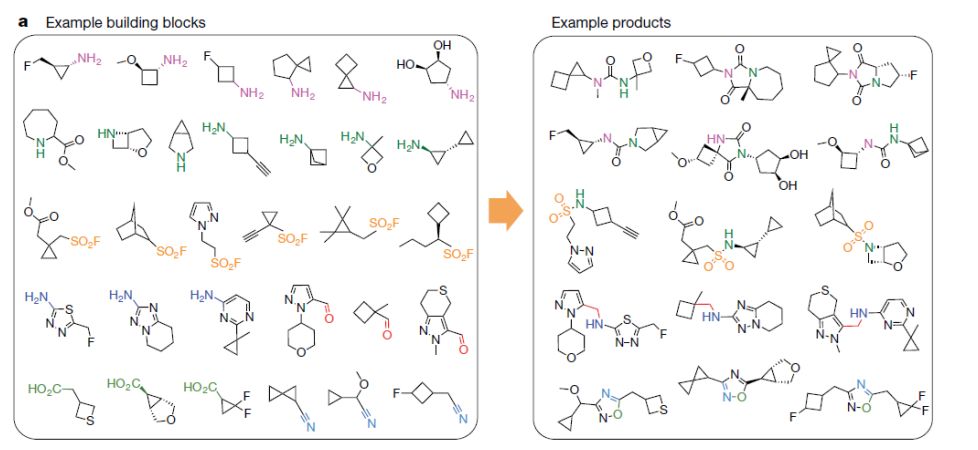
图1. 分子枚举库
图片来源Nature
分子对接
1.38亿个分子被用于对接,配体与受体大约产生了70万亿个复合物模拟结构,计算总计需要约43,563个核时。拿到对接后的打分数据,排名前列的分子通过拓扑聚类以及骨架分析来减少冗余。为了发现新的化学表型,与ChEMBL库中已知的D4 受体抑制剂进行了比较,ECFP4指纹相差在0.35以内的被排除。在所选择的589个分子中,成功合成了549个(93%)。在top 1,000 ranking clusters,通过视觉检查选择124个分子,通过对接分数也选择了444个。在功能实验中,几种排名较高的分子被证明为有效的。例如,ZINC621433143的IC50达到了2.3nM,ZINC465129598和ZINC270269326的IC50分别是24nM和17nM,而ZINC464771011的IC50达到了10nM。
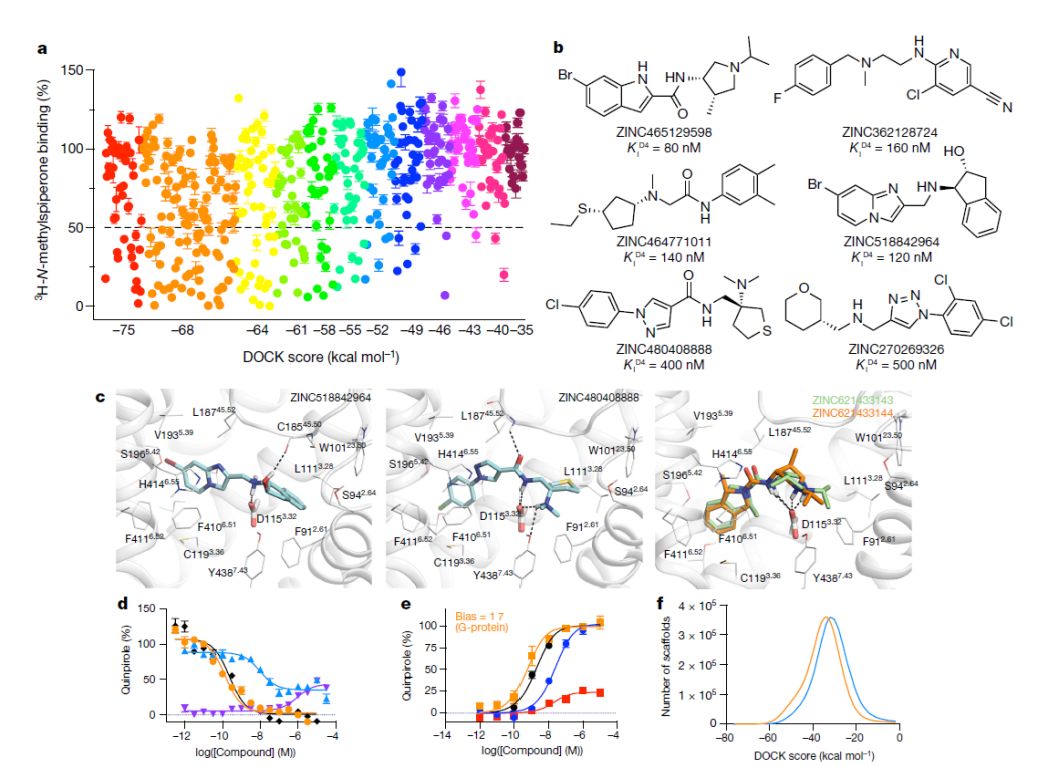
图2. 测试分子对接靶向D4受体的549个分子
图片来源Nature
编者按
文章的思路是虚拟筛选的经典策略,不过重心放在了化学枚举库的构建以及后期的分子聚类分析之间,按照此文的思路,可以应用到日常的虚拟筛选工作流中,不过,如何构建一个独特的分子枚举库就是一个很有趣的事情了。
参考文献
Lyu, J., et al., Ultra-large library docking for discovering new chemotypes. Nature, 2019. 566(7743): p. 224