
一个机器学习(ML)框架可利用基于网络的分析来识别ICI治疗生物标志物(NetBio),并做出稳健的预测。
背景介绍
在过去的几年中,免疫检查点抑制剂(ICIs)大大改善了癌症患者的临床治疗。不过一个主要的局限性是,只有少数患者对免疫治疗有反应(在实体肿瘤中~30%),且ICI治疗后可能发生毒性。急需一种方法来识别在给药前就能检测免疫治疗应答的生物标志物,提供ICIs临床使用的信息,并提高癌症患者的生存率。
许多研究报道PD-L1表达与非小细胞肺癌的ICI反应呈正相关。然而,令人惊讶的是,其他研究报道PD-L1表达与ICI治疗反应之间没有显著相关性,一些研究甚至显示ICI应答者表现出较低的PD-L1表达水平。需要确定新的生物标记物,以稳健地预测免疫治疗反应。因此,必须开发出成功的方法来识别ICI治疗患者的生物标志物,并最终使ICI治疗的效益最大化。
网络生物学,为识别可靠的生物标志物提供了一种强有力的手段。具有相似表型作用的基因倾向于在蛋白质-蛋白质相互作用(PPI)网络的特定区域共定位,这一趋势已被用于识别比单基因方法更能预测表型结果的基因模块。此外,研究者之前曾报道过,利用患者衍生的类器官模型的药物基因组学数据,可以通过网络邻近性识别预测癌症患者总体生存的药物反应生物标志物。总之,证据表明,基于网络的方法提供了预测性和低噪声的生物标志物,但该方法预测大量癌症患者对ICI治疗反应的有效性尚未被验证。
主要内容
韩国浦项科技大学&延世大学的Sanguk Kim等人提出了一个机器学习框架,该框架利用基于网络的分析来识别ICI治疗生物标志物(NetBio)。研究者整理了700多例ICI治疗的患者样本,包括临床结果和转录组数据,并观察到基于NetBio的预测,准确预测了三种不同癌症类型的ICI治疗反应——黑色素瘤、胃癌和膀胱癌。此外,基于NetBio的预测优于基于其他传统ICI治疗生物标志物的预测,如ICI靶标或肿瘤微环境相关标志物。
基于网络的免疫治疗反应预测综述
基于之前的工作,作者通过选择ICI靶标附近的通路来识别与ICI反应相关的生物通路(图1a, b)。STRING PPI网络(STRING score >700)包括16,957个节点和420,381条边。首先,研究者采用网络传播,使用ICI靶标(例如,nivolumab的PD-1或atezolizumab的PD-L1)作为种子基因,在网络上传播ICI靶标的影响(图1a)。网络传播的一个特点是,越接近ICI靶标的节点影响分数越高。接下来,研究者选择了影响分数高的基因(top 200基因),并鉴定了这些基因富集的生物通路(Reactome pathway)(图1b)。通过使用所选择的生物通路来预测免疫治疗反应,并将这些通路视为网络生物标记物(NetBio)。
为了进行基于ML的免疫治疗-反应预测,研究者使用NetBio作为输入特征;阴性对照使用了基于基因的生物标志物(即免疫治疗靶基因)、基于肿瘤微环境的生物标志物或从数据驱动的ML方法中选择的通路(图1c)。为了使用监督学习来训练ML模型,研究者使用不同的训练数据集和测试数据集的组合来广泛地衡量预测性能的一致性。研究者进行了(i)研究内预测,其中训练和测试数据集来自单个队列;或(ii)跨研究预测,其中两个独立的数据集用作训练和测试数据集(图1d)。
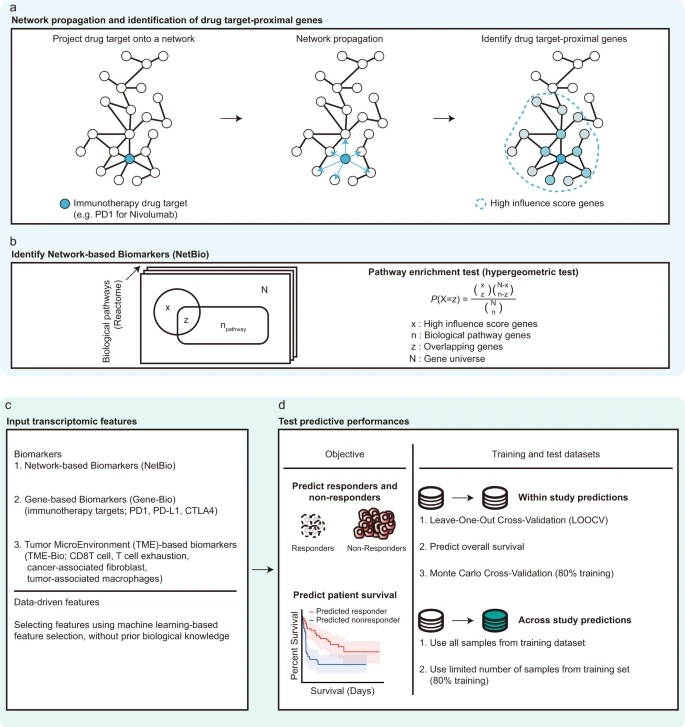
图1. 一种基于网络的机器学习方法来识别免疫治疗相关的生物标志物。图片来自Nat. Commun.
研究内交叉验证表明,基于NetBio的ML可以对ICI治疗反应和总生存期做出一致的预测
NetBio的转录组可以做出一致的预测性能来预测ICI反应(图2)。相比之下,当使用药物靶标表达时(例如,PD-1用于尼鲁单抗和派姆单抗,PD-L1用于atezolizumab, CTLA4用于伊匹单抗治疗的患者),预测性能较差。使用NetBio训练的ML模型,在所有4个数据集中始终保持准确的预测,而使用药物靶标表达水平做出的预测不太一致,药物靶标仅在黑色素瘤队列中可准确预测。
此外,在三个具有总生存数据的数据集中,使用基于NetBio的ML预测为ICI应答者的患者的总生存时间持续延长。
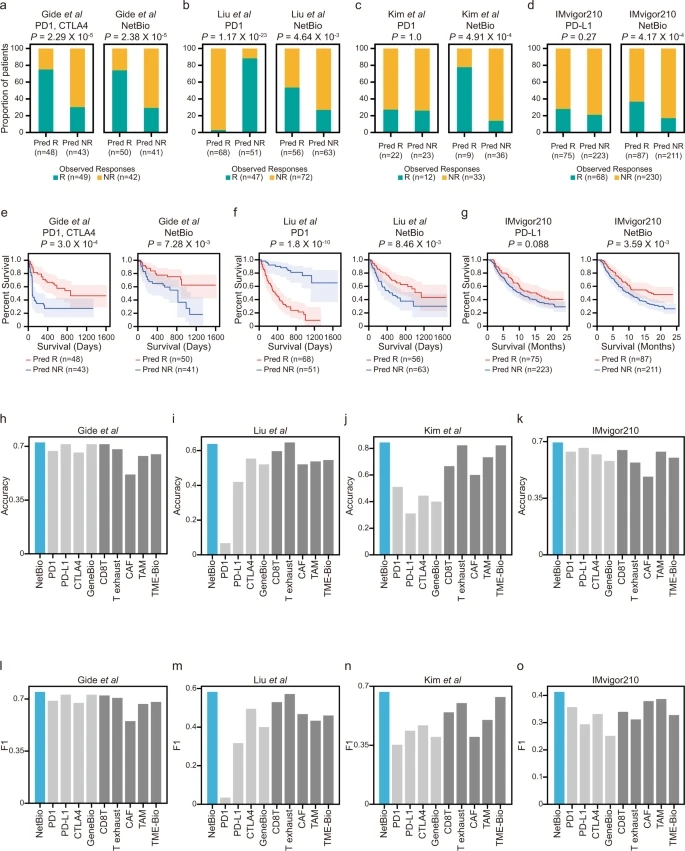
图2. 免疫治疗患者药物反应和总生存期的预测。图片来自Nat. Commun.
接着,研究者将NetBio的预测性能与之前确定的其他ICI相关生物标志物进行了比较,发现在大多数情况下,研究者的方法在所有四个癌症数据集上都更好(图2h-o)。
跨研究预测表明,基于NetBio的ML可以在其他独立的黑色素瘤数据集中做出一致的预测
一个精确的ML模型的关键方面包括:(i)它的泛化能力;(ii)它在训练样本很少的情况下的一致性。为了测试ML模型的泛化性,研究者使用Gide等人的黑色素瘤数据集来训练ML模型,并在三个独立的黑色素瘤数据集上测试其预测性能(图3a)。基于NetBio的ML在两个外部数据集中显示AUCs >0.7(图3b, c;Auslander AUC = 0.79;Prat AUC = 0.72),其余数据集为0.69(图3d;Riaz)。与基于BetBio的ML相比,使用其他生物标记物的预测显示出高度变化的预测性能(图3b-d)。
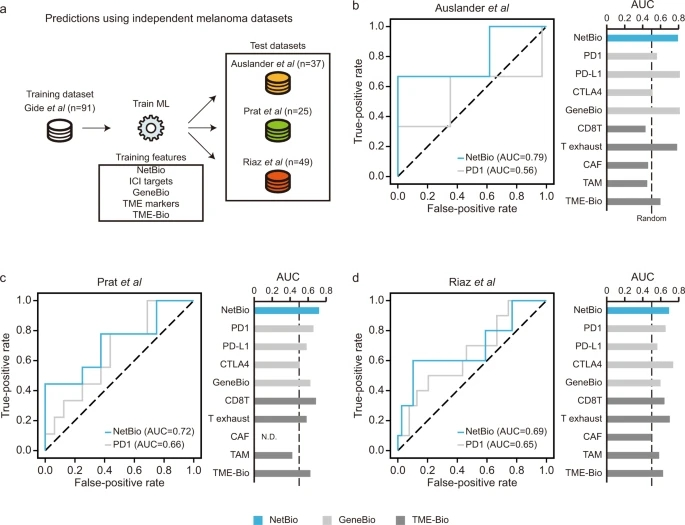
图3. 三个独立的黑色素瘤数据集的预测性能。图片来自Nat. Commun.
基于NetBio的预测优于纯数据驱动的特征选择方法
与纯数据驱动的ML预测相比,基于NetBio的ML模型,能够持续提高预测性能(图4)。具体来说,对于数据驱动的ML模型,研究者选择了训练数据集中最能区分应答者和非应答者的K个数字特征(其中K等于NetBio的数量),并使用所选特征训练ML模型(图4a)。在11个不同的任务中,发现基于NetBio的预测表现出明显优于基于ML的特征选择(图4b;t检验P = 3.3×10−3)。
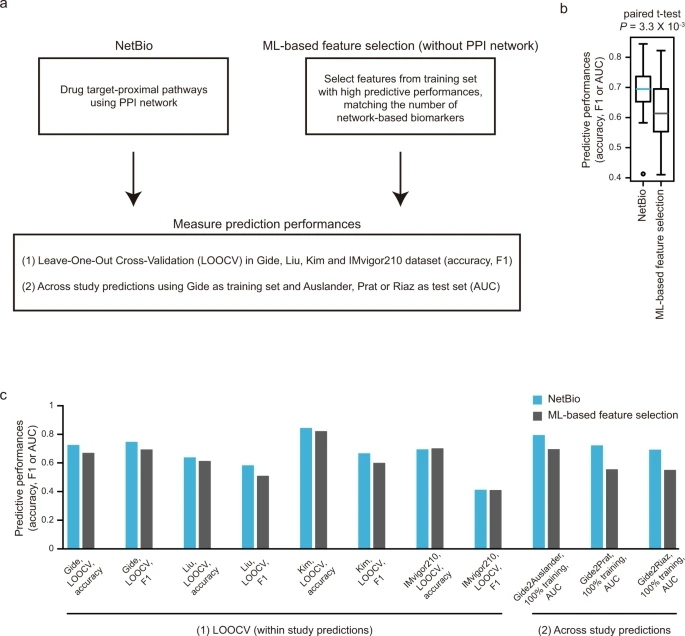
图4. 基于机器学习的特征选择的预测性能比较。图片来自Nat. Commun.
此外,在对黑素瘤队列进行预测时,均观察到了性能改善(跨研究预测;图4c),表明网络引导选择有助于减少ML模型的过拟合。这表明,可以通过利用基于网络的生物标志物选择来识别可靠的转录组生物标志物。
基于NetBio的预测再现了TCGA数据集中的免疫微环境
研究者使用Gide或Liu数据集(黑色素瘤队列)预测TCGA数据集(TCGA SKCM)中黑色素瘤患者的ICI反应,Kim数据集(胃癌队列)预测TCGA胃癌(TCGA STAD), IMvigor210数据集(膀胱癌队列)预测TCGA膀胱癌(TCGA BLCA)患者,并将预测的药物反应与(i)肿瘤突变负担(TMB)或(ii) TCGA患者的免疫环境相关(图5a)。
基于NetBio的预测成功地再现了免疫微环境(图5b)。正如预期的那样,Gide和Liu队列表现出相似的免疫微环境特征,与白细胞比例、CD8 t细胞比例呈高度正相关,与M2巨噬细胞比例呈高度负相关(图5b)。
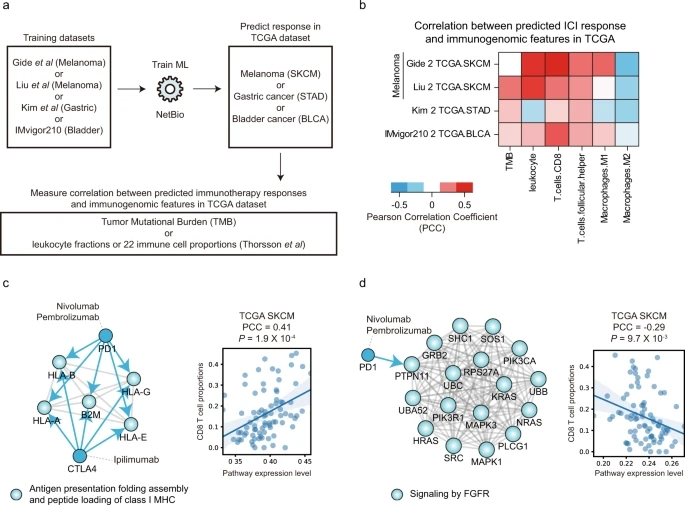
图5. 基于NetBio的预测再现了免疫微环境。图片来自Nat. Commun.
研究者进一步研究了哪些NetBio通路,与免疫细胞比例高度相关。在使用Liu数据集时,在最重要的通路中(前10个特征重要系数为负),FGFR信号通路与CD8 T-细胞比例的相关性最高,该通路的表达水平与细胞比例呈负相关(图5d)。
NetBio通路表达水平与膀胱癌患者的免疫细胞浸润有关
研究者使用了不同的免疫表型,包括(i)免疫沙漠(少于10个CD8 T细胞),(ii)排除(CD8 T细胞邻近肿瘤细胞),和(iii)浸润(CD8 T细胞接触肿瘤细胞)表型,并将趋化和吞噬途径的表达水平与免疫表型进行了比较(图6b, c)。
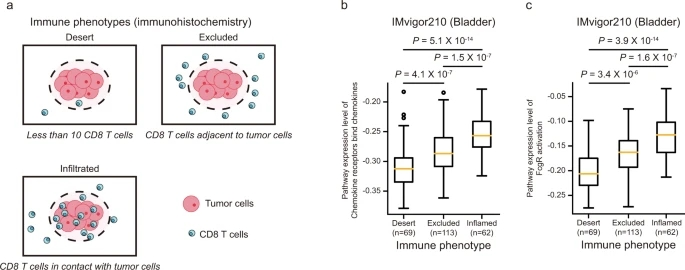
图6. NetBio通路在膀胱癌中的表达水平与基于免疫组化的免疫表型一致。图片来自Nat. Commun.
与免疫荒漠或排除表型相比,免疫浸润表型表现出最高的通路表达水平(图6b, c;Mann-Whitney U P < 0.05),提示NetBio通路可捕获膀胱癌白细胞浸润分数。研究结果表明,NetBio可以持续揭示与免疫治疗反应相关的免疫微环境的通路。
将 NetBio 与 ML 模型中的肿瘤突变负荷 相结合,可以提高对接受 PD-L1 抑制剂治疗的膀胱癌患者的预测
虽然高肿瘤突变负荷(TMB)水平与ICI治疗的获益增加相关,但ICI应答者和无应答者的TMB水平往往有显著重叠,这表明仅TMB不足以预测ICI疗效。因此,研究者测试了将NetBio与基于TMB的预测器相结合是否能提高预测性能(图7a)。
结合NetBio表达水平和TMB改善了atezolizumab治疗膀胱癌患者的总生存预测。atezolizumab是一种PD-L1抑制剂。使用LOOCV预测ICI治疗反应,仅使用TMB训练ML模型,预测有反应组和预测无反应组之间的1年生存率差异为18%。当同时使用TMB和NetBio时,1年生存率差异增加到22.3%。
Raf激活通路在两个亚组间表达显著差异。从PPI网络来看,Raf激活通路的成分包括HRAS、KRAS和JAK2与PD-L1的直接相邻(图7e),这表明该通路可能在药物治疗过程中发挥机制作用。
研究者测试了当(i) PD-L1表达低,模拟PD-L1抑制,以及(ii) TMB水平高时Raf激活是否影响总生存。在PD-L1表达低、TMB水平高的膀胱癌患者中,Raf激活通路对总生存率有统计学显著影响(图7f;P = 0.025)。
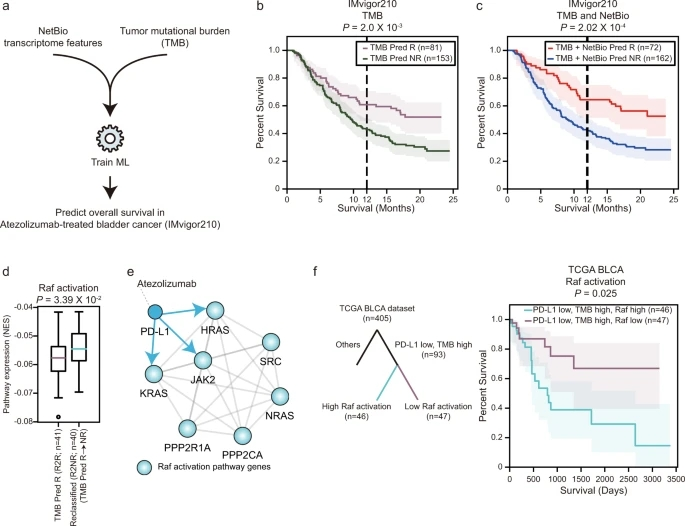
图7. 结合基于网络的转录组特征和TMB提高了atezolizumab治疗膀胱癌患者的总生存期预测。图片来自Nat. Commun.
总结
本文测试了基于网络的生物标志物发现管道是否可以对免疫治疗做出稳健的预测。基于NetBio的ML表现出一致的预测性能,优于基于GeneBio、TME-Bio的预测或纯数据驱动方法。该工作能够为使用ICI治疗的精准医疗提供有趣且新的研究机会。
参考文献
Kong, J., Ha, D., Lee, J. et al. Network-based machine learning approach to predict immunotherapy response in cancer patients. Nat Commun 13, 3703 (2022). https://doi.org/10.1038/s41467-022-31535-6