

背景介绍
AutoDock Vina (Vina)、AutoDock4 (AD4)、AutoDock-GPU、AutoDockFR以及AutoDock-CrankPep,都是AutoDock套件中的对接程序。与套件中其他对接程序相比,Vina是使用最广泛的程序,这可能是因为它易于使用和速度快,而且是开源的。
世界各地的研究小组在Vina源代码的基础上进行了修改和构建,改进了搜索算法(QuickVina2),使界面对用户更加友好,允许用户通过界面修改评分条款(Smina),并对糖类(Vina- carb)和含卤(VinaXB) 化合物改进了打分函数,排序及得分(Vinardo)。
遗憾的是,AutoDock套件中还有一些方法在Vina程序中是不可用的,因为它们是专门通过AD4评分功能或AD4程序实现的。这类方法的例子包括与大环柔性对接、专门的金属配位模型、显式水分子建模、粗颗粒配体模型和配体的不可逆结合。AD4允许用户修改大量的对接参数,可直接访问一些内部引擎部件,这使其非常适合开发新的对接方法。相反,Vina界面是高度专业化的,其特点之一是执行对接时所需的用户输入非常有限。因此,如果不对源代码进行重大修改,就不可能实现额外的功能。
事实上,由于搜索复杂度的不同,AD4比Vina慢100倍。性能上的巨大差异是由于Vina中使用了更好的搜索算法,它是一种蒙特卡罗(MC)迭代搜索,结合了BFGS基于梯度的优化器。与AD4的拉马克遗传算法和Solis-Wets局部搜索相比,Vina的搜索效率更高,用更少的对接函数评价给出更好的对接结果。
AutoDock Vina可以说是用于分子对接的最快和最广泛的开源程序之一。然而,与AutoDock套件中的其他程序相比,它缺乏对特定功能的建模支持,如大环或显式水分子。为此,来自美国斯克里普斯研究中心的Stefano Forli教授等人,在AutoDock Vina 1.2.0中改进实现了上述功能。此外,研究者还实现了Python绑定,以促进脚本编制和对接工作流的开发。相关的研究成果以 “AutoDock Vina 1.2.0: New Docking Methods, Expanded Force Field, and Python Bindings” 为题发布在国际著名期刊Journal of Chemical Information and Modeling上。
1.评分函数扩展和改进
一个主要的改进是Vina中AD4评分函数的可用性。它允许用户使用相同的基于Vina MC的搜索算法访问它,并以同样的效率探索其能量景观(energy landscape),这有助于大规模虚拟筛选。
在性能方面,在Vina 1.2.0中,使用AD4评分函数进行能量评估所需的平均时间,几乎是使用Vina评分函数的3倍。这是由于存在额外的静电势以及需要为每个可移动的原子插入去溶剂化图。
在AutoDock Vina 1.2.0中,研究者添加了对加载外部格点文件的支持,在AD4和Vina评分函数中启用了所有这些方法。
研究者扩展了Vina和AD4评分函数,以支持水合对接方法(原子类型W)和宏观循环取样方法所需的原子和伪原子的新原子类型。此外,研究者还添加了硅(原子类型Si)的参数,以满足用户更好地支持公共存储库(如ZINC数据库)中涵盖的化学空间的要求。
2.新的对接方法
Vina现在可以同时对接多个配体。这种功能可能会在基于片段的药物设计中得到应用,在这种设计中,结合相同靶标的小分子可以生长成具有潜在更好亲和力的更大的化合物。
开发水合对接协议,可用来模拟直接参与配体-受体相互作用的水分子。该方法基于与球形水分子明确水合的配体对接,可用于预测单个水分子的位置和作用(即桥接或置换),并改进配体构型预测。水分子由W型单个原子表示,并被添加到每个氢键矢量末端的配体分子中。在对接过程中,W原子随配体移动,不参与分子内相互作用,并允许与蛋白质重叠。
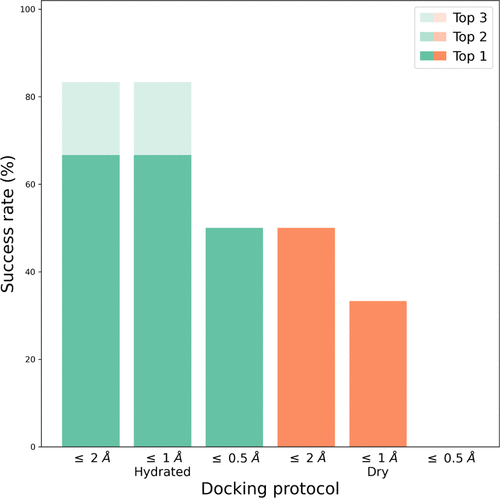
图1. 使用AutoDock4.2评分函数,考虑top 1、top 2、top 3构象,有或没有水合对接协议的6个配体对HSP90的对接成功率。图片来源于J. Chem. Inf. Model.
研究AD4最常用的方法之一是AutoDock4Zn,这是一种用于模拟锌配体的专门力场。该方法在Vina中的实现结果如图2C所示。该方法能够再现AD4原始工作中报道的改进的对接性能,并显示了与配体晶体构型和最佳锌配位几何形状的良好重叠。
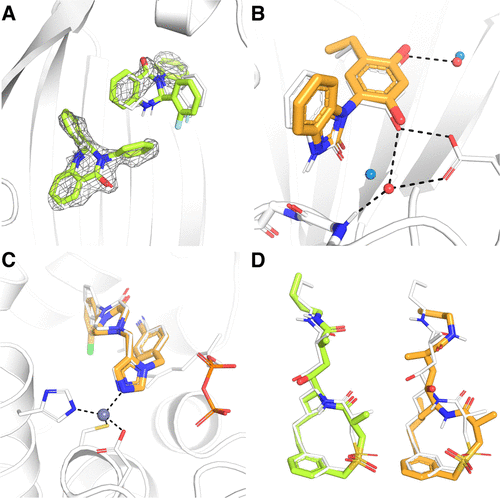
图2. AutoDockVina 1.2.0用于对接(A)多个配体(PDB 5×72);(B)使用AutoDock4的水合对接协议(PDB 4ykq)与水分子;(C)在锌存在下使用AutoDock4Zn力场(PDB 1s63);(D)柔性大环化合物. 图片来源于J. Chem. Inf. Model.
大环对接是一项具有挑战性的任务,因为很难通过建模获得不同构象的扭转变化来对柔性环取样。当前AutoDock Vina 1.2.0中大环采样的实现与AutoDock-GPU中相同,不同于原来的方法,其使用了虚拟原子。由每个先前断裂的键连接的原子,加上一个虚拟原子。每个虚拟原子与其母原子之间的距离对应断裂键的长度,1-3角与原始键的几何形状相匹配。在对接过程中,一个线性势吸引每个虚拟原子与相反的母原子重叠,以适当的距离和1-3个角度恢复断裂的键。
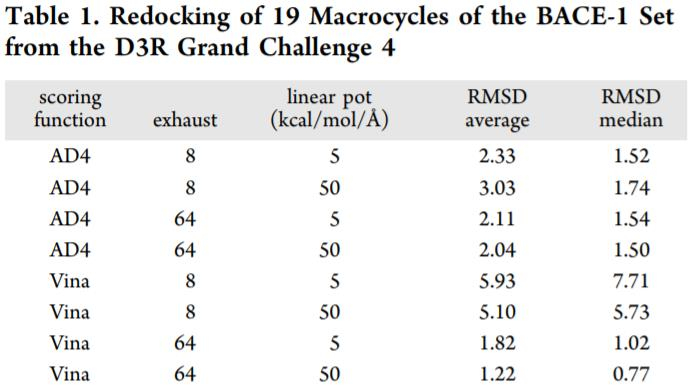
表1. 19个来自D3R Grand Challenge 4的BACE-1组大环的重新对接。表格来源于J. Chem. Inf. Model.
3.Python对接
Python接口提供了以下功能:
① 创建AutoDock Vina引擎的实例(评分函数选择、CPU核和随机种子);
② 读/写一个或多个PDBQT文件;
③ 计算Vina亲和力图;
④ 读/写Vina亲和力图和读AutoDock亲和力图;
⑤ 随机化输入配体的方向和位置(randomize_only);
⑥ 评估当前构象的能量(score_only);
⑦ 执行本地优化(local_only);
⑧ 设置蒙特卡罗全局搜索参数(穷尽性、输出构象数、最大评估值等)。
因此,基本的Vina计算可以配置和执行如下:

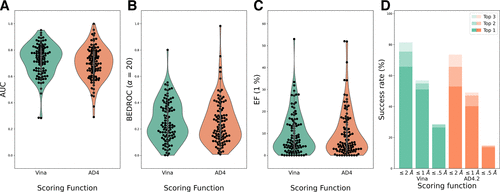
图3. DUD-E数据集活性化合物的早期识别和晶体构象预测。图片来自J. Chem. Inf. Model.
结论总结
本篇文章研究者开发了对接程序的一个新版本AutoDock Vina 1.2.0 ,它支持AutoDock 4.2评分功能、批量对接,同时实现了Python绑定。这项工作实现了AutoDock4和AutoDock Vina程序功能的统一。源代码地址: https://github.com/ccsb-scripps/AutoDock-Vina。
参考文献
Jerome Eberhardt, Diogo Santos-Martins, Andreas F. Tillack, and Stefano Forli, AutoDock Vina 1.2.0: New Docking Methods, Expanded Force Field, and Python Bindings, J. Chem. Inf. Model., Article ASAP. DOI: 10.1021/acs.jcim.1c00203