
引言
在药物发现的早期阶段,如何对没有成药前途的分子进行尽早排除以避免不必要的生物学和临床试验至关重要,类药性则是其中一个关键概念。关于类药性的研究已持续了几十年,最早可追溯到上世纪80年代的Lipinski“类药五规则”(Ro5)。但随着药物研究的进一步发展,Ro5这类启发性的类药性判别方法受到越来越多的质疑。此后在2012年Bickerton提出的QED(类药性的定量估计)方法则慢慢替代了Ro5,尤其是在机器学习领域,如分子生成模型。本研究中,来自波兰科学院的Bartosz A. Grzybowski搭建了几种不同的深度学习模型(多层感知机、图卷积网络、自编码器)来进行了类药性预测,同时也对比了多种非药数据集(NOC、PDB及ZINC)和多种分子表示(RDKit、Mold2、MCS、ECFP4、Mol2vec)对预测准确率的影响。作者也使用这些模型的贝叶斯神经网络对预测结果的确定性进行分析,并整合多个模型进行预测使预测准确率达到了93%。此外,作者通过对模型输出的随机性误差和知识性误差进行分析得出结论:93%的类药性预测准确率很可能是当前数据集的极限。
数据集准备
药物数据集由2,136个FDA批准的小分子药物组成(此后简称为DRUGS)。DRUGS来自DrugBank的 2,635条相关数据,作者从中过滤掉了(1)无机化合物和重金属配合物,(2)平衡离子(包括富马酸盐和柠檬酸盐)和(3)10个包含128个以上原子的分子(由于图卷积网络(GCNN)无法计算过大的分子),剩余分子被中性化并转化为Canonical SMILES。
作者在研究中使用了3个非药数据集:(1)ZINC15,约1.9千万个有机化合物;(2)Network of Organic Chemistry(NOC),包括来自Reaxys和专利的约6百万个分子;(3)来自PDB的约1.3万个配体。3个非药数据集均经过与DRUGS类似的清洗与预处理及去重,并删去与DRUGS中的药物相似的分子(ECFP4的Tanimoto相似性大于0.85)。
作者选择了5种分子表示进行研究:(1)RDKit中的200个描述符;(2)777个Mold2描述符;(3)2,048位ECFP4指纹;(4)一个二进制向量,其中每维表示在药物和非药物数据集中最频繁出现的3,000个最大通用子结构(MCS)之一的存在与否;(5)分子图的张量表示,每个分子使用两个矩阵表示:原子特征矩阵和一个邻接矩阵。对RDKit和Mold2两种分子表示,在模型训练前在训练数据集上进行了标准化,包括删除方差为零(即对于所有化合物为零或完全相同)的描述符及减去均值并除以标准差。对分子图的张量表示,其原子特征矩阵包含了原子杂化和与每个重原子相连的氢原子数这两个特征。
由于非药数据集比DRUGS的分子数目大几个数量级,因此阴性数据集必须加以平衡。除非另有说明,否则作者将对较大的(非药物)集合进行随机下采样;也就是说,作者随机抽取了与DRUGS大小相同的非药样本,并对多个(通常是三个)样本计算平均准确率。在模型选择期间,将包含药物和相等数量的非药物的每个平衡数据集按9:1的比例分为训练集和测试集,并将较大的部分用于五重交叉验证。测试集用于测试在整个训练集上训练的分类器(即用于交叉验证的分类器)的效果。
结果与讨论
评价QED预测效果
类药性的预测之所以必须使用多参数模型是因为类药与非类药分子无法简单的通过任何单个描述符区分(图1)。作者首先测试了目前广泛使用的多参数模型QED对药物分子与非药分子的区分能力,并以QED作为本篇研究的基线模型。如表1所示,QED准确率在42-57%的范围内,因阴性数据集而异。作者自己尝试复现当初发表QED模型的论文中的结果,得到的准确率为65%左右。这些相对普通的准确率数值体现了不同数据集中QED分布之间的显着重叠(图2)。
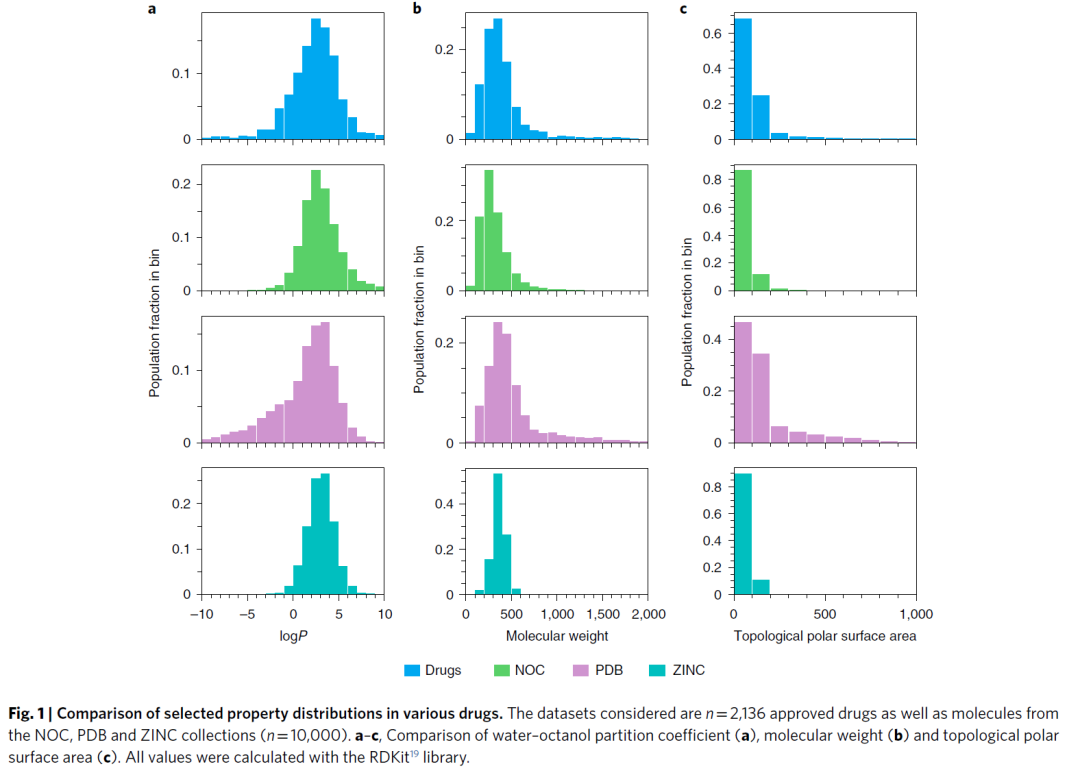
图1 药物分子与非药分子间常见理化性质的分布比较
图片来源:Nature Machine Intelligence
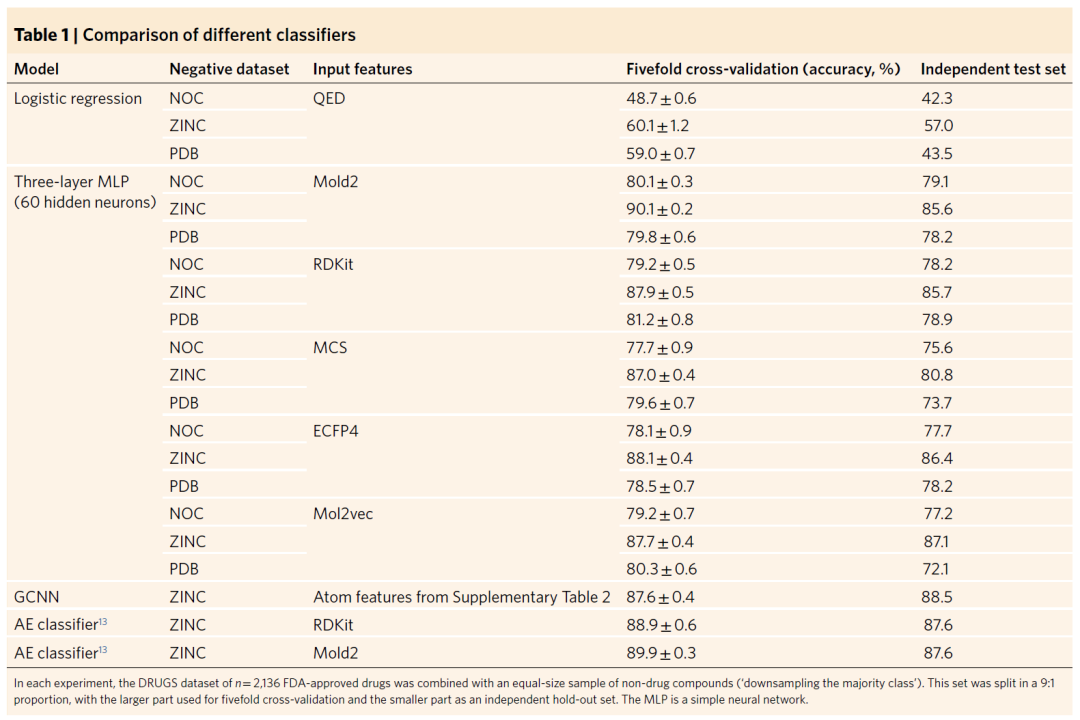
表1 不同类药性预测模型的预测效果对比
数据来源:Nature Machine Intelligence
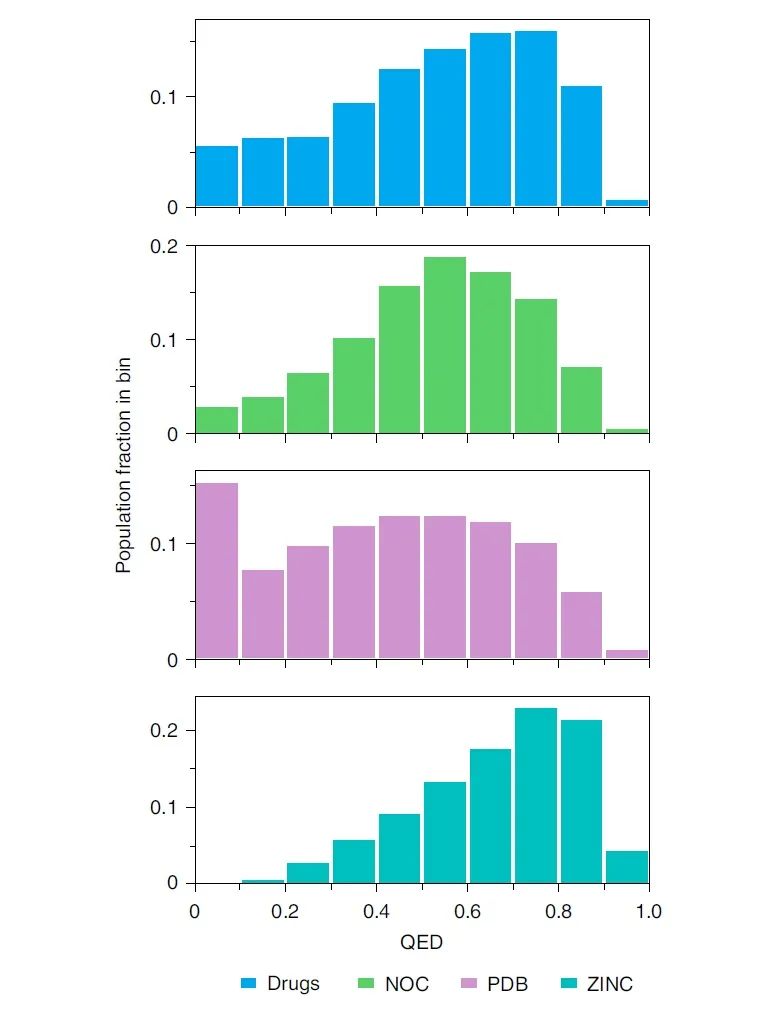
图2 药物与非药分子的QED分布对比
数据来源:Nature Machine Intelligence
非药数据集的重要性
作者研究了RDKit、Mold2、MCS、ECFP4和Mol2vec共5种分子描述符及ZINC、NOC和PDB共3个非药数据集的不同组合对具有60个隐藏单元的MLP类药性预测模型预测效果的影响。从表1中的数据可以明显看出:(1)对于给定的一组非药数据集,准确率仅在很小程度上取决于所使用的描述符的选择;(2)使用不同的非药数据集明显影响准确率:如果使用ZINC则准确率约为81-87%,而如果使用NOC或PDB则准确率约为72-79%。那么问题来了,这些非药数据集中存在的分子在多大程度上不同于真正的药物?哪一组最不像药?
为了回答这个问题,作者采用标准的二元分类法来迭代地优化用于构建分类器的可靠阴性(RN,“最可能的非药物”)的集合。首先,将非药数据集的所有分子均视为真正的非药物来构建分类器,即使用整个非药数据集与DRUGS来训练模型;然后使用该分类器对非药数据集的分子进行分类,被分为非药的分子组成RN;继而使用RN与DRUGS进行模型训练,并对不在RN的非药分子再次进行分类,被分为非药的分子则被纳入扩充RN;迭代以上这些步骤直到不再有新的分子需要被纳入RN。另外,作者也尝试了另一种类似的方法,即首先将整个非药数据集视为RN,然后迭代缩小RN直到RN不再缩小。最终,不在RN种的非药分子数目越少,则认为此非药数据集“最不像药”。使用RDKit描述符的MLP模型作为分类模型进行上述实验后发现“最不像药”的非药数据集是ZINC(表2),这也能一定程度地解释表1中的准确率差异。作者建议将ZINC用作非类药分子的阴性组,并在本文的后续研究中只使用ZINC。
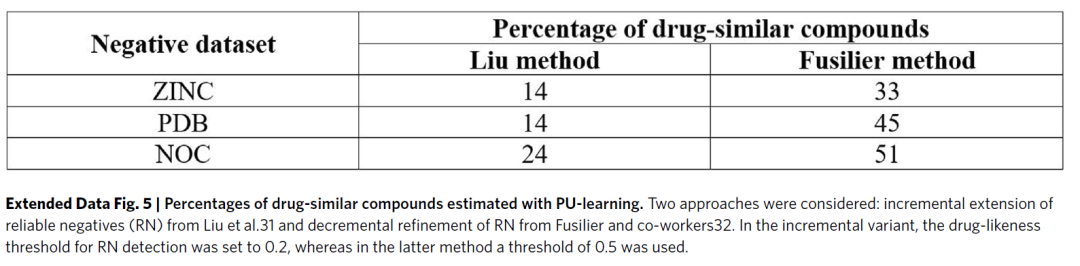
表2 非药数据集中的类药分子数目
数据来源:Nature Machine Intelligence
训练更复杂的类药性预测模型
作者还进一步研究了更复杂的深度学习模型在类药性预测中的效果,共包括(图3):(1)基于自编码器(AE)的分类器;(2)分子图卷积网络(GCNN);(3)AE和GCNN各自的贝叶斯神经网络(BNN)版。由于使用了ZINC作为阴性数据集,AE和GCNN仅比MLP略有改善,达到了约88%的准确率(表1)。
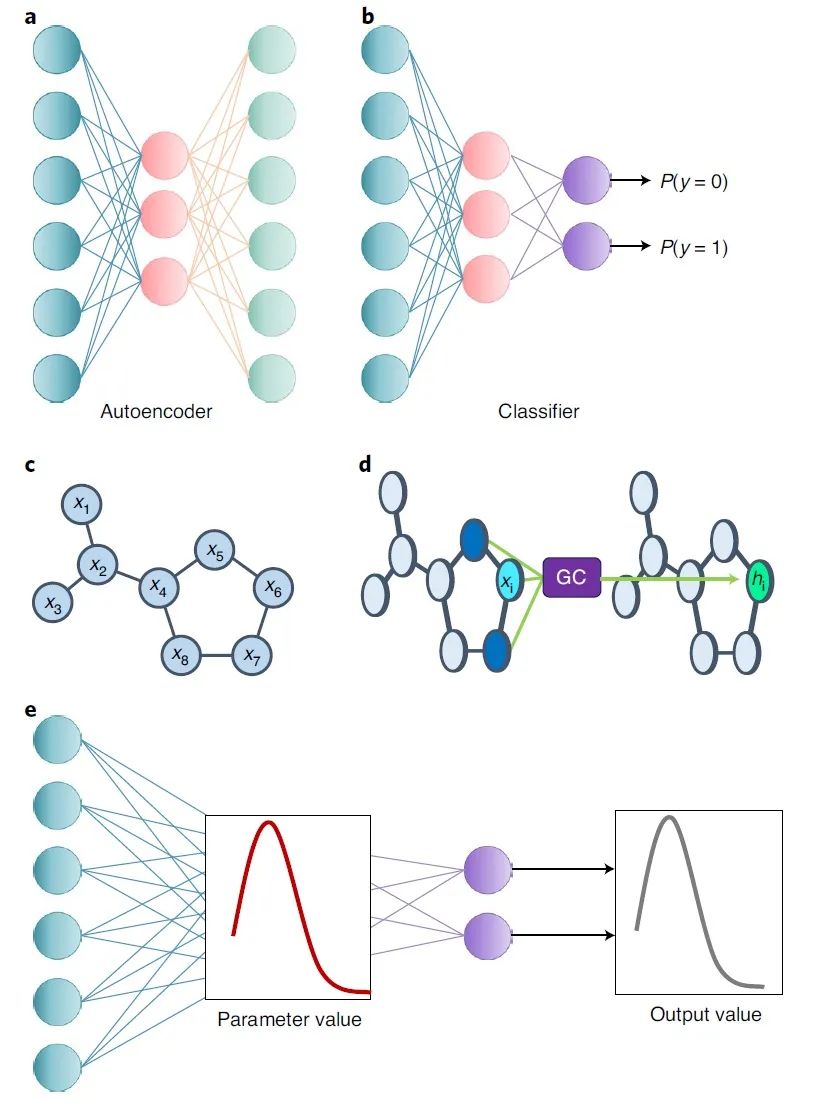
图3 文中涉及的神经网络架构的图示
数据来源:Nature Machine Intelligence
但是,当使用BNN来比较两者间预测的不确定性时,这些模型之间存在显著差异。简而言之,BNN在预测的同时可以进行误差估计。总误差(预测方差)可以分解为两个部分:随机性的(不可减少的,数据固有的)和知识性的(从模型本身产生并随数据量的增加而减少)。图4表明,同一测试集上的AE和GCNN在预测的类药性数值和预测方差方面具有差异。从虚线定义的四个象限中,非对角线区域(左上角和右下角)最为有趣,因为在这些位置,两个分类器的预测不一致。例如,考虑用AE正确分类而用GCNN错误分类的药物(右下角),其中一些药物的GCNN的预测方差较高,而AE的预测方差较低,这时可以通过选择不确定性较低的预测结果而实现更准确的分类。
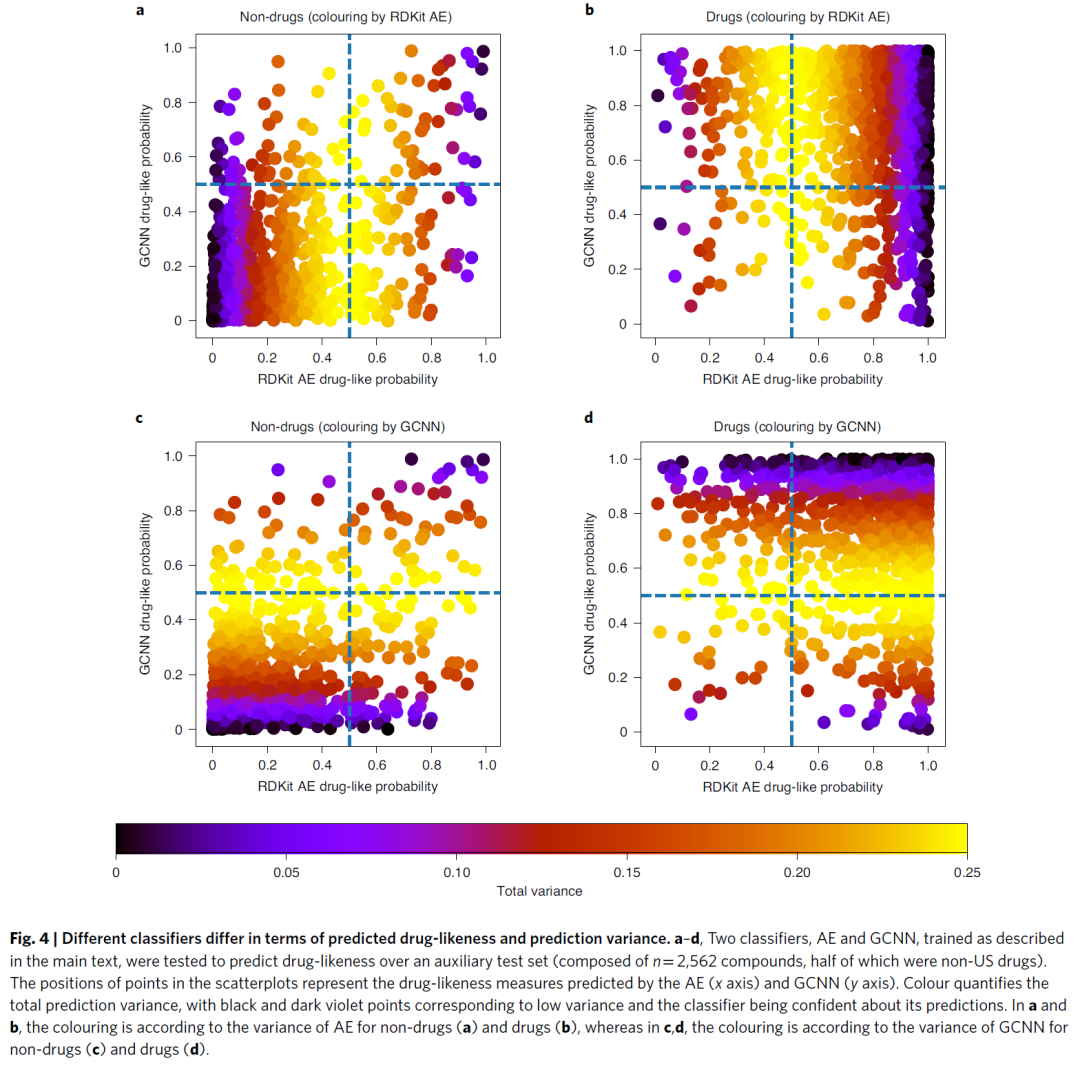
图4 不同模型的类药性预测结果与预测方差
数据来源:Nature Machine Intelligence
因此,作者通过组合不同的分类器并保留不确定性较小的预测来提高整体准确率。作者研究了图5所示的三个模型及其组合的效果并在由1,281种世界药物(FDA未批准)组成的不相交的药物集和ZINC上进行了评价。单个模型的准确率为约88%,但组合模型则可以达到约93%(GCNN + AE为93.0%,GCNN + Mol2vec为92.6%,AE + Mol2vec为92.9%)。此外,组合模型也可以得到更好的灵敏度-精确度曲线(图5)与ROC曲线(图6)。
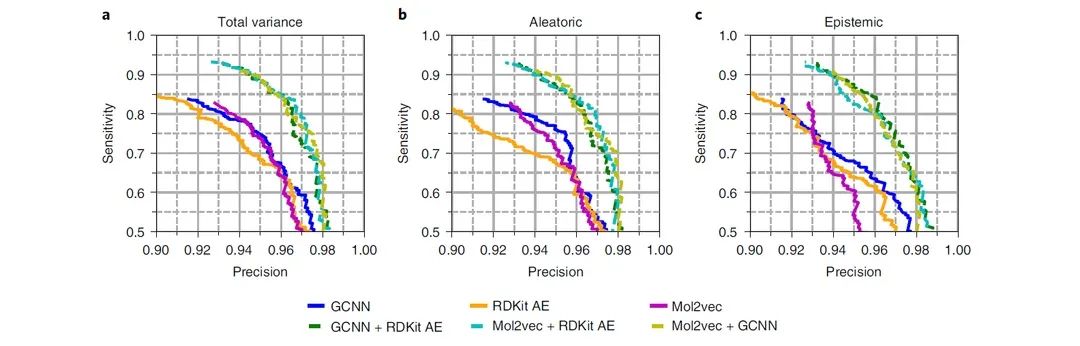
图5不同模型的灵敏度-精确度曲线
数据来源:Nature Machine Intelligence
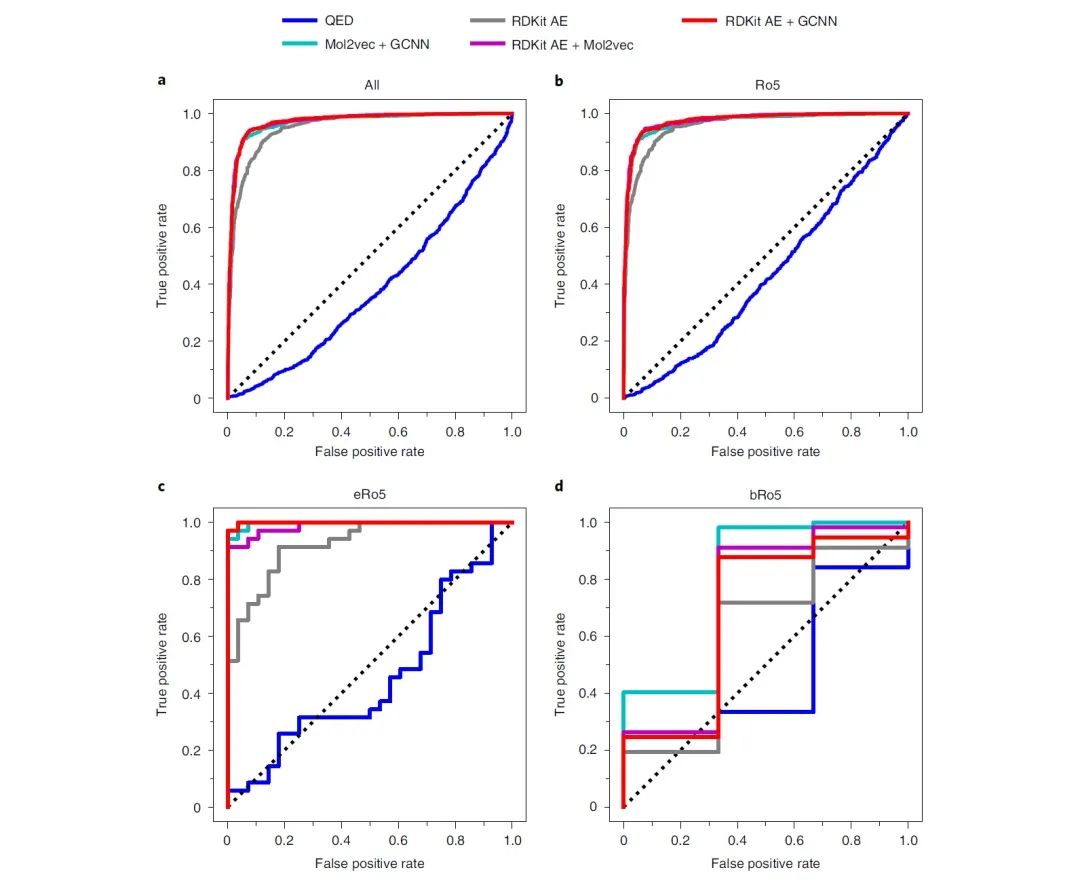
图6 不同模型的ROC曲线
数据来源:Nature Machine Intelligence
作者还观察到,总方差在很大程度上由随机性误差决定,而知识性误差对方差的贡献仅在约7%-15%之间。如对于RDKit AE + GCNN和GCNN模型,随机性的(“不可减少的”)方差的占比高达90–93%,这表明进一步增加数据只能略微地提高预测的确定性(减少误差),而模型存在的误差绝大多数是来自训练数据本身固有的特征(类似于图像处理中照片的模糊部分)。
局限性
尽管组合分类器具有令人满意的灵敏度-精密度特征,但需要强调的是,该方法或任何其他AI方法都几乎不能摆脱在化学上无意义甚至可笑的预测结果。例如,AE / GCNN分类器错误地将常见的烃(例如苯、环己烷或甲苯)识别为类药分子。此类预测产生的原因是现有数据集所固有的——具体来说,尽管碳氢化合物的存在固然丰富,但它们仅占ZINC等数据库中独特分子结构的0.03%。当针对仅约2,000种FDA批准的药物训练分类器时,平衡后的阴性组样本平均仅包含约1个碳氢化合物,因此不太可能学到这种知识。当然,可以通过扩大数据集来改善这种情况,但FDA每年新批准的药物数量非常有限。另一种解决方法则是通过纳入其他模型来避免问题。作者尝试通过构建预测毒性的分类器来实现此目的,希望这些分子被预测为有毒的,从而排除出类药范围。奇怪的是,作者得到了更荒谬的结果:这些分子比通常的药物分子更类药。作者认为这与所使用的毒性数据集ClinTox与Tox21中收录的分子通常与药物分子结构类似有关,这也暴露了另一种广泛使用的数据集中可能存在的数据偏差。基于这些结果,作者认为目前唯一可行的方法似乎是在组合模型中纳入启发式的算法筛除已知的非类药分子,虽然这种方法非常不优雅。作者尝试了删除(1)拓扑极性表面积和形式电荷等于零的化合物(如烃),(2)包含少于三个重原子的化合物和(3)含有化学毒药中常见结构模式的化合物。总的准确率虽然不会发生明显的变化,但是可以消除明显的令人反感的分子。
作者同样尝试使用其模型去区分临床失败的候选药物与上市药物,结果不出意外是几乎无法区分。假设这些失败的候选药物已由专业药物化学家进行了全面评估,因而没有任何明显的非类药特征,那么这些结果则提醒我们,药物的最终临床成功与否不仅可以反映其化学结构的特征,还可以反映出经济和知识产权因素,或者是竞争对手的类似产品疗效更优异。
代码
Computer codes underlying this work are made freely available for non-commercial uses under a Creative Commons Attribution Non Commercial-No Derivatives 4.0 International (CC BY-NC-ND 4.0) license and are deposited at https://zenodo.org/record/3776450.
参考文献
Beker, W., Wołos, A., Szymkuć, S. et al. Minimal-uncertainty prediction of general drug-likeness based on Bayesian neural networks. Nat Mach Intell 2, 457–465 (2020). https://doi.org/10.1038/s42256-020-0209-y. Published Date: 12 August 2020