

背景介绍
设计变构调节剂,需要先了解蛋白质的哪些部分可以与配体实现高亲和力结合。然而,这是一项艰巨的任务,尤其在结合位点处于“隐藏”的情况下,将会变得更加困难。所谓“隐藏”位点(以下简称隐位点),指的是一些有配体与之结合时明显,但没有配体时难以检测的位点。因此,当人们在蛋白质上找到一个隐位点时,可能并不能保证该蛋白质是可药的。此类药物设计领域关注的隐位点,它也必须是变构的:结合位点的构象必须影响蛋白质的功能。尽管如此,发现隐藏口袋的能力,还是为药物设计开辟了另一种有前景的途径。
当前,为了研究结合位点(包括隐型和非隐型),人们已经开发了各种算法,如:
1.基于分析分子动力学(MD)模拟,如EPOCK、POVME和TRAPP等。尽管这些算法对于表征已知袋的行为是有用的,但是它们对结合袋位置先验知识的依赖,制约了它们在未被充分研究的蛋白质上的应用。
2.以静态结构(例如PDB文件)作为输入,如LIGSITE、KVFinder、COACH、COFACTOR、ConCavity、FPocket、galaxsite、ghecom、DoGSite和FTMAP等,虽然使用单一的结构可以减少计算时间,但很难找到存在于所有构象中的隐蔽位点。虽然这些程序非常强大,但它们需要专门的模拟,这就阻止了它们在预先计算的轨迹上的使用。
3.EPOSBP, trj_cavity, MDPocket, D3Pockets和NanoShaper都是基于几何的口袋发现器,但它们忽略了物理化学性质,而且都有自身的局限性。
4.CryptoSite采用了一种不同的方法,它在算法中不使用探针计算自己的MD轨迹。然而,该程序要求用户定义感兴趣的区域,限制了对缺乏任何关于隐位点位置信息蛋白质的适用性。
当前的每一种策略都有自己的局限性,缺乏通用性。而机器学习(ML)在复杂数据中发现趋势的能力,使其成为预测小分子结合位点这一重要任务的一种有前景的技术。机器学习具有显著改善结合位点预测的潜力,包括预测晶体结构中不存在的隐袋。因此,将ML和其他尖端计算技术应用于结合位点的预测,将对药物设计过程产生重大影响。
主要内容
基于结构的药物发现工作,需要了解药物结合位点在靶蛋白上的位置。为了解决寻找可用药位点的挑战,来自美国约翰·霍普金斯大学的Albert Y. Lau等研究者,开发了一种名为TACTICS的机器学习算法(基于轨迹的构象分析,以识别隐蔽位点),它使用一个分子结构集合(如分子动力学模拟数据)作为输入。首先,TACTICS使用k-均值聚类选择少量构象,这些构象代表数据的整体构象异质性。然后,基于蛋白质运动和几何学,TACTICS使用一个随机森林模型来识别每个选定构象中潜在的可结合残基。最后,使用片段对接对潜在结合口袋中的残基进行评分。相关的研究成果以 “Finding Druggable Sites in Proteins Using TACTICS”为题发布在国际著名期刊Journal of Chemical Information and Modeling上。
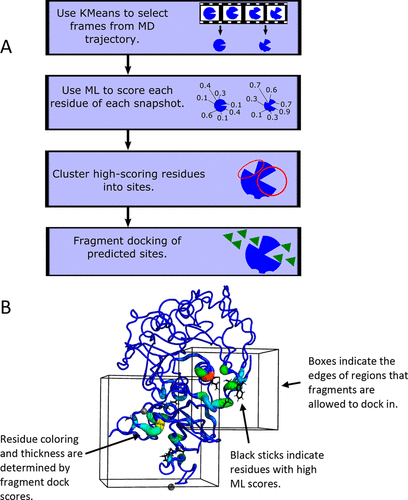
图 1. TACTICS的设计。图片来源于J. Chem. Inf. Model
为了演示TACTICS的能力,研究者将该算法应用于三个蛋白质中,分别是SARS-CoV-2蛋白质(主要蛋白酶和甲基转移酶),以及芳基载体蛋白。结果如下:
1. SARS-CoV-2主要蛋白酶
SARS-CoV-2主要蛋白酶(M-pro;nsp5),负责切割病毒RNA基因组翻译产生的多蛋白。由于该蛋白的配体结合特性已经得到了较为充分的研究,M-Pro是检验TACTICS重新发现已知结合位点能力的一个很好的选择。
TACTICS识别出主要蛋白酶活性位点。在模拟的各个点上,TACTICS识别出残基,包括H41、F140和H163。结合位点,如图2A和B等所示。在图中结晶配体的左侧,TACTICS识别出其他残基为可用药残基。因为TACTICS的设计重点是,寻找隐蔽的位点并尽量减少误报,所以它使用ml_std_thresh过滤掉分数不变的残差。
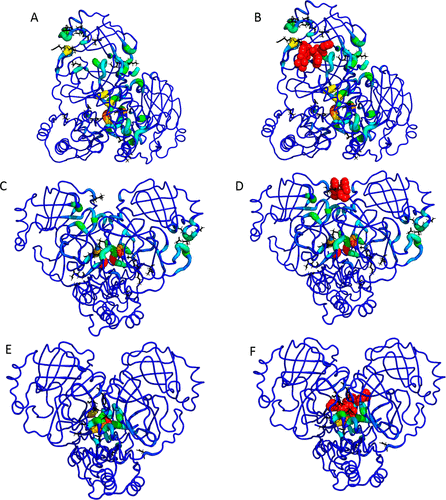
图2. 从TACTICS的输出中为SARS-CoV-2主要蛋白酶选择的图像。图片来自J. Chem. Inf. Model
TACTICS还在主要蛋白酶上,确定了一个已知的变构位点。一个配体在PDB ID 5RGJ的这个位点结晶;虽然晶体结构有一个配体结合到每一个二聚体的前聚体,这两个配体彼此是非常接近的。这种配体已被证明,可以降低蛋白酶的活性。结合位点,如图2C和D等所示。在所示的构象中,口袋比容纳来自5RGJ的配体所需的要大。因此,更大的配体,可能会结合到这个位点上。
二聚体界面,是TACTICS识别的另一个潜在药物靶点。PDB ID 5RFA有两分子的化合物x1187,结合在二聚体界面上。每个分子都与两个原聚物相互作用。实验结果表明,x1187抑制了二聚体和蛋白酶活性,证实了二聚体界面的重要性。
2. SARS-CoV-2 2′-O RNA甲基转移酶
在COVID-19大流行期间,有希望的药物靶点之一是SARS-CoV-2 2 ‘ -O RNA甲基转移酶(MTase)。该酶负责RNA帽的形成,这是病毒RNA入侵的关键。在这里,研究者使用REST2对SARS-CoV-2 MTase的构象空间进行有效采样,利用TACTICS识别已知和隐型结合位点。
TACTICS在识别距离nsp10最远的结合位点和催化位点方面最为成功。除了识别已知的与RNA结合的残基外,TACTICS还识别了RNA结合袋中包含其他残基的构象。在某些构象中,在螺旋α9和α10之间的环中的某些残基(残基235-241)包含在口袋中;关键残基包括N235、P236和I237。L27和S202对于控制扩展口袋的访问尤为重要,图3C和D等显示了这个扩展口袋。

图3. TACTICS定位MTase RNA结合位点,并预测与之相连的口袋。图片来自J. Chem. Inf. Model
在另一种构象中,RNA结合袋包括L27、Y222和H225;这种构象如图3E和F等所示。Folding@Home识别了TACTICS预测位点附近的残基;Folding@Home鉴定的残基,包括R19-D26和K137 F152。可以想象,结合这些残基的配体,可以部分填充RNA位点;配体诱导的邻近区域的构象变化,也可能影响RNA位点的结合能力。
TACTICS识别SAM结合位点,如图4A和B所示。TACTICS识别了残基D99和D130,它们都与竞争配体Sinefungin形成氢键。TACTICS还发现了一些附近的残基,包括G71、M131和Y132等。应当注意的是,TACTICS在相对较少的帧中,发现SAM结合残基。这可能表明了TACTICS在寻找特定类型口袋方面的能力局限性。
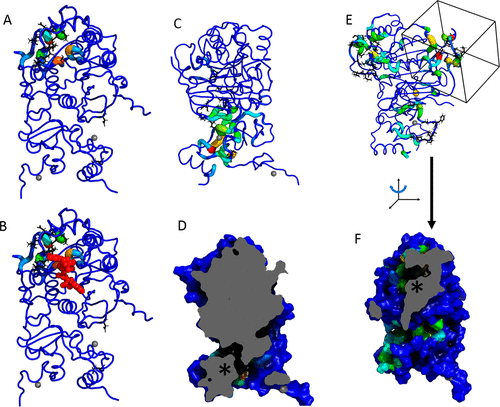
图4. TACTICS在MTase中发现了其他已知和预测的结合位点。图片来自J. Chem. Inf. Model
TACTICS预测nsp10在nsp10/16接口上的结合位点,位点如图4C和D等所示。nsp10上的潜在重要残基,包括S72、C74、Y76、T111、L92,而nsp16上的潜在重要残基,包括V78、S105和D106。
除了在二聚体界面上的位点,TACTICS还识别了另一个可能通过变构抑制破坏活性的位点。新的预测结合位点如图4E和F等所示。它位于RNA结合位点的另一边,潜在的重要残基,包括K123、L163、Q266、R283和E284。
3. 芳基载体蛋白(ArCP)
ArCP没有活性位点,也没有已知的小分子结合位点(除了PP臂)。虽然它有已知的蛋白质结合位点,但蛋白质-蛋白质结合位点与配体结合位点有很大的不同。
在apo-ArCP中,TACTICS预测了两个结合位点。一个预测位点涉及螺旋α1和α2,如图5A和B等所示。α1上的潜在可结合残基,包括Q23、R27和E31,而α2上的关键残基,包括R54、R57和W61。
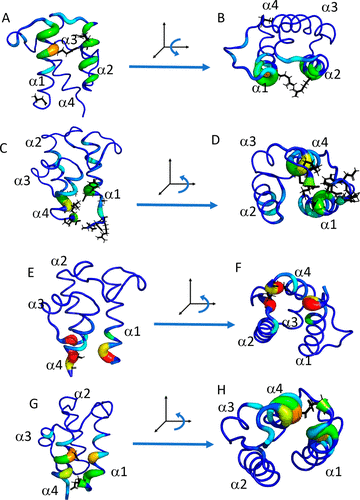
图5. TACTICS输出显示apo-ArCP的预测结合位点。图片来自J. Chem. Inf. Model
另一个预测位点位于N和C端附近。位点非常灵活,观察到各种构象。在一个构象中(图5C和D等),该位点包括α1残基H17、D20和Y21,以及α4残基M87、L88和E93。在另一个构象中(图5E和F等),该位点包括α1残基R16、H17和D20, α4残基S91和P92,以及loop2(在α2和α3之间)残基Y67。在第三种构象中(图5G、H和S12),该位点包括α1残基D14、H17、A18和Y21,以及α4残基N84、Q85和L88。
在holo-ArCP中,TACTICS预测了N和C端形成的结合位点,该位点如图6A和B等所示。该位点与“Apo 2”位点的位置相同;apo和holo结果的对接得分和构象灵活性非常相似。TACTICS还预测了一个主要位于loop1 (α1和α2之间)上的结合位点,它包括残基L39、H40、S43和A48。
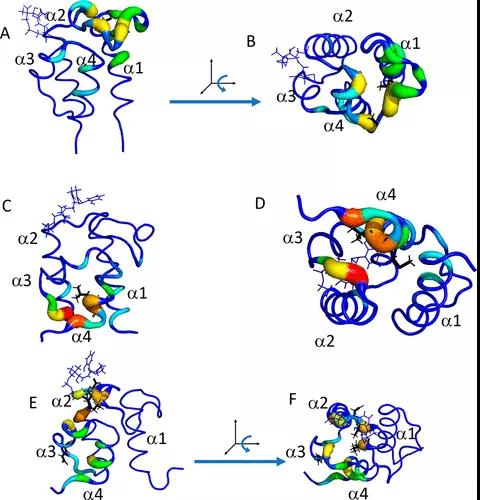
图6. TACTICS对holo-和loaded-Arcp中结合位点的预测。图片来自J. Chem. Inf. Model
在loaded-Arcp中,TACTICS预测了包含螺旋α4和loop2(在α2和α3之间)的结合位点,该位点如图6C和D等所示。螺旋α4上潜在的重要残基,包括L86, M87和R90。在loop2上可能重要的残基,包括Y67和R68。TACTICS还预测了loaded-Arcp的另一个结合位点,该位点如图6E和F等所示。预测位点,包括loop1(在α1和α2之间)、α2和α3中的残基。在loop1和α2上可能重要的残基,包括I46、L50、s52-pp-salicylate arm和L55。Y75是α3上潜在的重要残基。
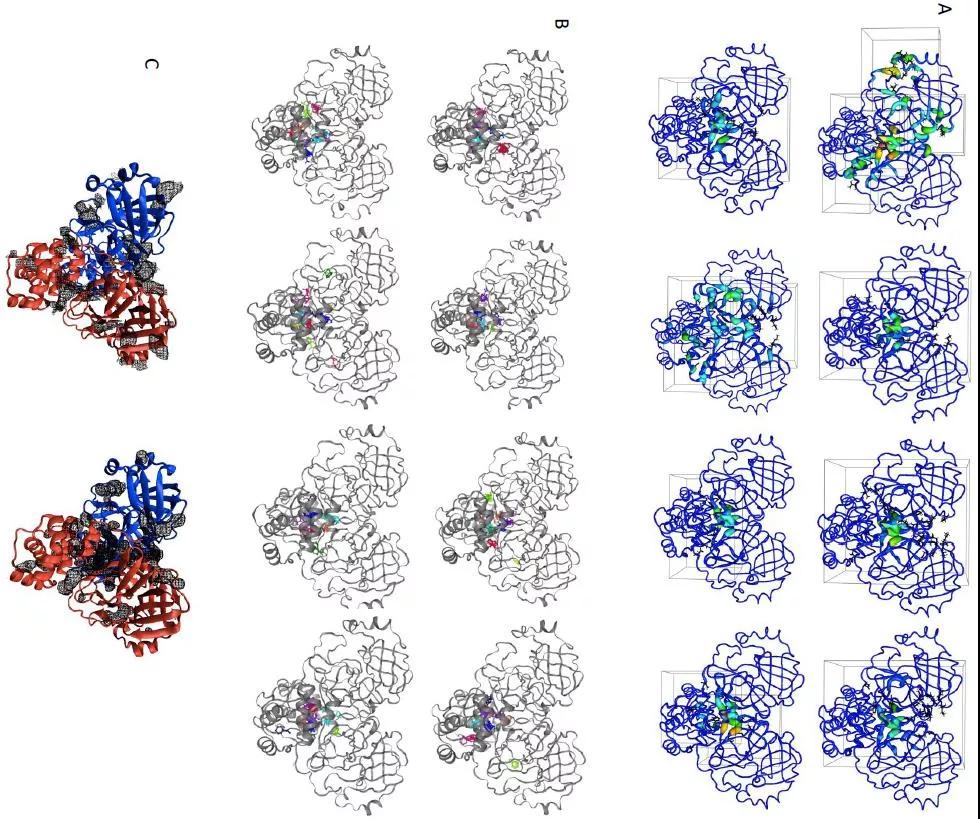
图7. SARS-CoV-2 M-Pro前20 ns口袋预测的比较。图片来自J. Chem. Inf. Model
结论总结
本文章介绍了TACTICS算法,通过分析MD模拟,来寻找蛋白质中的药物位点。TACTICS首先将轨迹聚集起来,选择几个帧进行分析。然后,对每个选定的帧应用随机森林ML模型。该模型取一个蛋白质构象,并在该构象中对每个残基的成药性进行评分。该模型在晶体结构数据库上进行训练;为了捕获构象多样性,每个蛋白质包含多个结构。由ML算法识别的位点经过片段对接以进一步表征其可结合性。
目前,TACTICS是唯一一个既可使用专门为MD轨迹设计的ML模型,又可接受已生成的MD数据的免费程序。TACTICS公开访问地址:https://github.com/Albert-Lau-Lab/tactics_protein_analysis。
参考文献
Daniel J. Evans, Remy A. Yovanno. et al. Finding Druggable Sites in Proteins Using TACTICS. Journal of Chemical Information and Modeling 2021 61 (6), 2897-2910. DOI: 10.1021/acs.jcim.1c00204