
deepHPI基于深度学习,为宿主-病原体蛋白相互作用(HPPIs)预测问题提供了更稳健和准确的解决方案。
背景介绍
病原体的爆发,持续威胁着世界各地的公众健康。基因组学、大数据和计算生物学的进展,正在增强人们对病原体及其相关毒性的系统生物学的理解。这类科学正成为开发精准医疗等平台的关键。宿主和病原体是涉及传染病的生命实体。因此,确定这些蛋白-蛋白相互作用(PPIs)有助于揭示疾病途径和提高耐药性的方法,并最终加速药物和其他治疗方法的开发。
PPIs是由蛋白质的序列决定的,此前人们提出的大多数方法,都基于已知的PPIs,将这些知识外推到新的接口中。这类方法往往以失败告终,因为它们忽略了蛋白质中对识别PPIs很重要的短而稀疏的特征。
用机器学习(ML)预测HPIs的一个重要问题,是如何解决训练和测试都无法获得非交互数据的这个缺陷。目前,人们还没有开发出用于物种间PPI预测的类似资源,这促使科学家们提出了经电脑模拟的方法来生成负反馈的HPIs数据集。
主要内容
美国犹他州立大学的Rakesh Kaundal等人,为了给用户提供一个基于ML框架的更稳健的HPI预测服务,开发了deepHPI。这是第一个使用卷积神经网络(CNN)模型进行HPI预测的服务器。此web服务部署在高性能计算集群中,使用户能够在单个请求中提交大量序列。它对生成的宿主病原体网络进行了丰富的可视化,并通过各种蛋白质注释资源的外部链接进行了增强。deepHPI提供四种宿主-病原体模型类型:植物-病原体、人-细菌、人-病毒和动物-病原体,利用其可操作性进行了广泛的分析和案例研究。
在线访问网址
http://bioinfo.usu.edu/deepHPI/
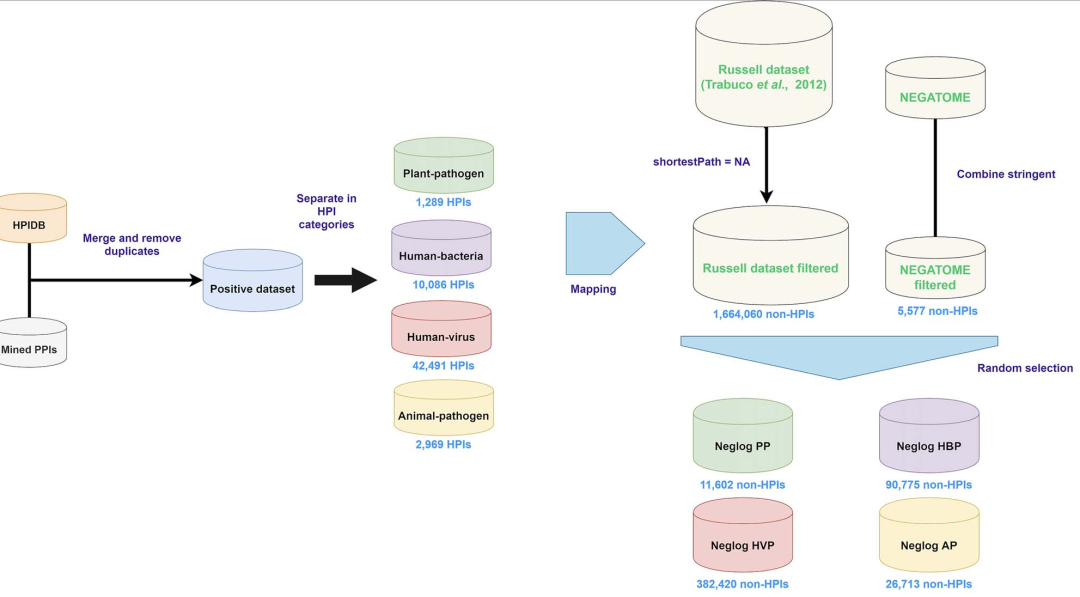
图1. 数据集集合图。图片来自BIB
真实PPIs的收集和数据预处理
图1展示的工作流程图是不同数据集集合的概述。此外,研究者从文献中提取经过实验验证的PPIs,并将其划分到不同的数据集,增加了植物-病原体和动物-病原体数据集的真阳性PPIs数量,也增加了人-病毒和人-细菌数据集的PPIs数量。将HPIDB与手动采集的PPIs合并,删除重复项。通过删除至少一个蛋白质序列短于30个氨基酸的相互作用,超过5000个氨基酸或具有非传统的氨基酸编码(例如X和/或U)。
预处理后共收集到15811个宿主与6621个病原体蛋白之间的56834个相互作用。然后将数据分为四类:植物病原体、人类细菌、人类病毒和动物病原体。人类病毒收集到11011个宿主蛋白和2930个病原体蛋白的42491个PPIs,人类细菌收集到4353个宿主蛋白和2863个病原体蛋白的10086个相互作用;在动物病原体中获得了2969个宿主蛋白和657个病原体蛋白的相互作用,在植物病原体中获得了573个宿主蛋白和382个病原体蛋白的1289个相互作用。
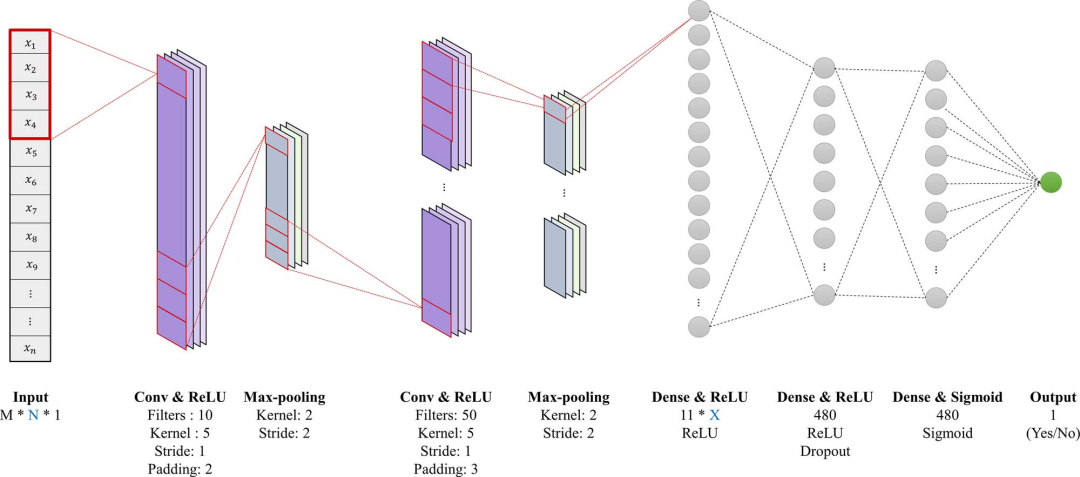
图2. “正向”合成网络的生成与性质。图片来自BIB
CNN架构
在这项研究中,研究者使用PyTorch来实现CNN架构,之后该架构用于HPI数据集建模。为每个数据集类型和每个特征描述符优化网络参数,不便于管理。因此,研究者选择使用最小的植物-病原体数据集来识别网络参数,然后将这些参数应用到其他数据集。
图2中定义的CNN架构实现了最高的性能,它是为其他数据集类型(人类病毒、人类细菌和动物病原体)实现的。
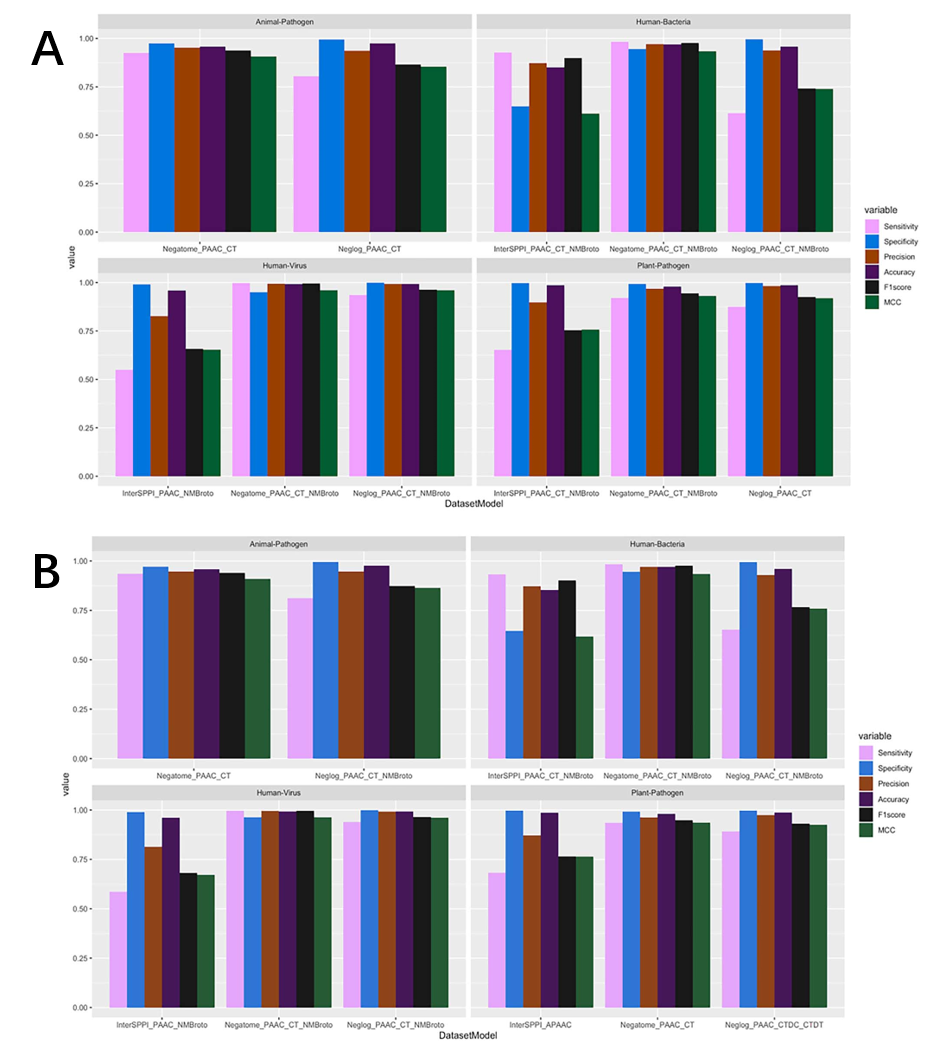
图3.各数据类型最佳模型的敏感性、特异性、精密度、准确度、F1评分和MCC的(A)5次交叉验证和(B)独立测试评估结果。图片来自BIB
负数据集性能比较
为了确定哪个负数据集和真实的PPIs更适合建模,研究者比较了每个模型类型/数据集的最佳特征性能,并讨论了该数据集的优缺点。
总的来说,可以观察到Negatome和Neglog是构建HPI模型的最佳数据集(图3)。以敏感性为主要优化标准的Negatome优于Neglog和InterSPPI,而以特异性为主要优化标准的Neglog优于Negatome和InterSPPI。
混合模型取得了最好的结果。杂化伪氨基酸组成、共三元和归一化Moreau-Broto特征(PAAC_CT_NMBroto),杂化伪氨基酸组成和归一化Moreau-Broto特征(PAAC_NMBroto)。根据交叉验证结果,PAAC_NMB roto特性表现最佳。同时,杂合伪氨基酸组成、组成和过渡特征(PAAC_CTD C_CTDT)、杂合伪氨基酸组成和并三联特征(PAAC_CT)、PAAC_NMBroto和PAAC_CT_NMBroto在独立测试中表现最好。
以下是研究者的开发模型在几个案例中的表现:
1. 植物病原性HPIs模型特征的表现:研究者选择PAAC_CTDC_CTDT和APAAC作为植物病原菌,在 deepHPI服务器上实现的模型,其中一个模型灵敏度最高,另一个模型速度最快。
2. 人类-细菌HPIs建模特性的性能:基于数据集的性能评估,研究者选择了PAAC_CT_NMBroto和CT作为在deepHPI服务器上实现人类-细菌的模型,其中一个是最敏感的选项,另一个是最快的选项。
3. 人类-病毒HPIs建模特性的性能:研究者将PAAC_CT_NMBroto和CT应用于deepHPI预测人类-病毒HPIs,其中PAAC_CT_NMBroto模型灵敏度最高,CT模型速度最快。
4. 特征在动物-病原体HPIs建模中的表现:研究者将PAAC_CT_NMBroto应用于deepHPI,作为预测动物-病原体HPIs的敏感模型;同时,CT作为快速模型在deepHPI中实现。
deepHPI web服务器及实现
web服务器提供了两个可预测的选项,敏感和快速。为了实现对植物-病原菌、人类-细菌、人类-病毒和动物-病原菌的敏感性预测,研究者分别采用了PAAC_CTDC_CTDT、PAAC_CT_NMBroto、PAAC_CT_NMBroto、PAAC_CT_NMBroto模型。在快速预测选项APAAC下,分别使用植物病原体、人类细菌、人类病毒和动物病原体的CT模型。
在可视化页面中,有蛋白质节点度、基因本体术语的信息,以及到公共数据库(UniProt、AmiGO和NCBI)的链接 (图4B)。用户还可以下载SVG或JSON格式的网络,可以在Gephi、Cytoscape等网络分析软件中打开。
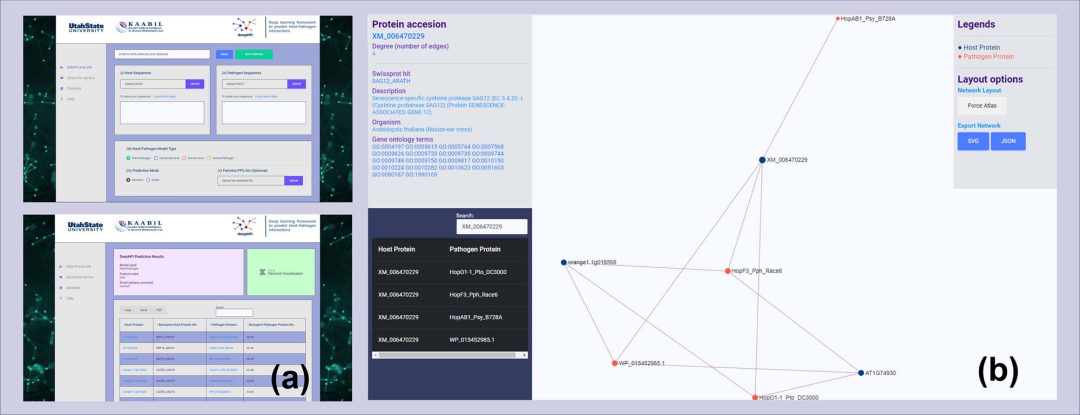
图4. deepHPI接口。图片来自BIB
与其他现有工具的比较
为了对模型进行性能评估,研究者将deepHPI与现有工具HVPPI进行了比较。在HVPPI web服务器中,每个查询有3个序列的限制,而在deepHPI上,每个查询的限制是10,000个序列。研究者下载了三个人类蛋白(P15822、Q05086和Q6FHJ7)和三个SAR-Cov2蛋白(E、M和N),并在两个服务器上进行预测。HVPPI预测了三种蛋白质之间的相互作用,而deepHPI预测了九种蛋白质之间的相互作用。由于研究者的模型包含了来自不同数据库的数据,deepHPI预测了更多的交互,准确率更高。
总结
通过这项工作,研究者评估了CNN模型预测宿主病原体PPIs的性能。结果表明,混合特征模型在每种数据类型(植物病原体、人类细菌、人类病毒和动物病原体)中都表现不错。研究者在deepHPI web服务器上实现了最好的模型,从而能够预测和可视化产生的宿主病原体网络。
参考文献
Kaundal, Rakesh, et al. “deepHPI: a comprehensive deep learning platform for accurate prediction and visualization of host–pathogen protein–protein interactions.” Briefings in Bioinformatics 2022, 23(3), 1-11.