
引言
网络理论为研究复杂系统提供了有效的分析工具。意大利博洛尼亚大学Maurizio Recanatini教授近期在JMC综述了网络科学在药学研究中的应用。作者首先介绍了构建网络的数据来源,然后展示如何使用网络研究药物相关系统的一些范例,其中专门介绍了基于网络推理的应用以及布尔网络动力学(Boolean networks dynamics),最后作者认为在未来几年中,结合机器学习和3D建模方法的网络应用程序将成为计算药物发现中必不可少的工具。
背景
复杂性是生命系统的一个显着特征,网络科学允许捕获整个系统的行为,尤其是在系统出现的特性方面,这些特征是系统各部分之间相互作用的结果,而不仅仅是它们的总和。网络用点和线分别表示元素和元素之间的关系。图论解决了网络的数学描述,使得可以用图代表网络。网络的元素称为节点(nodes),它们之间的连接被称为链接(links)或边(edges)。基于网络的方法在一定程度上已经影响了寻找新疗法的方式。将网络理论应用于药物发现的方法有很多,在药物化学/药物设计领域中,使用基于网络的方法的主要方向是靶标识别(target identification)和药物重利用(drug repurposing),进一步的应用还有化学空间的分析和药物不良反应或毒性的预测。
数据与数据库
在构建网络时,要考虑的第一个也是最重要的问题是我们用于构建这些模型的材料,即我们通常所说的“数据”。高通量实验技术所提供的信息正在以前所未有的速度增长。如今,我们可以访问几乎每个疾病治疗领域的涵盖数百万个分子以及数千种蛋白质和基因的化合物、靶标和疾病的数据库。
化学数据库
根据其内容可以将公共化学数据库分为六类,即(1)化学信息,(2)生物活性,(3)药物,(4)天然产物,(5)商业可得性,(6)片段。通常,化学数据库中包含的所有类型的数据都可用于药物设计目的,但对于涉及网络应用的问题,最有用的是生物活性、药物和天然产物的数据。在这方面,最受欢迎的数据库是CHEMBL和PubChem,它们提供有生物活性的化合物的信息,尤其是活性测定和靶标信息的数据。DrugBank则包含已经批准和还在实验过程中的药物的数据,可以成为靶标识别和药物重利用研究的重要信息来源。在纯化学方面,ChemSpider是理化和光谱数据以及化合物名称、同义词和标识符的非常丰富的来源。在表1中,总结了上述数据库的主要功能。

图片来源JMC
使用化学数据集时要考虑的最重要问题是其内容的预处理,这是一项关键操作,可能需要很长时间,但必不可少,以降低获得误导性结果或建立错误模型的可能性。特别是,数据集的处理应考虑化学、生物学和项目标识等多个方面,例如,化学结构的表示和非标准化结构(盐,离子等)在数据集中的存在,针对相同化合物的不同的生物活性数据,通过不同实验室得到的结果的可重复性,活性缺失、拼写错误或标签错误的化合物以及不正确的标识符。
生物数据库
从系统角度看待靶标识别或药物重利用研究时,必须在网络中包含由大量生物技术所产生的越来越多的生物学数据。《核酸研究》杂志于2019年出版的《分子生物学数据库收集》(Molecular Biology Database Collection)列出了1613个数据库列表,简要描述了新条目并最终更新了旧条目。这些数据库可以分为:(1)核酸序列和结构以及转录调控;(2)蛋白质的序列和结构;(3)代谢和信号传导途径、酶和网络;(4)病毒、细菌、原生动物和真菌的基因组学;(5)人类和模式生物的基因组学以及比较基因组学;(6)人类基因组变异、疾病和药物;(7)植物;(8)其他。
对于药物设计领域可能感兴趣的是,例如那些包含有关蛋白质信息的数据库,这些数据库既可以是一般序列(例如SMART,UniProt),也可以是单个蛋白质家族的序列(例如GPCRdb,Kinomer);蛋白质结构或蛋白质-蛋白质相互作用(例如PDB,STRING);代谢和信号传导途径(例如Reactome);人类基因和疾病(例如DisGeNET)。在表2中,总结了这些数据库的主要功能。

图片来源JMC
表型数据
医疗保健的普遍数字化提供了数量非常重要的表型数据来源,主要源于电子健康记录(EHR)、可穿戴设备或应用程序。仅就HER而言,这些文档中的信息包括对个人健康/疾病状态的描述、临床测试结果、药物处方以及最终的不良反应。当然,隐私问题限制了此类数据的可用性,我们无法公开找到可访问的数据库。关于此类信息对药物研究的贡献,我们观察到表型和基因型数据的整合可能是迈向深入了解疾病发作和进展的生物学过程的必要步骤。
建立网络
鉴于有关分子、基因、蛋白质、细胞、组织和疾病的数据的广泛可用性,并且假定这些数据实体相互连接并代表或多或少的复杂系统,因此需要构建和可视化网络。可用于网络可视化和分析的计算工具不计其数,其复杂程度取决于数据集的大小和要执行的任务。Cytoscape是其中最受欢迎的工具之一,当然还有Gephi、Pajek和NetworkX等。(有关软件主要功能的详细信息,请参见表3)。这些软件工具可以可视化最大数量级为106个节点和边的大型网络。但是,当数据量进一步增加时,即使对于功能强大的工作站,由于加载大型矩阵的内存需求从而也变得无法满足,并且需要更高性能的计算来分析网络。解决方案可以是通过诸如Hadoop或Apache Spark之类的框架在大量内核上分发数据和流程。

图片来源JMC
研究特定药物系统的网络
分子数据集分析网络
在有机小分子环境中使用网络分析的一个相关示例是化学空间网络(CSN),这是由G. Maggiora和J. Bajorath提出和开发的框架。该化学空间被视为一个复杂的系统,可以将诸如生物学活性之类的特性与之关联。引入并验证了不同的相似性指标,以及对网络拓扑参数的分析表明,它们是可视化和分析中等大小化合物库的结构-活性关系(SAR)的强大工具。通过适当的度量标准和算法对CSN进行分析,可以发现共有潜在特征的化合物群落(簇)的存在,而这些共同特征不能从通用表格格式中立即看出来。为了说明简单的CSN应用,在图1中显示了62种ADP-核糖聚合酶(PARP)抑制剂的网络。网络解释了化合物之间的关系,它们之间的链接是根据基于指纹计算的成对相似性值得出的。如果抑制剂的结构相似性超过阈值,则由通过边(188)连接的节点(62)表示(请参见图1的图例)。节点根据效能着色。基于相似性计算的化学空间可视化有助于识别PARP抑制剂(网络的主要连接组件)的不同结构族,并且颜色编码使人们可以立即掌握化合物的SAR。
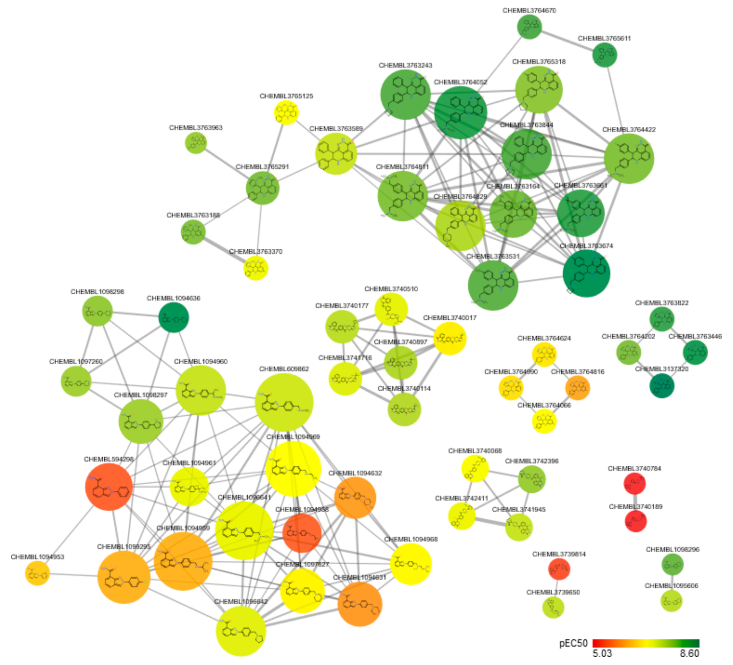
图片来源JMC
蛋白质结构网络
如果我们认为蛋白质像其他任何分子一样是相互作用元素(即氨基酸)的集合,则可以立即得出它作为一个复杂系统的信息,其中结构、动力学和最终功能可以被视为源自氨基酸之间关系的显著性质。在这种情况下,蛋白质结构网络(PSN)得到了广泛研究,随着蛋白质结构测定的分析/生物物理技术的快速发展,该网络方法非常适合处理结构-功能关系。通过将氨基酸(通常为Cα原子)视为通过链接连接的节点(如果它们之间的距离落在临界值之内)来构建PSN。对描述PSNs特征的参数进行分析有助于研究该蛋白的3D结构及其对变构调节、折叠和模型验证等问题的影响。特别地,分子动力学模拟以及最终的结合自由能计算可以协同地应用于药物研究。
人类疾病网络与药物发现
基因调控网络(GRN)、蛋白质-蛋白质相互作用(PPI)网络或代谢网络,是构建人类疾病表型-基因型关系基础的细胞机制的系统视图时必不可少的元素。通过利用系统生物学方法可以将复杂系统的不同层次结合在一起。作为复杂交互组的一个示例,在图2中,展示了一个从DrugBank数据库中生成的药物-靶标网络(DTN)。该网络显示了1636种被批准的小分子药物与1991种人类蛋白质靶标之间的相互作用。边代表7521个唯一的相互作用。从图中可以明显看出,该网络包括一个由3368个节点组成的大型连接组件,其中1510个节点是药物分子。对此类网络的分析提供了分子药理学领域的全局概况,并可能有助于确定药物研究的趋势或未来发展的可能领域。
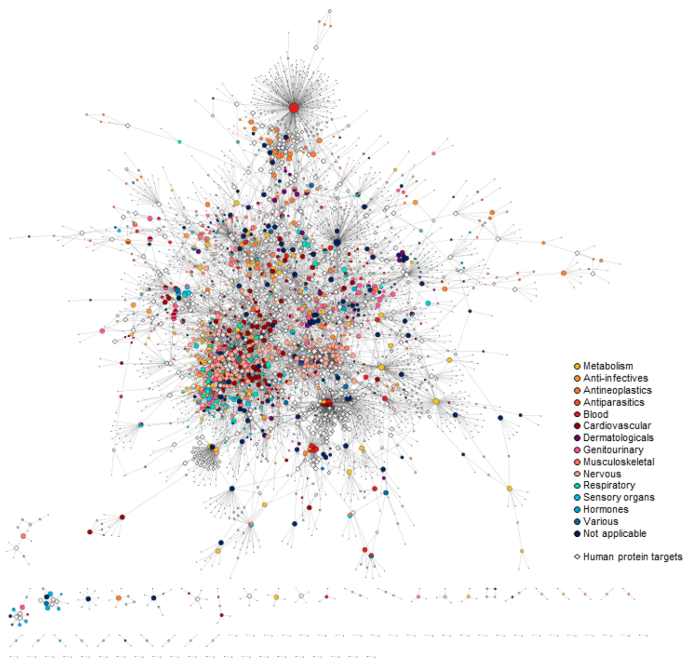
药物-靶点网络
图片来源JMC
基于网络的推理
在处理网络时,一方面要解决信息丢失的问题,这是生物系统研究中的一种常见情况,在这种情况下,难以获得相互作用的实验证据使网络本质上不完整,而另一方面新的数据还在不断增加。这个问题在网络理论中是非常基本的,从更严格的角度来看,推断网络中丢失链接的可能性是对两个尚未连接的节点之间的新链接的预测,具有更实际和直接的意义。
链接预测方法
在实践中,一种尝试预测潜在的药物-靶标相互作用(DTI)的方法通常是从诸如社会科学、通信网络、经济和金融等领域借来的。这些方法的目标是生成潜在DTI的列表,并根据一些预定义的指标对它们进行排名。起点是构建异构网络(heterogeneous network),在该网络上运行链路预测算法。通常,异构网络整合了从不同数据库获得的有关药物、靶标和药物-靶标相互作用的可用信息。
考虑到数据的可用性,这些方法的关键步骤是(1)药物-药物和靶标-靶标相似性的计算和(2)药物-靶标关联推论方法的应用。对于前者,在最初简单地使用指纹和一级序列分别比较药物分子和蛋白质后,已发展出更复杂和信息更丰富的相似性指标以便考虑到已知药物-靶标相互作用信息以及蛋白质和网络拓扑信息。
基于网络的DTI预测方法差异很大,通常取决于用户的偏好或专业知识,但最受欢迎的算法是从推荐算法或网络传播算法派生而来的,它们都属于所谓的基于相似性的算法。基于推荐算法的方法旨在预测基于先前计算的相似性分数(也称为协作过滤),确定节点对未连接节点的偏好。另一方面,在网络传播算法的框架内,包括了几种方法,这些方法通过模拟从种子节点开始的信息在网络中的传播来起作用。最著名的是Google页面排名算法,该算法使用随机浏览网页来计算其重要性。
在药物重利用中的应用
作为基于网络的方法可能对药物重利用产生影响的说明性示例,在这里,我们简要描述一项最近发表的名为研究项目Repethio(https://think-lab.github.io/p/rephetio/)的研究。在这项工作中,作者报告了构建异构网络以捕获药物和疾病之间的联系的情况(Hetionet版本1.0,https://neo4j.het.io/browser/)及其在预测新药/疾病的关联中的用途。Hetionet整合了来自公共资源的数据,由11种类型的约50000个节点与24种类型的约225万条边链接在一起。
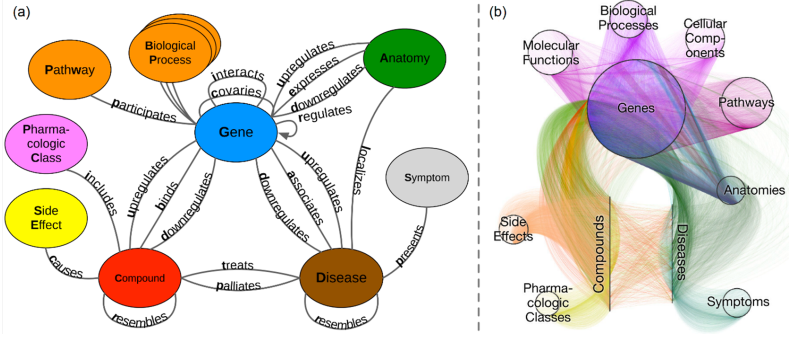
Hetionet 1.0版
图片来源JMC
Repethio项目清楚地解释了基于网络的数据分析如何影响药物研究,同时还考虑了将这种方法与功能强大的ML方法进行特征选择和预测相结合的适用性。此外,这是使用集成到在线平台中的公共数据的示例,该数据反过来向用户开放,这些用户可以访问该数据并利用其本地时间进行时间和资源密集的组装和集成工作。我们设想,越来越多的研究数据可用于公共领域,这种更频繁的举措得以开发,充分利用了大数据和网络科学的综合潜力。
网络动态
如果我们考虑对网络时间演化(即网络动力学)建模的可能性,那么可以将网络科学在药物研究中的应用向前迈出一步。为了了解该领域对于未来药物发现的潜在重要性,有必要简要介绍S. Kauffman于1969年提出的布尔网络(Boolean networks),该布尔网络的一般假设旨在解释控制细胞体内稳态和分化的调节回路。
布尔网络是有向网络,其构建方式是节点是基因,链接表示它们之间的功能连接。每个基因可以“打开”或“关闭”,并且一组规则或更新功能与每个节点相关联,以在随后的时间步长定义基因的状态。系统的动力学是从输入基因开始的,通过在每个离散时间步同时更新基于预定义规则的所有基因状态来计算。给定其构建方式,布尔系统是确定性的,并且具有有限数量的初始网络状态(2N,其中N是基因的数量,而2表示打开和关闭的两个状态)。经过多次迭代(时间步长)后,它将达到一个稳定的状态,可以是一个固定点或一个自循环的圆:可以按照不同的网络状态序列(轨迹)访问这种网络状态,并且它似乎吸引了系统,因此被定义为吸引子(attractor)。一个布尔网络可能存在多个吸引子,它们的集合以及从初始状态到吸引子状态的轨迹共同构成了attractor landscape。应当指出,布尔网络可以在正常条件下模拟系统的动态状态,但是它也可以通过修改更新规则,即设置某些节点的开或关而受到干扰。已经表明,吸引子状态对应于对外部刺激的细胞表型,因此,探索吸引子态导致了表型态的定义,其中表型包括所有网络导致系统指向相同吸引子的状态。
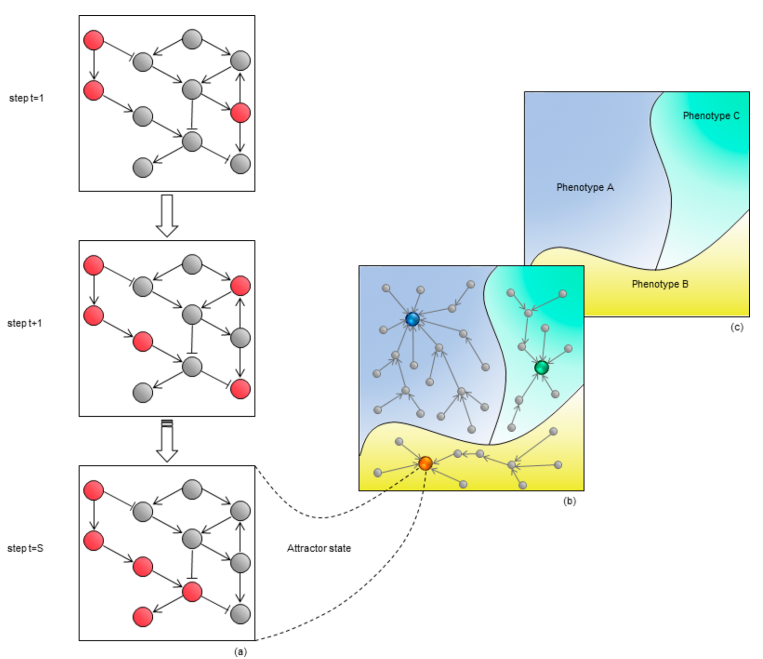
布尔网络动力学
图片来源JMC
总结
在新药的设计中,基于系统的网络模型所描述的场景非常有用并具有启发性。基于网络的观点可以让人们扩大视野,从中可以考虑药物发现过程。这不仅仅是技术上的改进,它最终还可以导致设计出替代性的药理干预范式。此外,如何将网络建模方法与目前在药物设计/发现中使用的成熟的计算技术(即分子建模以及机器学习)集成在一起也是一个值得思考和关注的问题。机器学习和深度学习方法已经在计算药物发现中被广泛使用,它们非常适合集成到网络构建技术和基于网络的预测方法中。
最后,必须强调基于网络的药物发现方法的局限性和开放性挑战。第一个问题是数据质量,即数据的类型和来源。此外,某些生物学数据可能是不完整的、有偏见的或稀少的,还有用于构建数据库的语言可能是不同的和/或不兼容的。所有这些严重限制了甚至建立网络的可能性。第二个挑战与数据的大小有关,因此也与网络有关。根据节点的数量和节点的度数,要计算的链接数量可能会大大增加,这再次限制了构建或分析网络的可能性,即使某些工具允许一个人处理多达数百万个节点和边。但是,随着分析方法变得更加详细或复杂(例如,网络动态),计算需求变得令人望而却步。克服计算问题的一种方法是将工作负载分布在云上,这是目前看来最好的技术选择,同时等待量子计算机的有效应用。
参考文献
Drug Research Meets Network Science: Where Are We? Maurizio Recanatini and Chiara Cabrelle. Journal of Medicinal Chemistry Article ASAP DOI: 10.1021/acs.jmedchem.9b01989