

嗅觉受体 (OR) 的氨基酸序列如何编码对各种配体的多样化反应?一个基于 OR 序列相似性和配体物理化学特征的蛋白质化学计量学 (PCM) 模型可以使用监督机器学习预测 ORs 对气味的反应。命中率高达 58%,发现了 64 对新的 OR-气味剂。
背景介绍
嗅觉受体(ORs)是G蛋白偶联受体家族的一类成员,具有敏感的气味辨别能力。但是受体序列中微小的差异可能表现出不同的功能,同时不同的序列也可能会表现出相似的功能。与OR结合的配体是各种各样的气体分子,OR 通过变构机制对配体作出反应。目前研究认为,配体亲和力是由口袋残基决定的,对相同气味有反应的 OR 在口袋周围具有更高的序列同源性。
OR 可区分大量挥发性分子并编码大脑中感知到的气味。OR 和气味之间的映射关系是理解气味感知的关键。机器学习在预测 OR 对气味的反应的研究中已经取得了巨大成功,但是稀疏的气体数据限制了模型的效果。目前常用的策略是通过特征选择来降维,从OR序列中筛选出重要残基,可改善模型效果。本文作者使用 OR 序列比对、 PCM和机器学习来预测 OR 对各种气味的反应。PCM 模型可提取 OR 序列相似性和气味剂的化学特征。除此之外,作者还在体外评估了相关残基的选择和模型预测结果。
主要内容
OR-气味配对数据集准备
本文作者检索了文献中hORs(人嗅觉受体)和 mORs(鼠嗅觉受体)与气体的样本数据,包括 1293 个 OR-气味剂对,由 390 种 OR受体和 244 种气味剂组成,然后又纳入了14400 对阴性气味剂,阴性气味剂在体外无反应。作者选用苯乙酮、香豆素、R-香芹酮和 4-色原酮作为目标气味剂,随后又加入了与 4 种目标气味剂具有相似化学结构的气味剂的数据。
分子模型建模
首先寻找正位配体结合口袋内的气味结合残基。小鼠OR作为原型,它对四种目标气味剂中的三种有反应。分子动力学模拟建立了与气味剂结合的3D同源模型。3D 模型是在保守氨基酸基序和覆盖 TM 结构域近 50%(95 个残基)的定点诱变数据的约束下构建的。在结合气味剂分子的 5 Å 距离空间内含有 17 个残基(以下称为 poc17,图 1B)。这些残基中的 14 个已被证明对通过定点诱变对气味的 OR 反应很重要。
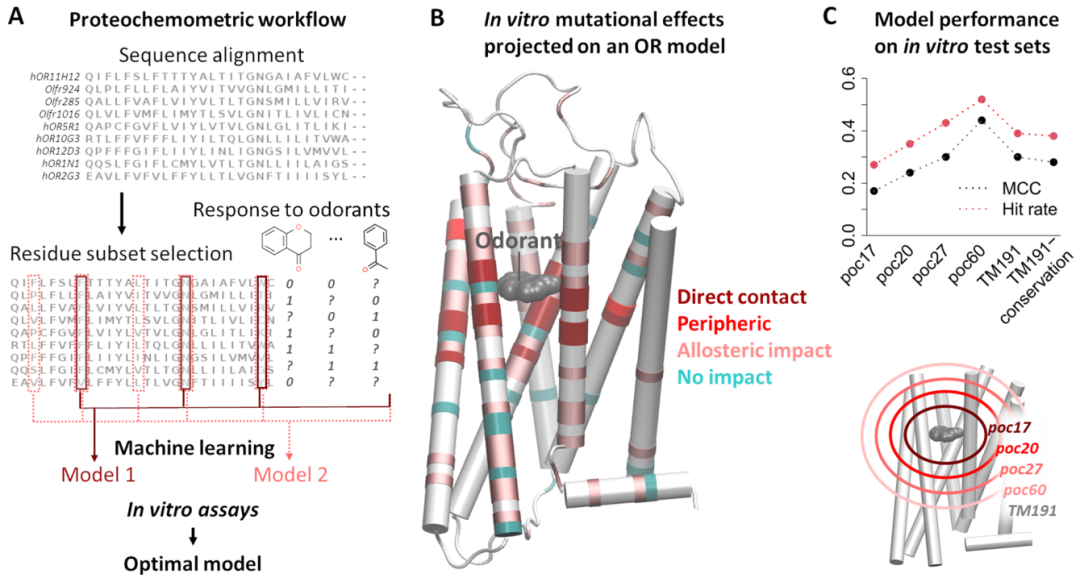
图1. 机器学习流程与残基选择。图片来源:AcsCentSci
结合位点残基突变
作者在poc17 周围诱导了 24 个点突变。体外剂量依赖性反应可以测定突变受体对五种配体的反应的影响。突变效应与文献中报道的所有 OR 突变都标注到了3D 模型上(图 1B)。其中包括 poc17 和 3 个外围残基在内的 20 个残基,组成了一个更大的正构口袋 (poc20)。poc20 内的突变影响了受体对大多数气味的反应。
作者尝试寻找最佳残基子集。根据上述结果选择了 5 个从小到大的残基子集:poc17、poc20、poc27、poc60 和 TM191。其中poc27 和 poc60 是口袋的延伸,距离结合气味剂周围 6 和 8 Å,分别包含 27 和 60 个残基(图 1C)。TM191 包含由 191 个残基组成的整个配体结合的区域。作者使用这些残基子集构建机器学习模型。
PCM与机器学习
从hORs和mORs的序列比对中,作者提取了5个启发式残基子集。PCM 模型是使用表 1 中的数据和气味剂的物理化学特征构建的。作者使用 5 折交叉验证训练和评估支持向量机 (SVM) 和 随机森林模型(RF)。模型预测了每个 OR-气味对的响应概率,概率 > 0.5 被归类为响应。Matthew 相关系数 (MCC) 用于衡量模型效果。结果表明RF 的表现优于 SVM。五个 RF 分类器的预测性彼此之间没有显著差异。
poc60 分类器平均表现最好。为了确定最佳残基子集,使用表 1 中 100% 的数据构建了五个最终 RF 分类器(poc17、poc20、poc27、poc60 和 TM191)。然后使用每个分类器筛选4个目标气味剂的新 OR 。每个分类器预测并给出OR对 4 种气味中各自预测的概率。
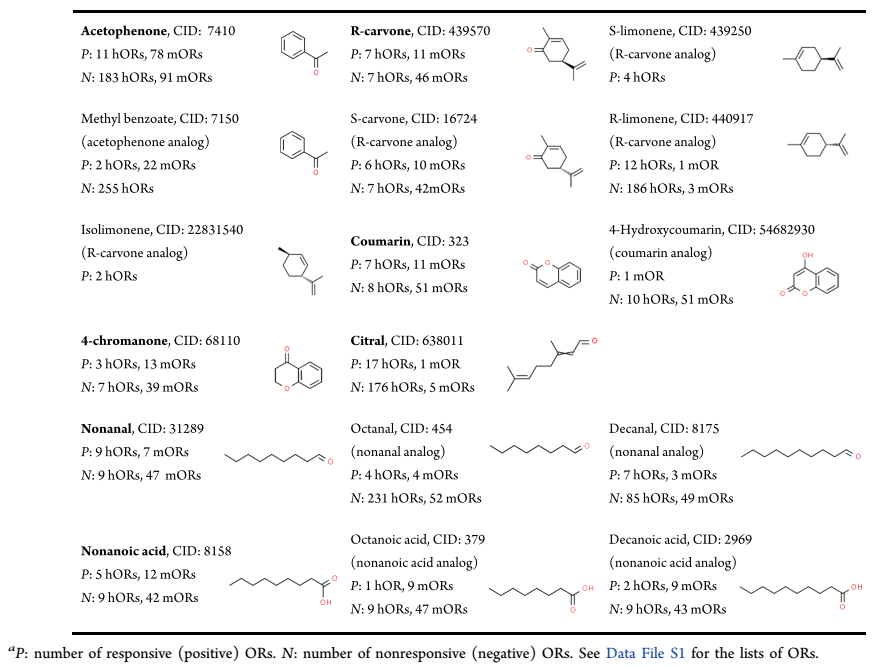
表1. 查询的气味剂(粗体)的化学结构、PubChem CID、训练数据及其类似物的信息。表格来源:AcsCentSci
相关残基的体外评价
作者在细胞功能实验中测试了所有五个分类器的预测。对于每个模型,作者测试了响应类中的所有 OR(任何气味的预测响应概率 >0.5)以及 60 个阴性对照 OR(异味剂的响应概率 <0.5)。这些 OR 已针对所有 4 种气味剂进行了测试。例如,在 poc60 的情况下,作者测试了响应类中的所有 20 个 OR,并从非响应类中随机挑选了 60 个阴性对照(图 2)。
对其他四个模型进行了类似的测试。当在 300 μM 观察到显著反应时才测量了剂量依赖性反应。否则,OR-气味是无反应的。poc60 分类器在体外测试集上表现最好(图 1C)。它对4 种气味剂的命中率在0.39-0.60, MCC在0.43-0.48之间(表 2)。因此,poc60 是受体对气味反应的最相关的残基子集。这些残基在 hORs 和 mORs 中表现出低保守型和高多样性,适应各种气体分子。这意味着 OR 序列中的氨基酸保守序列包含其功能的基本信息。因此,作者使用 TM 区域中的氨基酸保守性测试了另一个模型。事实证明,该模型几乎与使用氨基酸物理化学特征一样具有预测性(图 1C)。这表明用于描述氨基酸的特征类型并不重要,只要特征足以将序列差异传达给机器学习算法即可。
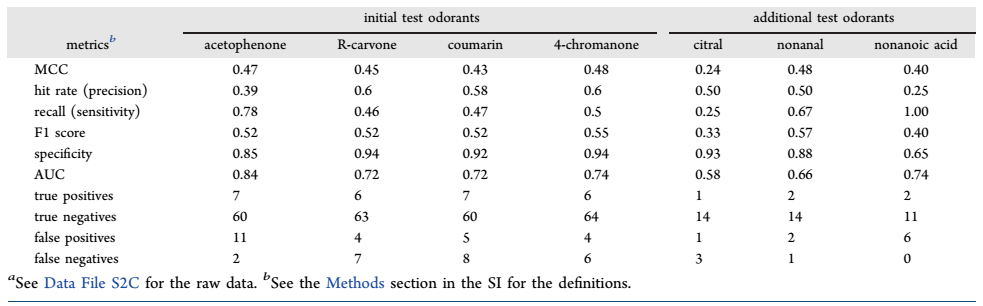
表2. poc60模型在预测新OR-气味剂的表现效果一览。表格来源:AcsCentSci
模型在其他气味上的预测
缺乏气味数据是模型实用性的主要限制因素。为了探索这一限制,作者通过 poc60 模型在外部测试集上的预测与使用的训练数据量的学习曲线,证明对于具有 15 个已知 OR 的气味剂,模型可以获得有意义的预测。在数据集的244 种气味剂中,只有 17 种 (7%) 符合此标准,其中 11 种含有芳香或环状结构。作者尝试了另外三种含有烷基链的气味剂:柠檬醛、壬醛和壬酸。按照相同的程序,作者在体外测试了所有 11 个预测对这三种气味中的任何一种以及 8 个阴性对照 OR 有反应的 OR(图 2B)。体外试验表明,该模型在壬醛和壬酸上表现良好,但在柠檬醛上表现不佳。
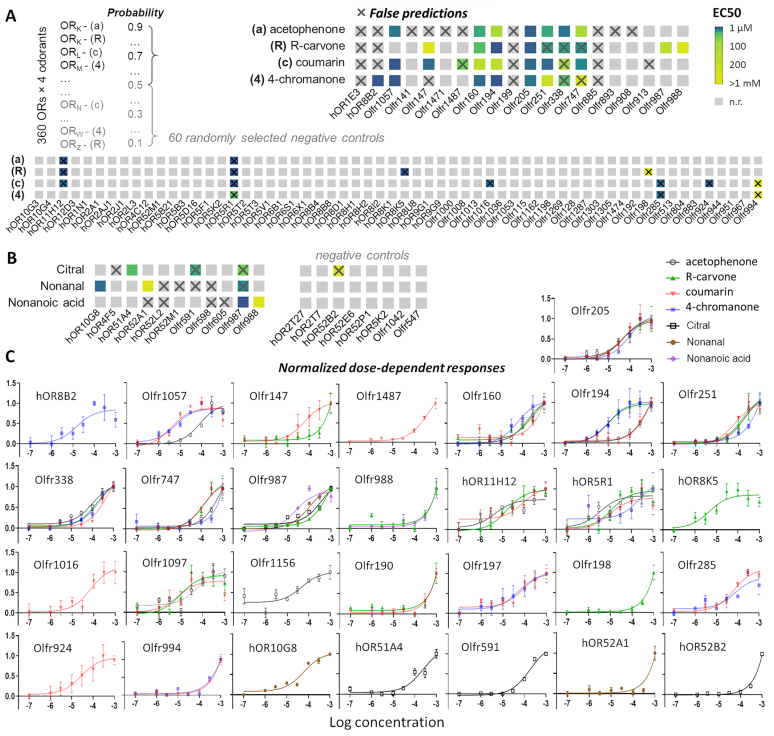
图2. 机器学习模型预测的OR-气味剂响应对的体外评价结果。图片来源:AcsCentSci
模型的泛化能力
作者评估了 poc60 模型在所有外部测试集数据上的泛化效果。五折交叉验证中,各折的命中率在 0.39-0.46之间,MCC 为 0.32-0.34。测试集中含有更加多样的OR和大量阴性对照的OR,Hana3A 细胞的 OR-气味筛选命中率低于 0.1。由于许多 OR 不能在异源细胞的膜上表达,因此作者难以估计ORs 对各种气味剂的一般反应率。作者在这些测试中,发现了 63 个新的 OR-气味剂对,涵盖了29 个 OR(见图 2C)。
该模型的特点在于具有较好的泛化能力。虽然外部测试集中的 95 个 ORs 中有 56 个是“新的”,模型仍然显示出良好的预测性。作者将 7 种气味剂从训练集中一一排除,重新训练模型,并在仅包含排除气味剂的测试集上计算性能指标,展示了对环状气味剂、苯乙酮、R-香芹酮和 4-色满酮的预测性(表 S8)。新气味剂的应用目前受到缺乏训练数据的限制,但是该模型特别适用于没有训练数据的新 OR。
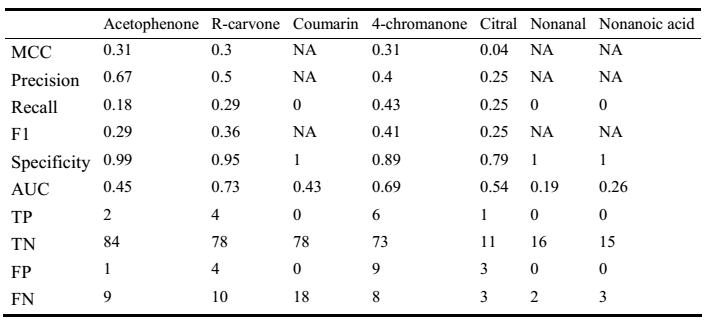
表S8. Poc60模型在“新”气味剂上的表现。表格来源:AcsCentSci
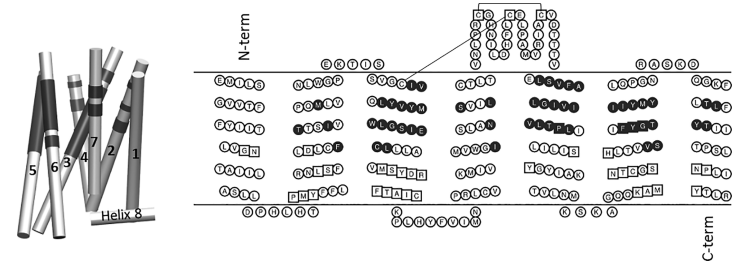
图3. 最佳响应模型中的残基位置分布图。图片来源:AcsCentSci
总结
本文展示了如何从OR和气味剂的配对样本出发,结合口袋周围的60个残基,预测OR对气味反应的变化。作者使用了不到20%的残基序列即可完成预测反应。在这个过程中,作者发现了保守序列附近四个关键的残基。建立的模型优于使用完整序列建立的模型。
OR配体的分子建模可以增加配体的信息,降低PCM模型的难度。后期研究者可以添加新的OR-气味剂样本改善模型效果。作者提出的建模方案主要侧重OR的结合区域序列的关键残基分析,它可以绕过高分辨结构和全序列比对的难题。基于人工和机器学习模型的特征选择则可以增强模型在稀疏数据集上的表现。如果将模型与定点突变等手段相结合,科研人员有望分析出OR特定功能与序列的关系。
参考文献
Xiaojing Cong, Wenwen Ren, Jody Pacalon, Rui Xu, Lun Xu, Xuewen Li, Claire A. de March, Hiroaki Matsunami, Hongmeng Yu, Yiqun Yu, and Jérôme Golebiowski. Large-Scale G Protein-Coupled Olfactory Receptor−Ligand Pairing, ACS Cent. Sci., 2022, XXXX, XXX, XXX-XXX.