

一种肽束缚策略,可用来筛选构象确定的多肽上的非天然侧链片段。
背景介绍
合理设计的蛋白质二级和三级结构,可作为生物分子相互作用的调节剂。控制蛋白质模拟设计的假设是,蛋白质表面的元素可以被缩小,以获得细胞渗透性、蛋白质水解抗性的配体,这些配体保留了天然蛋白质的结合相互作用。然而,这些模拟物往往有一个固有的局限性:根据定义,蛋白质的最小模拟物并不包含蛋白质的所有接触点,而接触点的丧失与同源受体的结合亲和力和特异性的降低有关。这一挑战的潜在解决方案包括增强带有亲电弹头的合成模拟物,以引起与受体的共价接触或包含非天然侧链,从而弥补由于无法与天然残基接触的口袋的非共价接触而导致的结合亲和力损失。
转录共激活因子CBP和p300的KIX结构域是转录因子的对接位点。图1A显示了在KIX的两个变构连接面上结合cMyb和MLL的反激活域的NMR模型。一个转录因子与KIX的结合增强了另一个转录因子的结合,这突出了KIX的可塑性。KIX与异常转录因子的相互作用会导致基因异常表达和疾病。人们已开发了几种方法,寻找一个或两个KIX结合表面的配体以阻断转录因子共激活因子的相互作用,这些方法包括结构引导设计、基于核磁共振的筛选、高通量筛选、噬菌体展示和二硫蛋白束缚等。由于KIX的柔性和其表面缺乏相邻的结合袋,使得发现有效配体变得困难──从上述策略设计或分离出来的许多小分子和多肽,在高微摩尔到毫摩尔范围显示出弱亲和力。
之前的研究中,研究者使用计算算法AlphaSpace来绘制蛋白质表面的地图,并在模拟肽上设计自然和非自然的侧链。该工具基于蛋白Voronoi图的几何特征开发,可用于蛋白表面结合口袋的鉴定,并设计非天然侧链增强配体对这些未被充分利用的结合口袋的亲和力和特异性。
AlphaSpace分析表明,几个MLL残基并没有最优地占据KIX上最近的平坦口袋(图1B),而非自然残基可以被设计成捕获隐蔽口袋。应用AlphaSpace可以显著改善最小MLL基序;优化后的序列具有多个非天然残基,与较短的天然序列相比,与KIX结合的亲和力为低微摩尔亲和力。具体来说,计算设计预测S-苄基半胱氨酸(代替M850)、2-甲基苯丙氨酸(代替F852)、异亮氨酸(代替V853)和酪氨酸(代替T857),可以成功占据KIX上的口袋(图1B)。
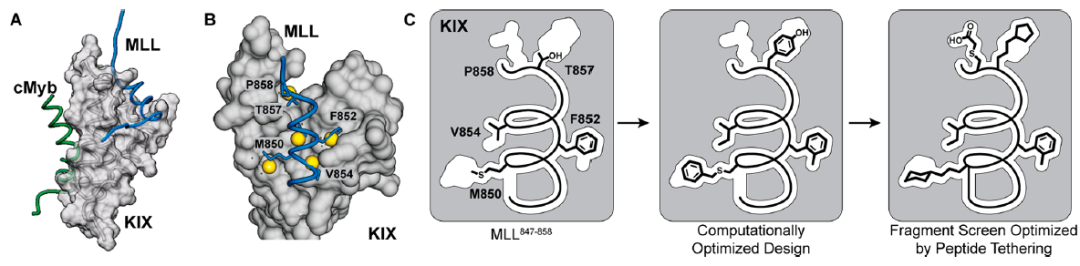
图1. KIX相关结构域和相关肽链。图片来源:JACS
主要内容
美国纽约大学的Paramjit S. Arora等人以计算方法为基础,描述了一种实验片段筛选方法,以确定与KIX接触的合成侧链。在蛋白质束缚或位点定向配体的发现中,利用工程或天然半胱氨酸残基与库中的片段形成共价连接并引导单个片段进入邻近的蛋白口袋。束缚增加了片段的局部浓度,与未束缚时相比,前者允许在较低浓度时对片段进行筛选。研究者设想肽束缚包括在合理设计的拟肽上放置反应基团,而不是蛋白质本身。在片段修饰的肽与蛋白孵育之前,该片段以共价方式连接到含有荧光团的肽上。片段对结合的相对影响通过荧光偏振分析来定量。计算分析表明,芳香族或脂肪族片段会导致结合增强,因为正在研究的口袋大部分是疏水性的。基于这一假设,肽上的半胱氨酸基团被修饰成一个主要由脂肪族和芳香基团组成的片段库。研究者在MLL肽上选择了三个不同的位置进行优化。在单个位点获得了KIX的亚微摩尔配体,从而得到了将识别侧链片段能力提高超过2000倍的计算-实验联合策略。相关的研究成果以“Peptide Tethering: Pocket-Directed Fragment Screening for Peptidomimetic Inhibitor Discovery”为题发布在国际著名期刊JACS上。
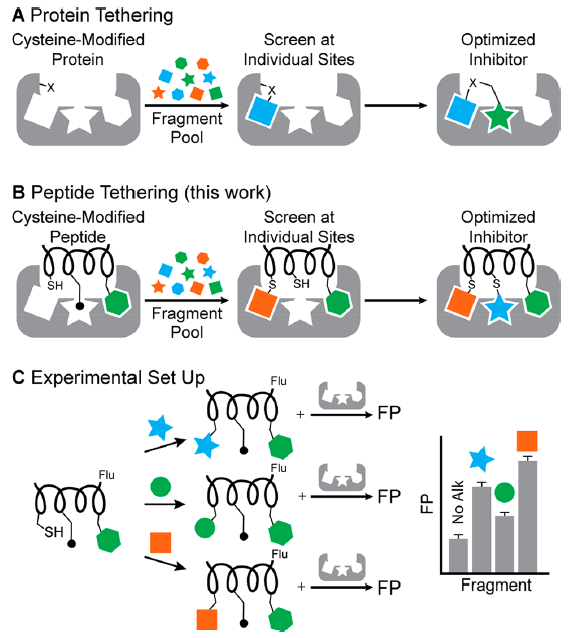
图2. 蛋白质、肽束缚以及实验装置。图片来源:JACS
肽束缚筛选侧链片段的MLL螺旋
研究者在MLL序列上采用肽束缚策略,以进一步改进计算过程。通过将先导线性肽转化为共价约束螺旋模拟物来开展实验筛选研究。用氢键替代(HBS)策略来稳定螺旋构象(图3A)。AlphaSpace分析表明,原生残基M850、F852、V853、T857和P858可以得到优化。用甲基苯丙氨酸和异光氨酸分别取代F852和V853,会导致同源袋几乎被完全占据,但需要通过实验筛选来确定850、857和858位置的最佳残基。将侧链片段筛选集中在计算优化的序列上,并寻找苄基半胱氨酸850、Y857和P858的类似物(图3B)。研究者合成了三个亲本HBS螺旋用于肽链筛选,半胱氨酸残基分别位于850、857和858位。
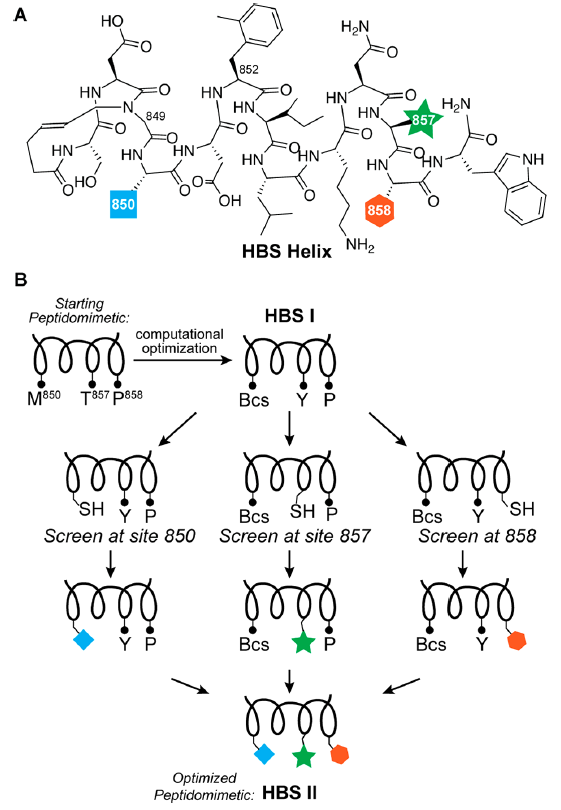
图3. 肽束缚筛选侧链片段的MLL螺旋。图片来源:JACS
作者创建了一个由芳香族和脂肪族片段组成的片段库,用其初步分析肽栓系提供优化配体的潜力。在一锅法合成中,烷基硫醇被预活化为5-硝基吡啶混合二硫化物(图4B)。片段与荧光标记肽孵育,经质谱确认,稀释后单独与蛋白孵育,然后分析荧光偏振(图2C)。荧光偏振测定提供了烷基化片段与KIX接触的电位的直接读数。
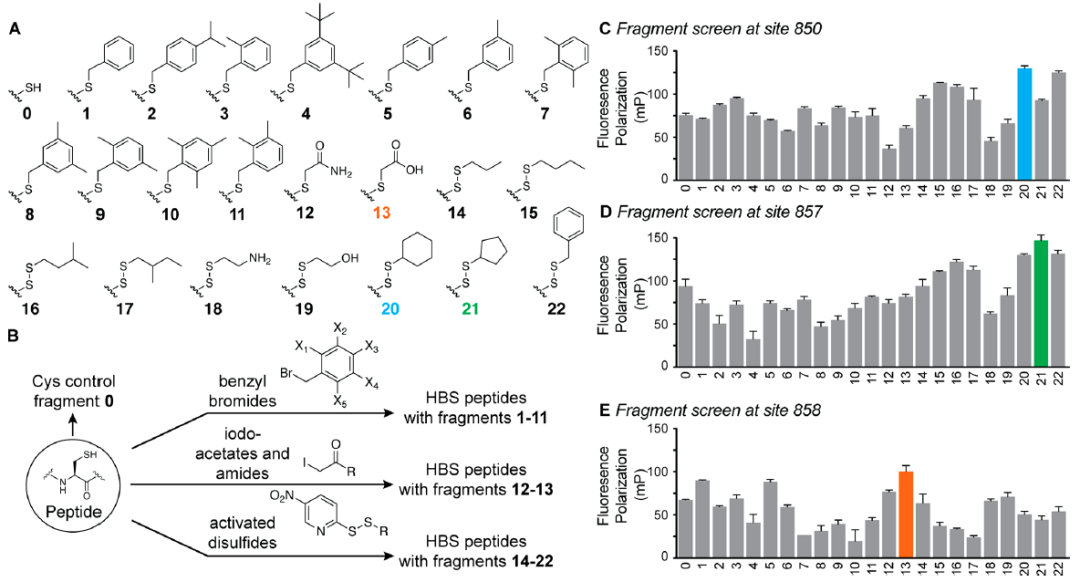
图4. 相关片段的实验研究。图片来源:JACS
图4C-E描述了850、857和858位点的片段筛选结果。柱状图显示了15 nM肽库和40 μM KIX的荧光偏振强度。850位点的筛选结果验证了计算结果。值得注意的是,研究者发现该位点上的疏水残基是有利的,这与之前对850位点上苄基半胱氨酸片段的AlphSpace预测一致。此外,实验筛选显示,环己基二硫片段20比芳香环更有可能在这个位置(图4C)。受到850位点筛选结果的启发,研究者评估了857和858位点的最佳片段。结果表明,环戊基片段21是857位点的首选候选(图4D),而碘乙酸片段13是858位点的首选候选(图4E)。
非天然侧链对MLL多肽结合亲和力的影响分析
研究者重新合成了一种HBS螺旋结构,该螺旋结构包含了来自这三种筛选器的先导物片段,以评估肽束缚法获得高亲和力结合物的成功。研究者推断,表面暴露的二硫键的有限稳定性可能限制了在生物分析中多肽模拟的潜力,并用碳氢键取代了用于片段筛选的二硫桥,从而获得HBS II(图5A)。接着,研究者评估了HBS II在竞争荧光偏振试验中抑制KIX-MLL相互作用的潜力。HBS II显示亚微摩尔抑制常数(Ki= 480±130 nM),这相当于与野生型MLL序列(MLL847-858)的结合亲和力增强约2000倍,与计算设计的多肽i相比,结合亲和力增加约50倍(图5B)。
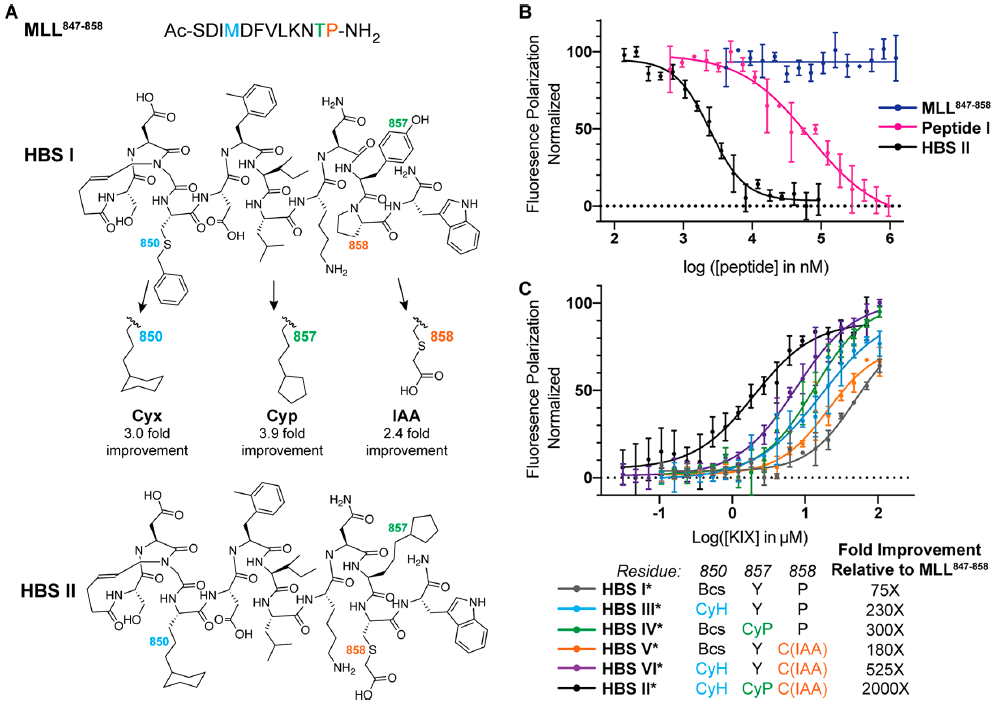
图5. 非天然侧链对MLL多肽结合亲和力的影响分析。图片来源:JACS
研究者在HBS II的850、857和858位点加入了一个实验筛选的单一片段,并测试了直接结合的能力(图5C)。结果表明,每个片段提供了一个约2到4倍的改进,与多价体系一致,每个接触点提供了一个小的结合能增益。这种结合模式支持了多个片段的加和效应,可以提供高亲和力配体的假设。
最后,研究者利用HSQC 15N核磁共振波谱滴定法,来评价HBS II在KIX上的结合位点。将0.5 mol和1 mol等量的HBS II加入到15N标记物中,会导致位于KIX MLL和cMyb面上的KIX主酰胺质子的化学位移发生变化(图6A)。图6B显示了MLL/KIX/cMyb蛋白复合物(PDB 2AGH)上主链酰胺的化学位移变化。结果表明,K659的位移最大,它位于MLL的链部附近,预测会与HBS II的850位接触。
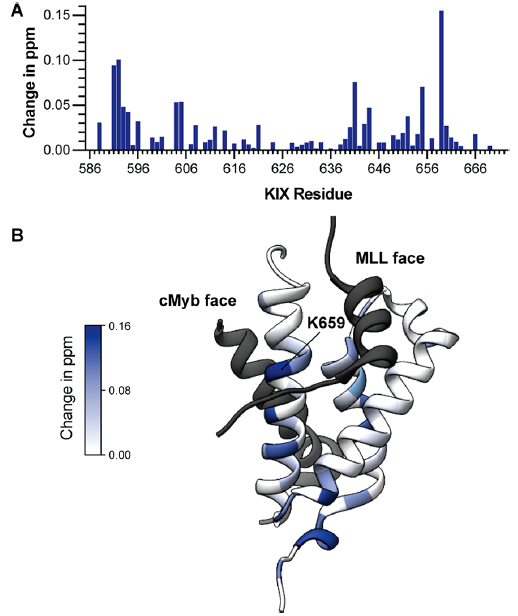
图6. 相关实验测定。图片来源:JACS
总结
本文介绍了一种计算和实验相结合来开发具有非典型侧链的蛋白质类似物的方法。计算方法AlphaSpace提供了蛋白质表面的地形图,并确定了未被充分利用的目标口袋,这些口袋可能被非自然侧链基团更好地占据。在计算方法的基础上,用实验方法筛选结合在特定的口袋侧链片段。研究者用该方法成功开发出KIX的肽拟似物,一种很难用传统设计和筛选策略开发的共价激活剂。与野生型序列相比,亲和力提高了2000多倍,从而促进了亚微摩尔抑制剂的KIX/MLL络合物的形成。
参考文献
Modell, Ashley E., et al. “Peptide Tethering: Pocket-Directed Fragment Screening for Peptidomimetic Inhibitor Discovery.” Journal of the American Chemical Society 2022 144 (3), 1198-1204 DOI: 10.1021/jacs.1c09666