简介
目前,一个上市的药物平均需要花费20-30亿美元,并且需要10年左右的研发时间。大部分经费都花费在了昂贵且耗时的湿实验部分,初始Hits阳性率太低以及(临床前)阶段的高损耗率。使用基于结构的虚拟筛选,Hits质量随着筛选化合物的数量而提高。尽管存在大量的化合物数据库,但是缺乏有效的灵活的方式使用计算机集群进行大规模的SBDD的手段。本文介绍VirtualFlow,这是一个高度自动化的开源平台,可以有效的准备化合物库并进行超大规模的虚拟筛选。VirtualFlow能够使用各种强大对接程序。本文准备了目前已知的最大的免费使用的配体库,配体库包含了超过14亿个可商业购买的分子。VirtualFlow可以探索广阔的化学空间,并可以准确的识别与目标蛋白具有高亲和力的分子。
VirtualFlow的特征
VirtualFlow可以在任何资源管理器上使用,例如:SLURM(https://slurm.schedmd.com),Moab / TORQUE(http://www.adaptivecomputing.com),PBS(http:// www.pbspro.org),LSF(https://www.ibm.com/us-zh/marketplace/hpc-workload-management)和SGE(http://gridscheduler.sourceforge.net)。VirtualFlow还可以理想地配置用于云计算平台,例如亚马逊的Web服务(AWS),微软的Azure和Google的云平台(GCP)。VirtualFlow能够在流程中自主计算配体库中的所有分子,该功能可通过自动提交批处理系统作业来实现,可以在运行时监视和控制流程。VirtualFlow程序包由两个可无缝协作的应用程序组成:VFLP(用于配体准备的模块)和VFVS(用于虚拟筛选的模块)。配体准备和虚拟筛选的过程是分离的。
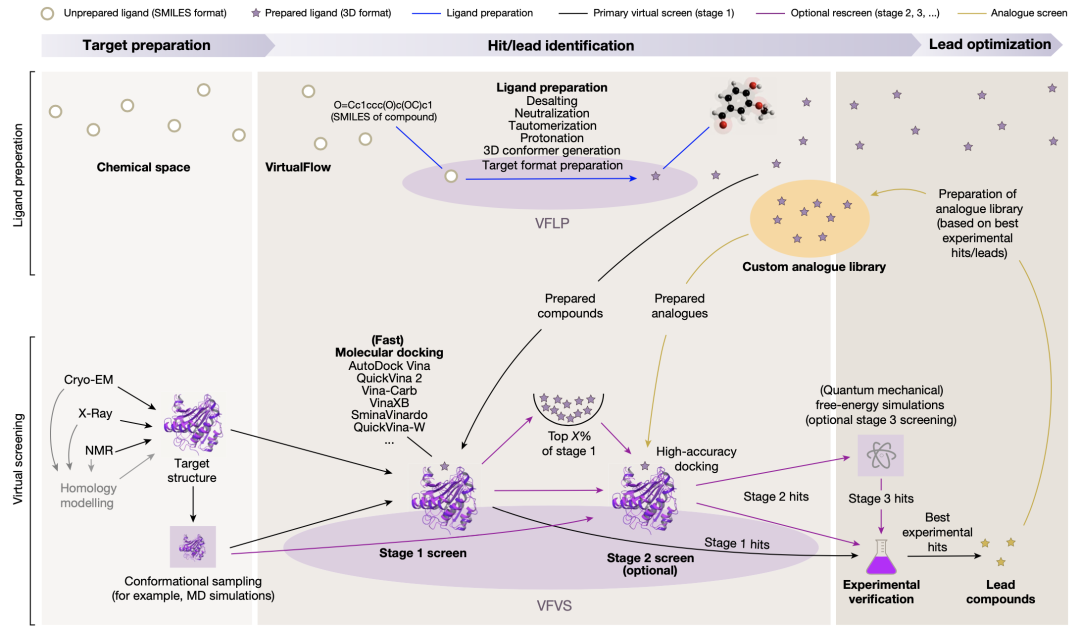
VirtualFlow在药物发现过程中的应用
图片来源 Nature
VFLP 模块
VFLP通过将配体数据库从SMILES格式转换为任何所需的目标格式(例如,基于AutoDock的对接程序所需的PDBQT格式)。VFLP使用ChemAxon的JChem软件包以及Open Babel来将处理配体分子,可以将配体分子进行脱盐处理,中和处理,生成互变异构体,计算特定pH状态下的配体质子化状态,计算其三维坐标并将分子转换成所需的格式。
市售化合物是化学领域中最令人感兴趣的空间,因为这些化合物很容易购买。目前可用的最大供应商库之一是Enamine的REAL库,其中包含约14亿个按需定制化合物。我们使用VFLP将REAL库中约14亿种化合物转换为PDBQT格式,并使该库可在VirtualFlow主页上免费使用,可通过图形界面进行访问。整个数据库具有六维属性,每个维对应于化合物的物理化学性质(分子量,分配系数,氢键供体,受体的数量,可旋转键的数量以及拓扑极性表面积)。
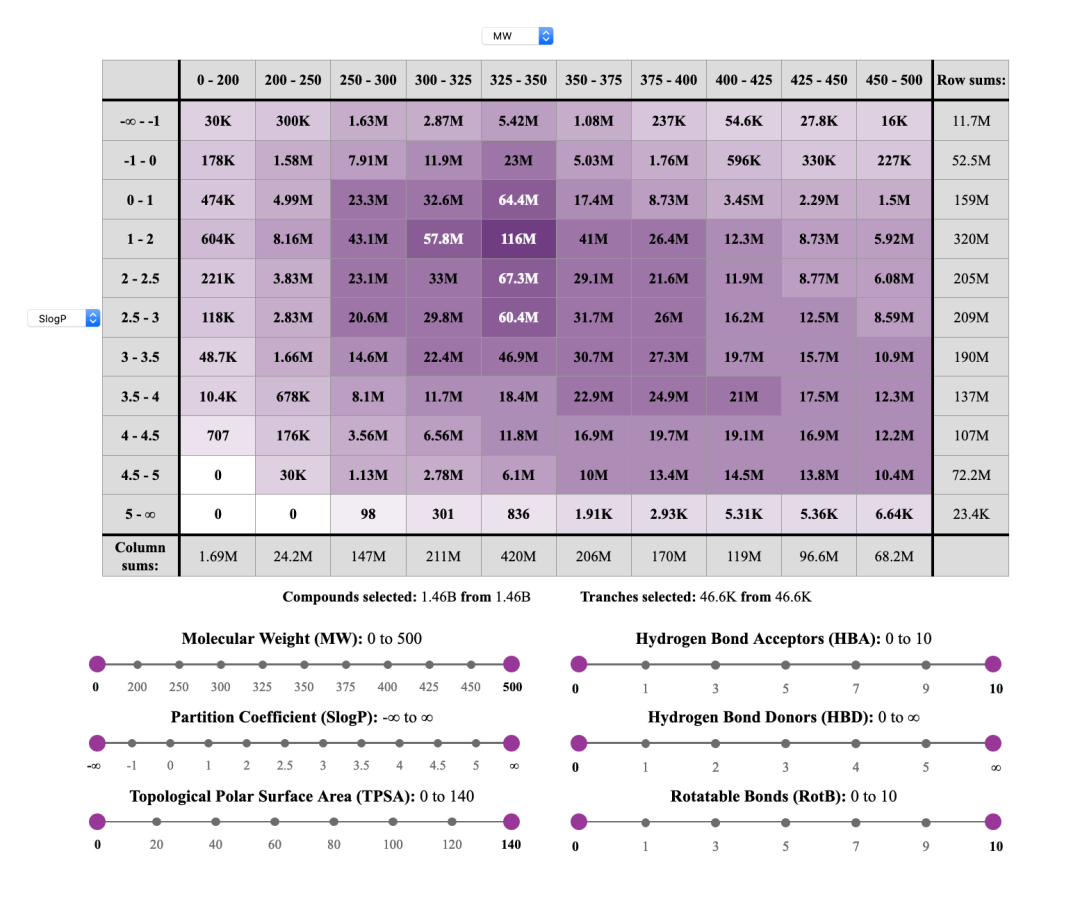
VFVS模块
使用VFVS进行虚拟筛选,用户需要指定一组对接方案。通过选择对接程序,受体结构和对接参数(包括受体上的预定义对接表面,受体上柔性残基)的严格性来定义对接方案对接例程。当前,VirtualFlow支持以下对接程序:AutoDock Vina,QuickVina ,Smina,AutoDockFR,QuickVina-W,VinaXB和Vina-Carb。由于VFVS支持各种不同的对接程序,可以重逢利用每个程序的独特优势。VFVS可以对每个配体执行多种对接方案,从而实现共对接以及集成对接。VirtualFlow还可以集成其他对接程序。VFVS还可以用于进行多阶段的虚拟筛选,以用于提高筛选的质量。在多阶段方案中,连续执行几个虚拟筛选。从前一个阶段前进到下一个阶段中的化合物数量将依次减少。随之而来的是对接精度和计算成本的增加。
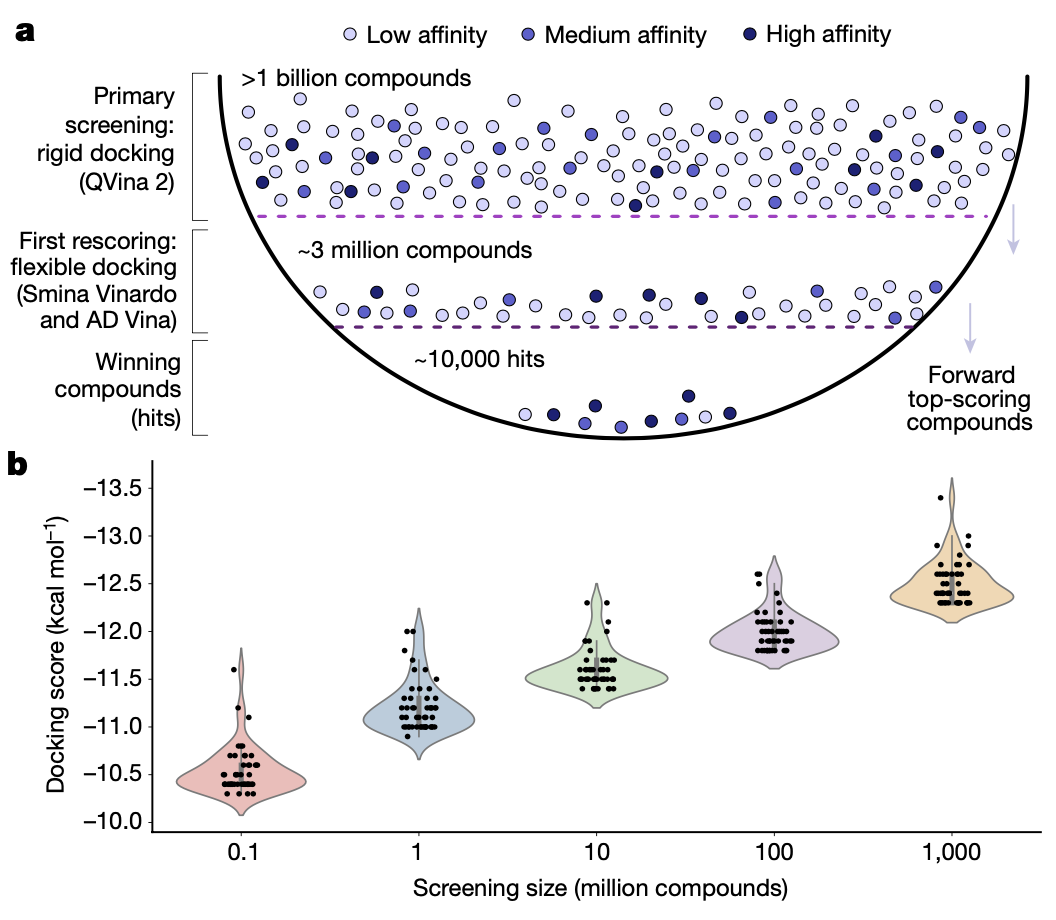
多阶段虚拟筛选的示意以及超大规模虚拟筛选的优势
图片来源 Nature
案例:VFVS 从13亿个分子中虚拟筛选
为了验证VFVS的性能,本文针对KEAP1靶点筛选了13亿种市售化合物的虚拟文库(ZINC 15数据库中约3.3亿种化合物11和Enamine REAL库中的约10亿种化合物)。这两个库之间存在一些重叠的化合物。这项工作使用异构Linux群集上的8,000个核,大约在4周内完成。
为了阐明超量规模虚拟筛选的优势,本文从REAL库的约10亿个化合物筛选中随机选择了不同规模的配体子集(0.1、1、10和1亿个化合物)。随着虚拟筛选规模的增加,对接后的平均得分也在增加,从而提高了筛选出具有更高结合亲和力的分子的几率。这会导致更高的真实命中率和实验结合亲和力,这是由本文计算出的概率模型所预测所得,并且已经通过实验证明了这一点。
为了演示多阶段虚拟筛选,本文将初次筛选中获得的排名前300万化合物进行rescore。在第2阶段,允许KEAP1与NRF2相互作用界面的的13个残基具有柔性。这种柔性设置可以解释氨基酸在蛋白结合界面上的动力学。在rescoring过程中,本文使用了两个不同的对接程序(Smina Vinardo和AutoDock Vina),并进行了两次重复,以进一步增加对接过程中采样的构象空间。多阶段筛选取决于选定的靶标和可用的计算资源。
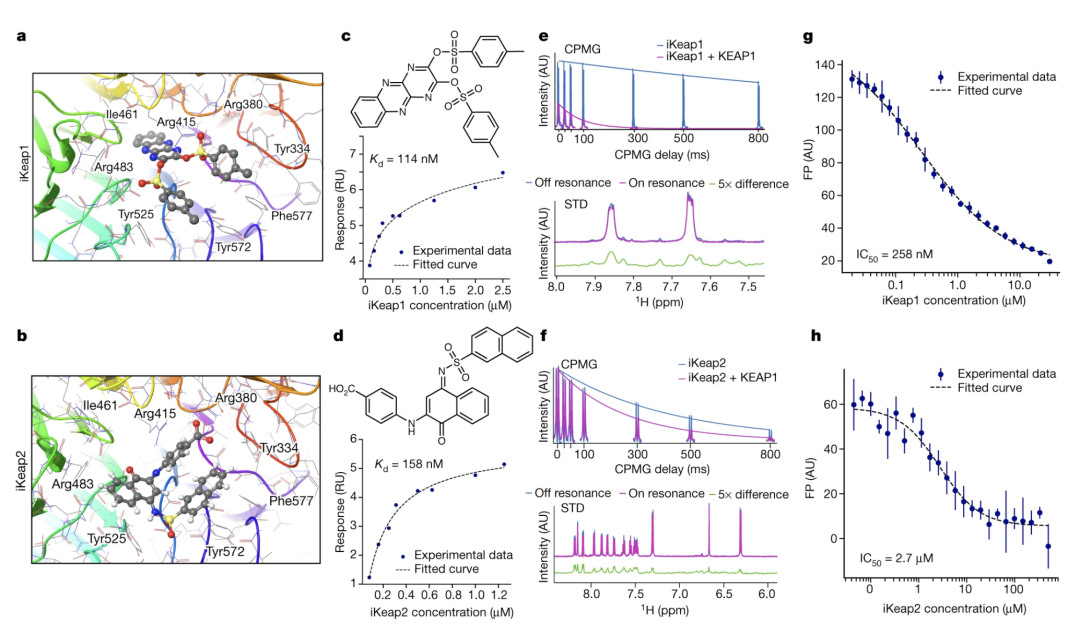
两个Hits(iKeap1和iKeap2)的对接Pose和实验验证
图片来源 Nature
手把手教您搭建virtual-flow
复现步骤
官网:https://virtual-flow.org/
1. 下载VFVS的算例
算例地址:https://virtual-flow.org/sites/virtual-flow.org/files/tutorials/VFVS_GK.tar
百度云地址:链接: https://pan.baidu.com/s/1X0Ge3pcM3K_rYN-0h9Dqyw 密码: s6q5
解压:tar -xvf VFVS_GK.tar
解压之后的文件
2. 进入input

README.md
Readme
smina_rigid_receptor1:配置文件信息
ligand-library 配体文件库
receptor 受体文件:内部为pdbqt格式
qvina02_rigid_receptor1 :配置文件信息
receptors
3. 进入tools文件
进入templates文件夹
配置文件信息都储存在all.crtl , todo.all中
todo.all中主要储存的是配体信息,按照下面的提示,等于cellections.txt中信息
如果你想要自己下载
a:下载配体文件库:
网站:https://virtual-flow.org/real-library
打开网站后,可以选择部分配体,也可以选择整个库下载,拖动图中紫色小球进行选择,本文选择的配体库如下图,点击Download,两个全部点击下载,本次选择wget
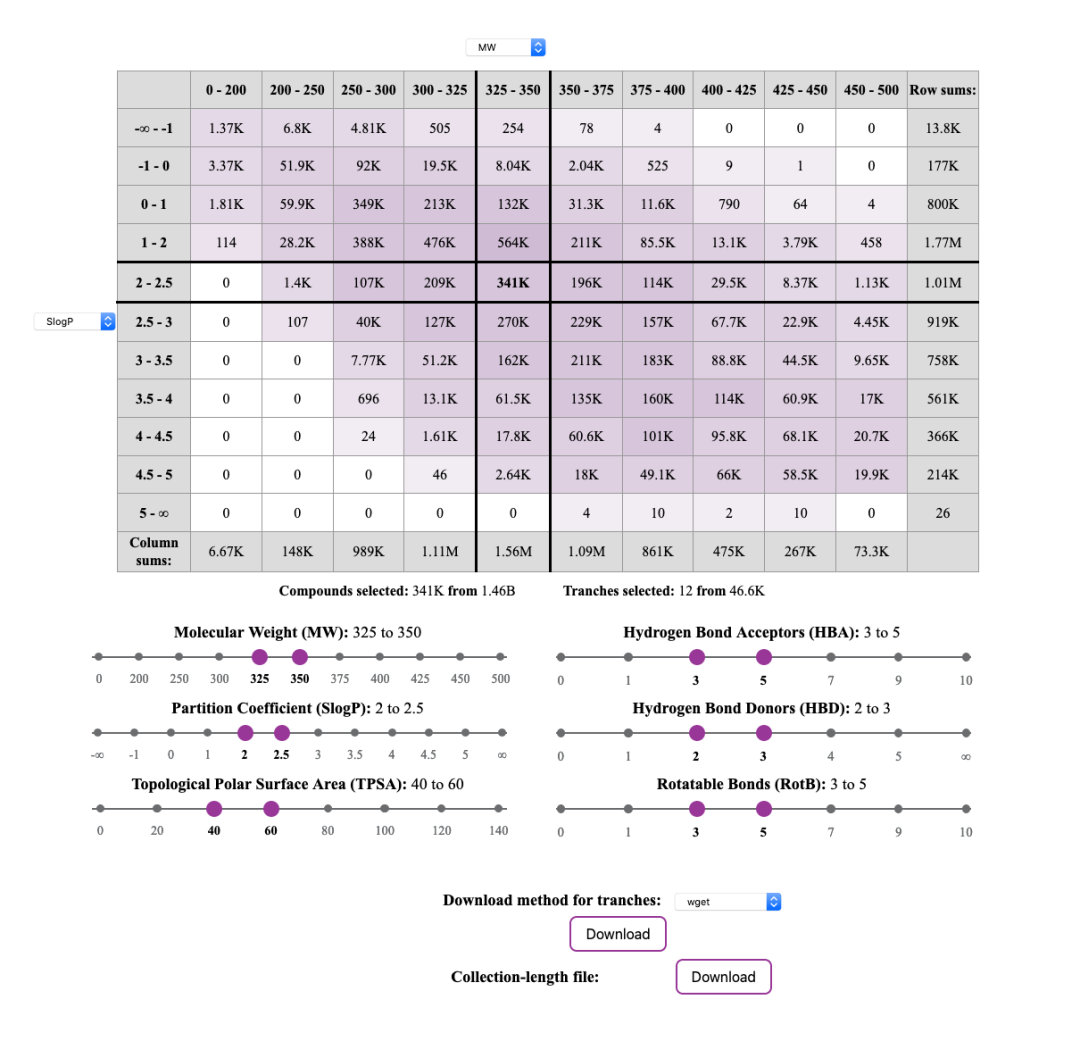
b:下载后的文件:
两个文件tranches.sh(第一个download),collections.txt(第二个download)

c:在终端下运行tranches,会下载一系列的文件
指令:sh tranches.sh/source tranches.sh
配体文件
collections.txt
信息:
4. all.crtl中修改配置信息
其中信息配置信息很多
这里挑几个重点讲:
文件中有标注,我这里就直接按照思维导图的方式标记出来了

备注:选择集群调度指令,对接软件可以使用多个中间要以‘:’号分割
5. 运行全部设置好了之后运行
进入tools文件夹
运行:./vf_start_jobline.sh 1 12 templates/template1.slurm.sh submit 1
其中12代表着12个节点
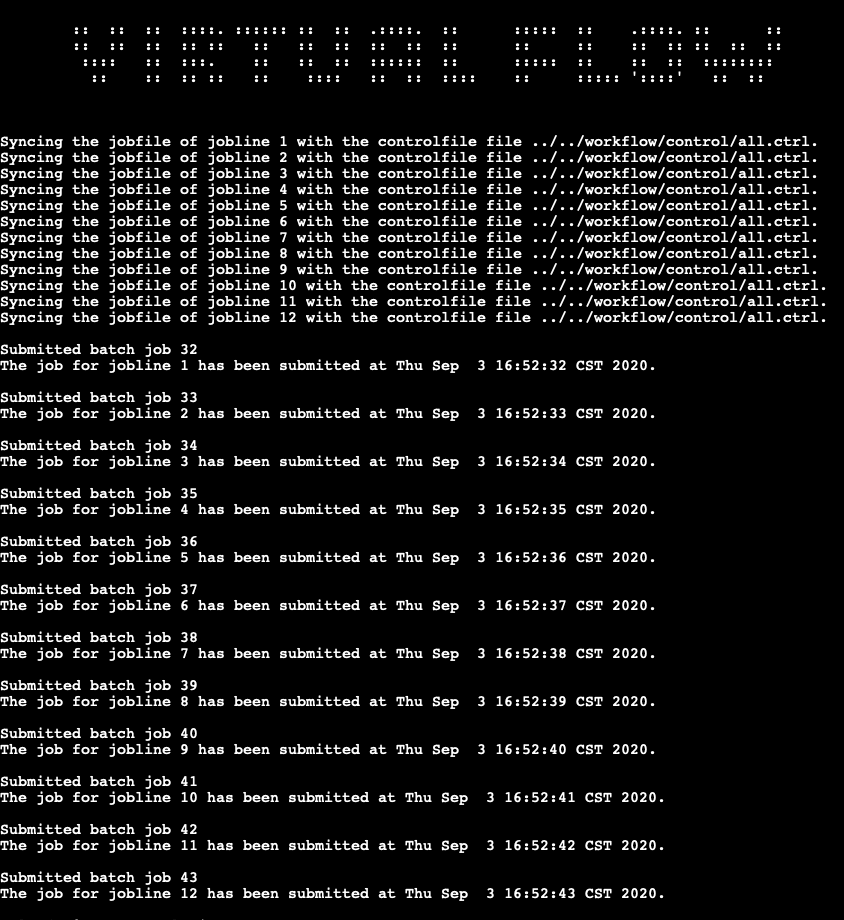
6. 运行完成之后可以使用以下指令查看前10化合物
./vf_report.sh -c vs -d qvina02_rigid_receptor1 -n 10
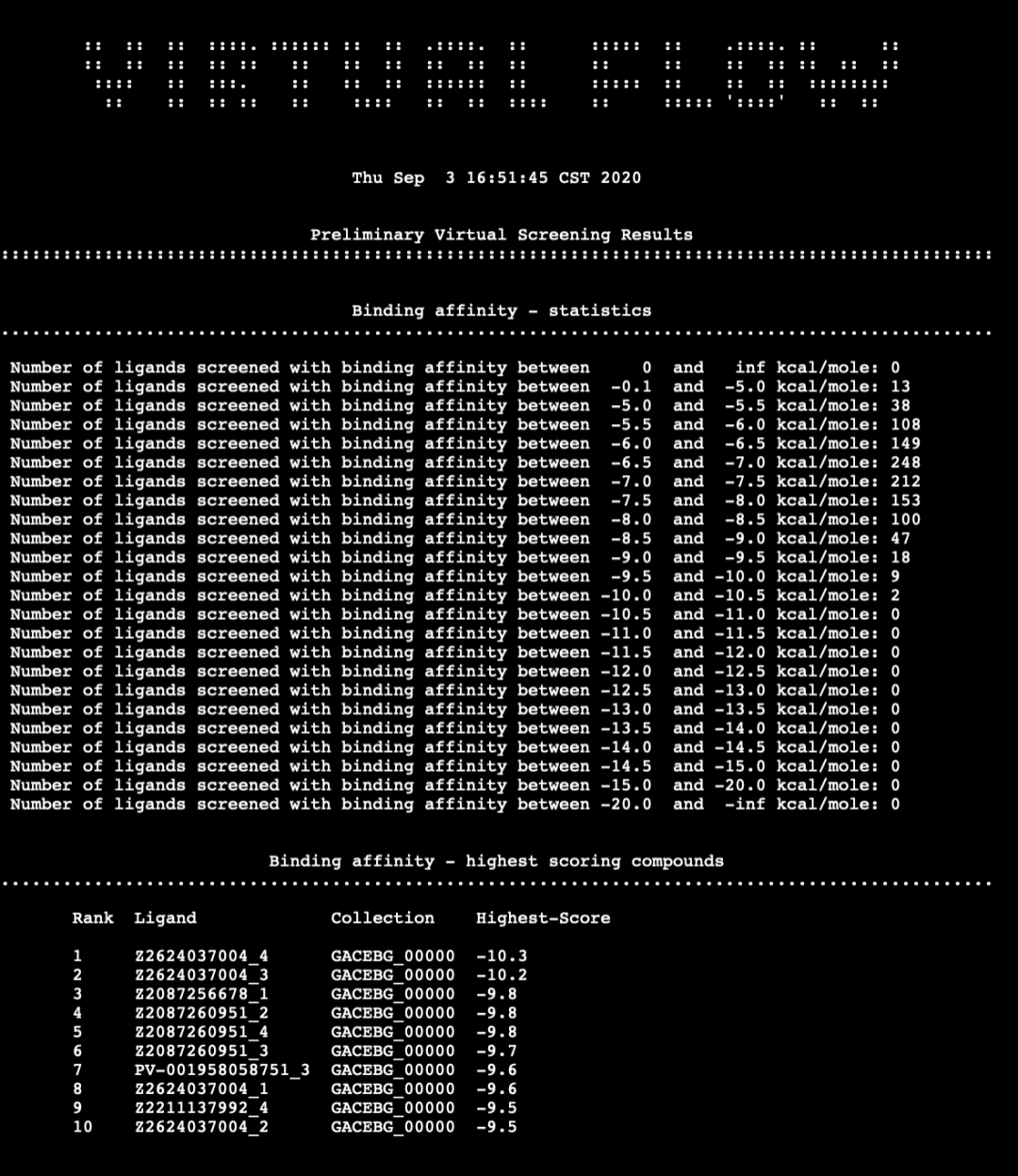
7. 运行完成之后进行的分析
7.1从github上下载VFtools
下载:wget https://github.com/VirtualFlow/VFTools/archive/master.tar.gz
解压:tar -xvzf master.tar.gz
重命名:mv VFTools-master VFTools
VFTools中的文件:

将bin文件夹加入到环境路径中:
命令:export PATH=”Path/to/VFTools/bin:$PATH”
注:这只是一个临时变量,尽可能修改bashrc文件,并加入上面的命令
7.2安装openbabel
提供两种安装方式
a:使用conda
conda install openbabel -c conda-forge
b:直接从原网下载并进行安装
http://openbabel.org/wiki/Main_Page
7.3 首先将筛选完成的化合物进行排名
cd <VFVS root directory>
#进入VFVS目录
mkdir -p pp/ranking
#新建pp/ranking
cd pp/ranking
#进入目录
vfvs_pp_ranking_all.sh ../../output-files/complete/ 2 meta_collection
#排序
输出文件/文件夹:
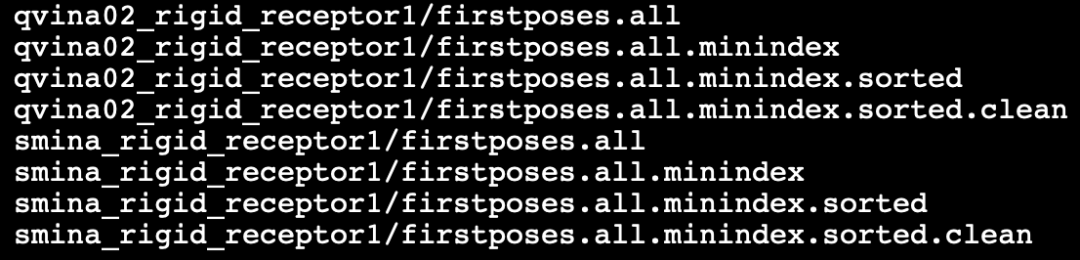
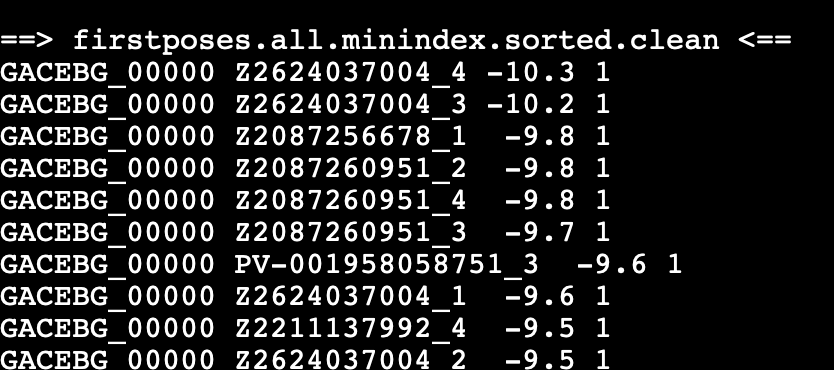
直接查看clean为后缀的文件:
head -10 *.clean
7.4 提取排名前列的化合物pose
在pp文件夹中新建docking_poses文件夹
mkdir -p docking_poses/qvina02_rigid_receptor1
cd docking_poses/qvina02_rigid_receptor1
查看前100化合物,重定向为compounds
head -n 100 ../../ranking/qvina02_rigid_receptor1/firstposes.all.minindex.sorted.clean > compounds
然后,提取前100个化合物的结构
vfvs_pp_prepare_dockingposes.sh ../../../output-files/complete/qvina02_rigid_receptor1/results/ meta_collection compounds dockingsposes overwrite
docking_poses中的文件以及文件夹:
直接进入dockingsposes.plain文件夹中
cd dockingsposes.plain
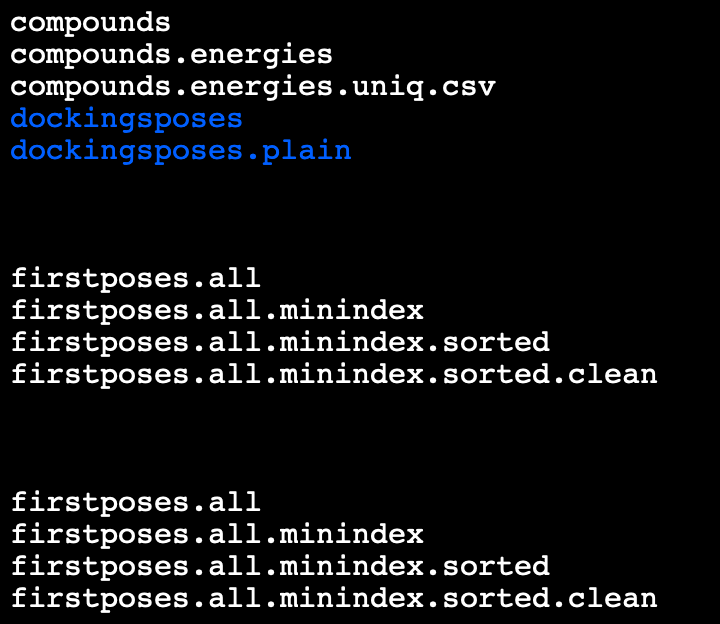
文件夹内容里面就是每个ligand排名第一的pose:
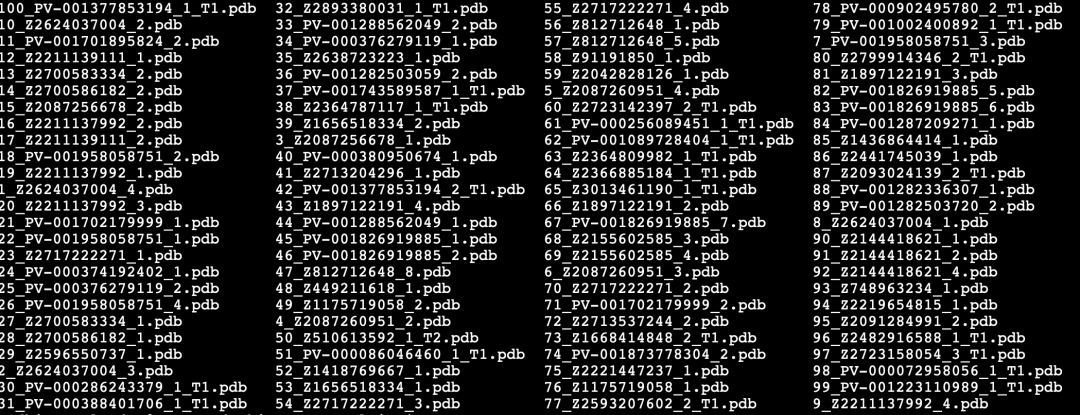
Pymol打开:
将pdb文件作为文本文件打开:
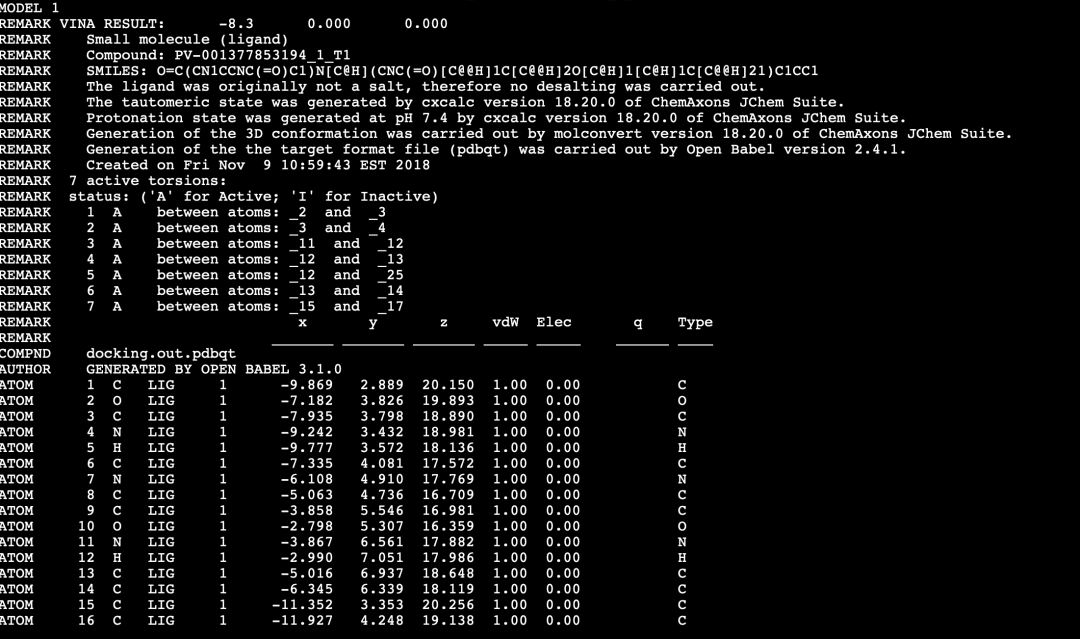
pdbqt格式,排名第一的model,对接得分为-8.3.有smiles格式以及一些标注,后续还有坐标格式
参考文献
Gorgulla, C., Boeszoermenyi, A., Wang, Z. et al. An open-source drug discovery platform enables ultra-large virtual screens. Nature 580, 663–668 (2020).