引言
今天给大家分享期刊Journal of Chemical Information and Modeling (JCIM)和Journal of Medicinal Chemistry (JMC)在药物设计和发现方面的一些前沿动态,供大家学习参考。以后我们也会在每周五定时推出各类期刊在生物计算方面的前言动态,敬请大家期待。
JCIM | 基于深度学习的共享隐式“指纹”配体设计
主要内容
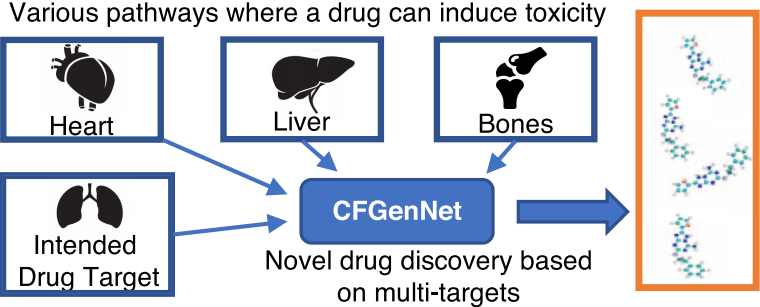
配体筛选在药物发现中一直有着举足轻重的作用。Raghuram等人于2021年5月14日在JCIM上发表了名为Deep Learning-Based Ligand Design Using Shared Latent Implicit Fingerprints from Collaborative Filtering的最新成果,提出了一种对配体进行筛选从而发现新药的方法,称为隐式指纹(Implicit Fingerprints)技术。
该研究通过隐式指纹技术对蛋白质进行表征来设计新的化合物,从而编码药物的性质。此外该方法在其设计新药物的能力,以及产生的新化合物的生物活性可靠度方面均得到了很好的验证。该方法将协作过滤模型和深度学习模型相结合对共享数据库中的配体和蛋白质进行筛选。该研究方法可在生成有用的新型药物化合物的基础上,进一步用于新药的发现。
评论
该研究方法与以往方法相比具有较高的精度,是一种很有前途的用于新药发现的方法。但该方法依赖训练数据集,因此在用于其他领域时需要对该方法的一致性进行评估。此外,该方法也存在一定的局限性,会产生一些错误或不合理的结构,从而影响筛选的准确率。
参考文献
Raghuram Srinivas, Niraj Verma, Elfi Kraka, Deep Learning-Based Ligand Design Using Shared Latent Implicit Fingerprints from Collaborative Filtering, Journal of Chemical Information and Modeling, 2021, ASAP. DOI:10.1021/acs.jcim.0c01355.
原文链接
http://dx.doi.org/10.1021/acs.jcim.0c01355
JMC | 从大量的毒性数据中筛选毒性信息
主要内容
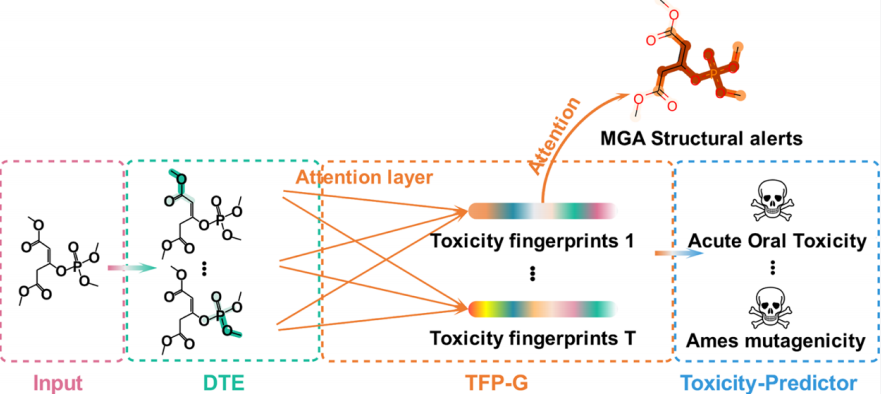
安全性一直是药物成功应用面临的一个主要问题,因此药物毒性靶点的准确预测同样极具挑战。2021年5月14日Wu等人在JCIM上发表了名为Mining Toxicity Information from Large Amounts of Toxicity Data的研究成果。该研究提出了新的MGA框架模型来预测药物化学毒性并揭示了子结构与化学毒性之间的关系。研究表明MGA方法在33个毒性数据集上均显示出了良好的预测能力,并能够提取毒性特征生成定制的毒性样本。此外,MGA还提供了一种检测结构警报和发现不同毒性机制之间关系的新方法。将非常有利于从大量的毒性数据中挖掘出不同的毒性信息。
评论
该研究提出的MGA框架,基于不同的毒性数据集通过对毒性基团的回归和分类显示出了良好的毒性靶点预测能力,该研究将有助于解决药物的安全性问题,有利于新药的研发及应用。
参考文献
Zhenxing Wu, Dejun Jiang, Jike Wang, Mining Toxicity Information from Large Amounts of Toxicity Data, Journal of Medicinal Chemistry, 2021, ASAP. DOI:10.1021/acs.jmedchem.1c00421.
原文链接
http://dx.doi.org/10.1021/acs.jmedchem.1c00421
JCIM | DPNet方法预测药物−通路
并用于药物再利用

主要内容
药物−通路关联的鉴定在药物再利用中起着重要作用。计算方法相比实验方法可以高效且经济的进行大规模的药物−通路相关性预测。2021年5月14日,Wang等人于JCIM上发表了名为Pathway-Based Drug Repurposing with DPNetinfer: A Method to Predict Drug−Pathway Associations via Network-Based Approaches的研究成果,开发了DPNet计算方法,以预测潜在药物−通路的相关性。DPNet: http://lmmd.ecust.edu.cn/netinfer/。
该研究开发的DPNet推断法具有效率高和低成本的特点,在预测药物-通路方面具有良好的表现。该方法不仅用于预测药物的目标蛋白,还可以通过选择预测类型对药物−途径进行预测。并通过该方法成功从以往另类药物中筛选出了用于癌症治疗的新药。总而言之,DPNet方法为大规模预测药物−途径相关性提供了一个强有力的工具。
评论
DPNet推断方法在较高预测准确度的同时也存在一些问题。如蛋白质活性位点无法直接确定,也无法预测药物的激活或抑制类型。在该研究组的下一步研究中,将对这些问题进行解决。该研究方法将有助于药物的发现和研发。
参考文献
Jiye Wang, Zengrui Wu, Yayuan Peng, Pathway-Based Drug Repurposing with DPNetinfer: A Method to Predict Drug–Pathway Associations via Network-Based Approaches, Journal of Chemical Information and Modeling, 2021, ASAP. Doi:10.1021/acs.jcim.1c00009.
原文链接
http://dx.doi.org/10.1021/acs.jcim.1c00009
JCIM | 采用机器学习基于化学结构
预测分子性质或者材料性质
主要内容
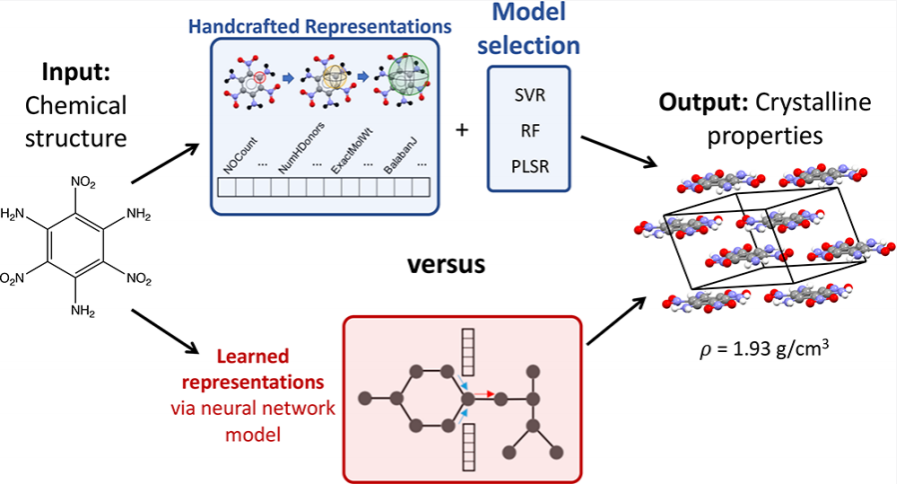
为了加速新的分子化合物的发展,化学界长期寻求的目标是仅从化学结构便可预测分子的性质。2021年4月26日,美国Awrence-Livermore研究组在JCIM上发表了名为Predicting Energetics Materials’ Crystalline Density from Chemical Structure by Machine Learning的研究成果,应用MPNN机器学习方法基于化合物结构对分子性质进行预测,加速了新分子化合物的开发。
该研究证明了在无任何晶体结构信息或量子力学信息的情况下,通过机器学习方法可以直接学习分子的化学结构从而预测分子的整体晶体性质,即使在具有挑战性的高密度分子上也能很好的预测,如TATB等。此外还通过多种机器学习模型预测了分子的晶体密度并阐述了该方法的性能及优缺点。该研究表明,基于消息传递神经网络的模型MPNN在预测结构和晶体密度方面表现良好,此外提供了基于MPNN的模型与具有特定物理意义的模型间的分析比较,解释了机器学习模型准确预测分子性质的原因。
评论
该研究的MPNN模型,首次再无任何晶体结构或量子力学信息的情况下,通过机器学习方法直接建立了化学结构与分子性质间的关联,根据晶体分子材料性质直接预测晶体结构。总而言之,MPNN模型可快速对候选对象进行筛选,并可用于加速新分子材料的设计、加快新分子化合物的开发。
参考文献
Phan Nguyen, Donald Loveland, Joanne T. Kim, Predicting Energetics Materials’ Crystalline Density from Chemical Structure by Machine Learning. Journal of Chemical Information and Modeling, 2021, ASAP. DOI:10.1021/acs.jcim.0c01318.
原文链接
http://dx.doi.org/10.1021/acs.jcim.0c01318
JMC | 一种改进配体设计和合理化SAR的策略
主要内容
水分子与蛋白质−配体的相互作用在药物发现方面举足轻重,但如何将这些相互作用转化为分子设计概念人们尚不清楚。2021年4月16日Tobias等人在JCIM上发表了名为Mapping Water Thermodynamics on Drug Candidates via Molecular Building Blocks: a Strategy to Improve Ligand Design and Rationalize SAR的研究成果,该研究可对设计具有特定的溶剂热力学性质的药物分子提供帮助。
该研究引入了一种基于高精度实验数据训练的计算方法,将从基于GIST的溶剂泛函中获得的溶剂化热力学特性分解为具有化学意义的BBs的单个贡献,通过计算每一个BBs中的溶剂化能量和熵分数,分析了溶剂相关亲和力的贡献。该研究成果将有助于凝聚态参数的设计并优化候选药物。
评论
分解成BBs是一种使用具有物理意义的模型获得溶剂化性质的有效手段。在该研究组的下一步计划中,将用合理的结构和热力学数据对该方法进行训练,并基于此方法对实验结果进行评估,以发掘该方法的全部潜力,同时也需要对不同的靶蛋白进行进一步的测试。
参考文献
Tobias Hufner-Wulsdorf, Gerhard Klebe, Mapping Water Thermodynamics on Drug Candidates via Molecular Building Blocks: a Strategy to Improve Ligand Design and Rationalize SAR, Journal of Medicinal Chemistry, 2021, ASAP. DOI:10.1021/acs.jmedchem.0c02115.
原文链接
https://pubs.acs.org/doi/10.1021/acs.jmedchem.0c02115