背景介绍
不断增长的公开或私有数据集为药物发现提供了重要的数据支撑,而机器学习算法如支持向量机(SVM)或深度神经网络(DNN)在巨大规模的数据集上运算成本很高。相较于经典计算机(CC),量子计算机(QC)计算速度很快,但是有很大的局限性。在化学信息学领域中,要克服的挑战之一就是需要将高维的分子描述符压缩降维以用于QC。本文提出了几种可行的压缩方法并结合SVM和Data Reuploading Classifier(DRC,类DNN算法)两种分类算法在QC上进行了测试,为建立药物发现领域的量子计算方法奠定基础。
数据集
1. SARS-CoV-2:132个分子,阳性阈值为6.65 uM;
2. 结核分枝杆菌:18886个分子,阳性阈值为100 nM、1 mM或10mM;
3. Cathepsin B:63331个分子,阳性阈值为20%;
4. Krabbe病:44809个分子,阳性标签与原作者一致;
5. Plague(Yersinia pestis):139861个分子,阳性阈值50%;
6. hERG:306587个分子,阳性阈值50%;
7. 结核分枝杆菌2:293937个分子,阳性阈值为MIC_50 < 10 ug/mL 或 IC_50 < 10uM 同时选择性SI > 10。
量子计算机
IBM ibmq_rochester,共有53个量子比特(如图1所示)。
描述符压缩
SARS-CoV-2数据使用的描述符为2048位的ECFP6,维度太高无法用于QC。作者提出以下四种压缩方法:
1. 主成分分析 (PCA);
2. 线性判别分析 (LDA);
3. 将2048位等分为x组,每组则有k位,将每一组看作一个二进制数并转为十进制(如图2所示);
4. 使用一种算法记录描述符中所有1和0的位置。
算法实现
量子SVM使用Qiskit库中的LS-SVM实现。SVM depth为默认值2,表示电路将重复2次;entanglement为“full”,表示所有量子位均相互纠缠;skip_qobj_validation为False,表示不输出警告;shots为2048。DRC使用2个量子比特模仿一个两层的有非线性特征的神经网络,使用描述符为MFF,为71735位向量。
主要结果
首先作者使用SARS-CoV-2数据集作为测试描述符压缩方法的一个示例数据集。通过上述4种方法将2048位描述符降至2或3位后使用QC进行SVM建模。结果如下:
方法1:CC:37%,QC:33% (N=3);
方法2:CC:40%,QC:39% (N=3);
方法3:CC:61%,QC:59.6% (N=3);
方法4:CC:59%,QC:59.25% (N=2);
之后,作者对四种方法的不同组合也进行了测试,结果如表1所示。其中结果最好的为方法1(PCA)+方法3。作者分析结果,认为之所以表现较好的原因可能是这样得到的数据离散度较大(如图3所示)。
另外,作者还测试了混合方法(QC执行部分计算,CC 执行剩余部分)以消除对 QC 的存储限制。作者使用 SVM 的方法 1 和方法 3 将数据点减少到6个维度。通过执行简单的幺正运算U(x1, x2, x3)将数据加载到单个量子位中,其中x1、x2 和x3是点的坐标。当维度大于三时,可以有U(x1, x2, x3, x4, x5, x6)到(U(x1, x2, x3), U( x4, x5, x6))。我们有一个类似的U(θ1, θ2, θ3)用于旋转bloch球体中的数据点,以制作一个双量子位连接层。对于相同的SARS-CoV-2数据集,准确率为61%。
之后,作者在结核分枝杆菌数据集上进一步测试了DRC方法。在表2 中,我们看到在 QC 上获得的准确度更接近在 CC 上获得的准确度,与 CC 相比具有轻微的时间优势。
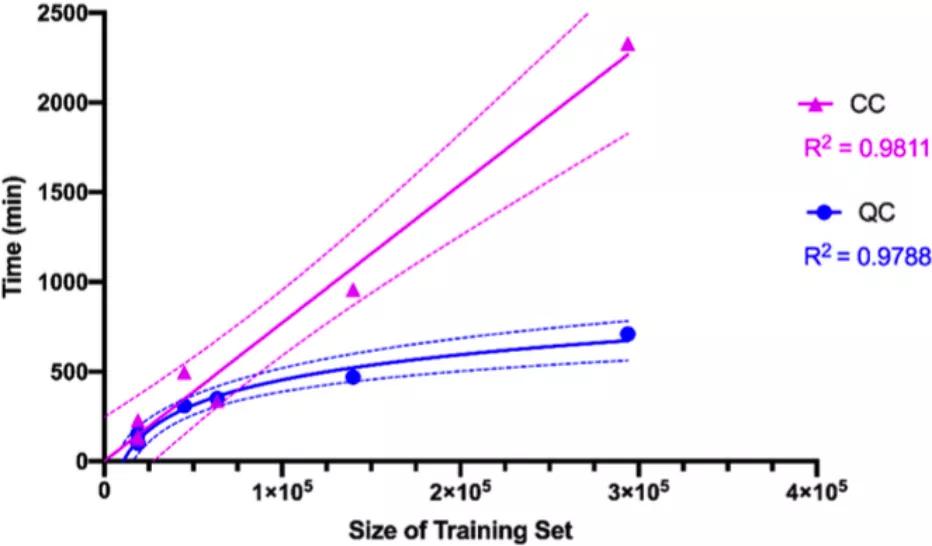
最后,作者在其他几种大规模的数据集上也应用了量子机器学习方法,数据集大小范围从44000~293000个分子(如表3所示)。作者发现传输数据是 QC 上运行此类大型数据集的主要时间开销。不同数据集大小的模型在 CC 上的计算时间明显是线性的,但对于QC 而言,时间趋于稳定(如图4所示)。
图表汇总
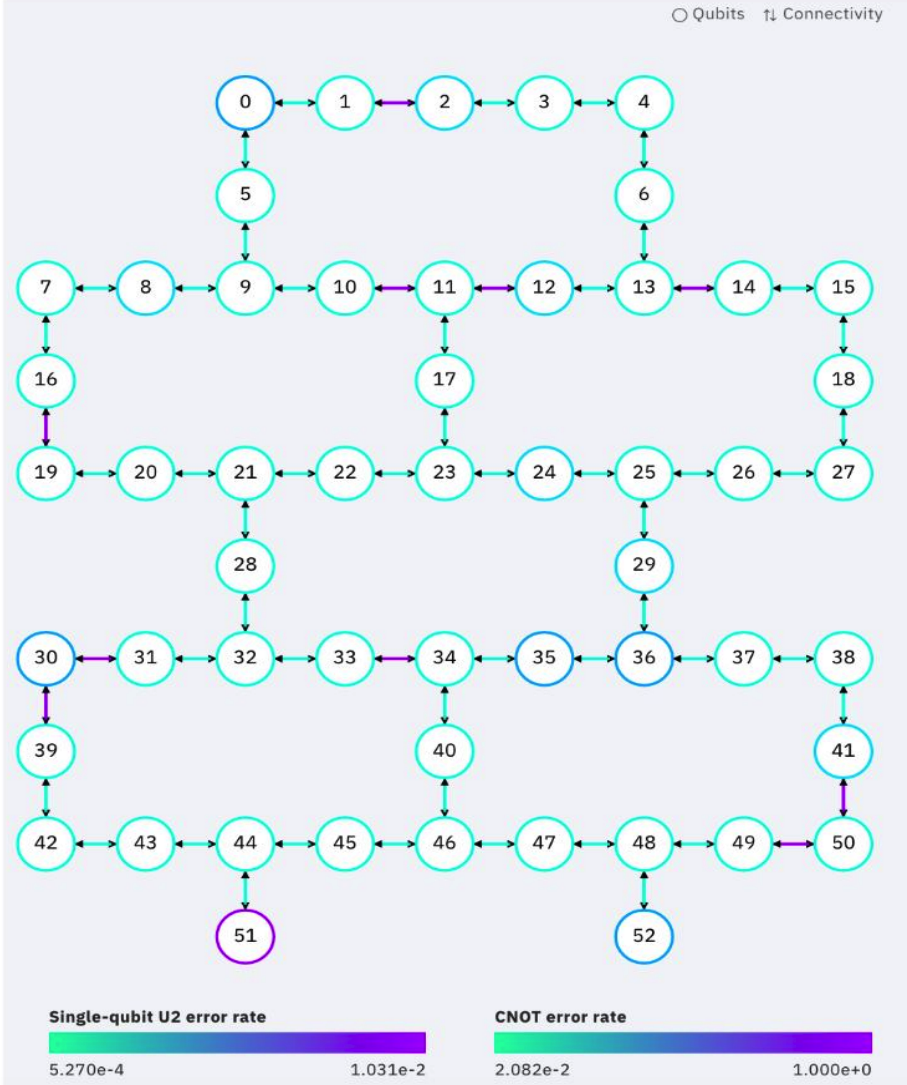
图1. IBM ibmq_rochester架构
图片来源于JCIM
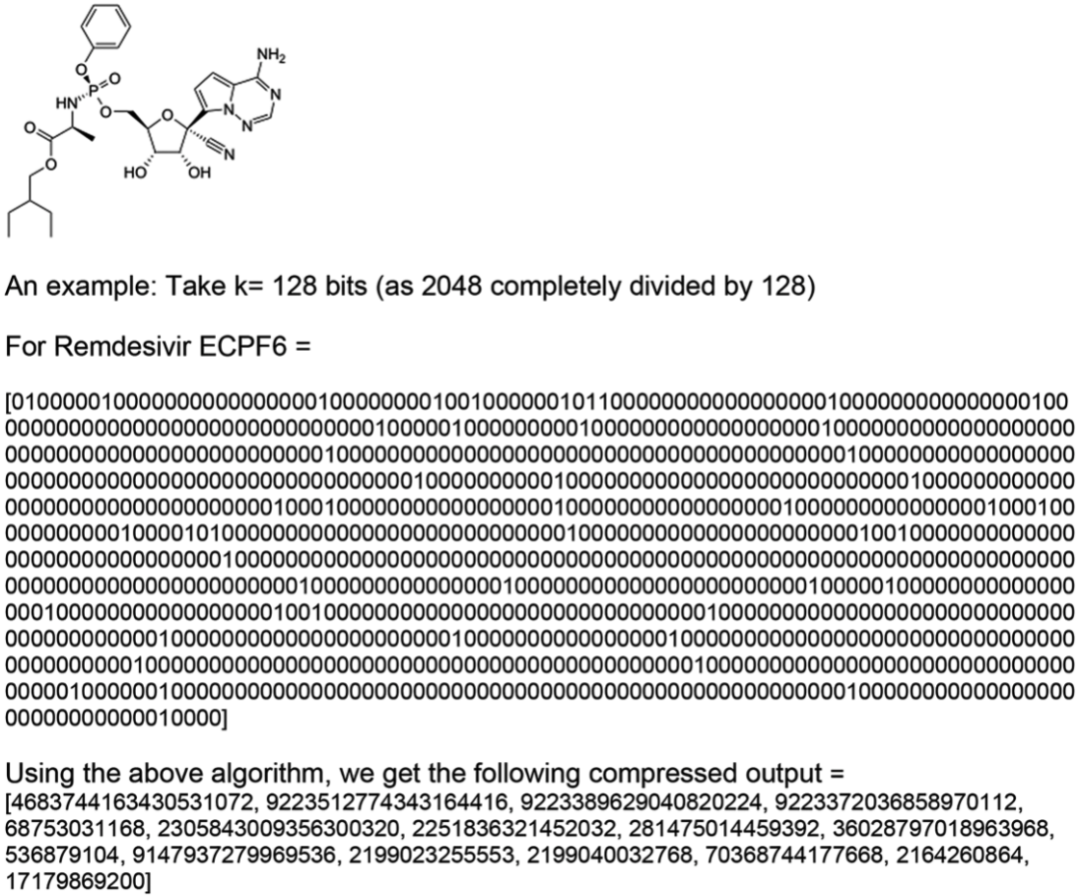
图2. 一种压缩方法图示
图片来源于JCIM
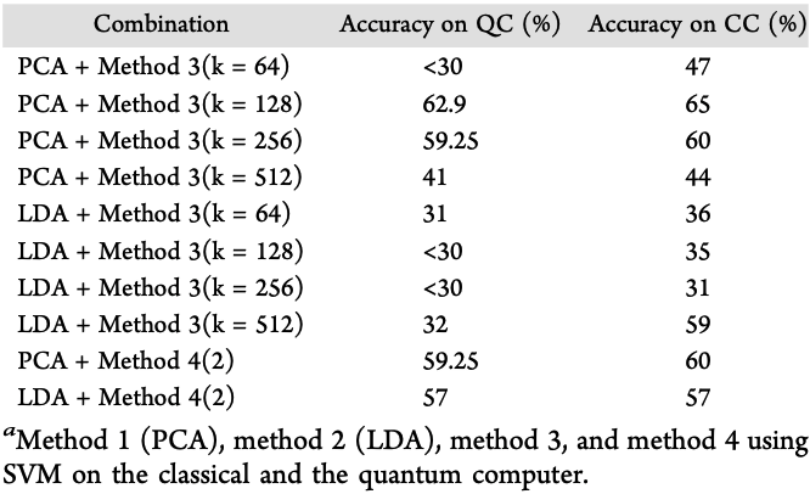
表1. 组合方法的预测结果
表格来源于JCIM
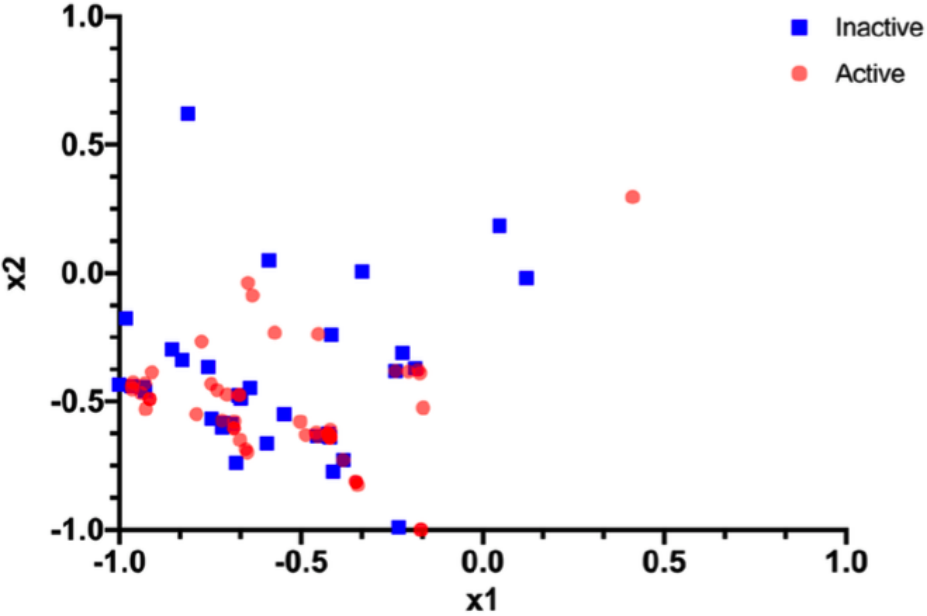
图3. SARS-Cov-2的数据分布
图片来源于JCIM
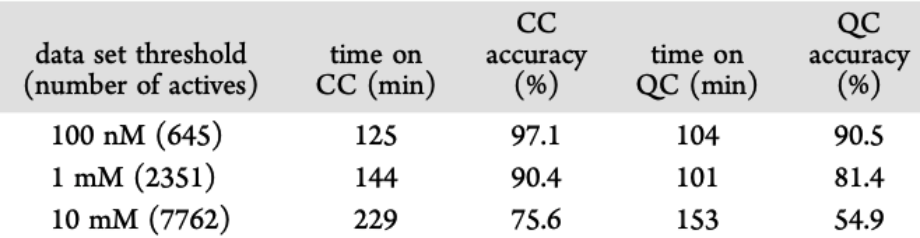
表2. 结核分枝杆菌抑制数据集(18,886种化合物)的准确性和运行时间结果比较
表格来源于JCIM
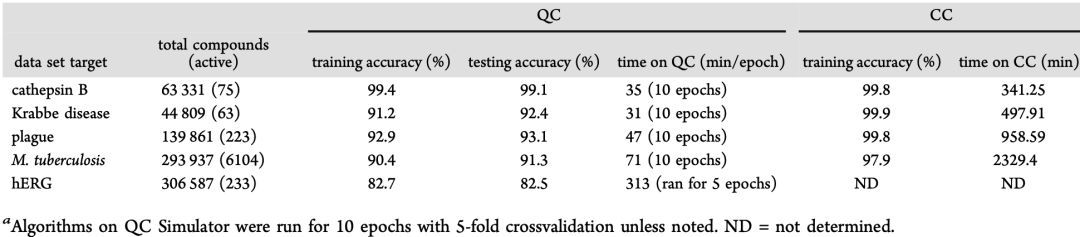
表3. 对比大规模数据集在CC和QC上的表现
表格来源于JCIM
图4. 比较量子计算机 (QC) 模拟器和经典计算机 (CC) 的数据集大小与运行时间
图片来源于JCIM
亮点总结
作者讨论了四种压缩分子描述符的方法及其组合,在QC上的机器学习模型进行了测试。作者发现将方法3和方法1结合使用时可以得到最佳结果。此外,作者同时应用了QC和混合方法来训练模型,对当前药物发现来说混合方法可能是最佳选择。当处理更大量级的结构-活性数据集时,作者发现计算机和基于云的QC之间的数据通信开销远大于在QC电路上执行所需的实际时间,证明QC可以处理数十万个分子数量级的“非常大”的药物发现数据集。本研究展示了在具有多个不同大小的独立数据集的QC上计算时间的非线性增长,远优于在CC上观察到的线性增长。目前在公共数据库(如PubChem)中积累的针对其他靶点和疾病的更大的高通量筛选数据集对SVM和深度神经网络以及其他计算密集型工具提出了重大挑战,而QC是克服其中一些限制并使计算成为可能的一种可行方法。随着量子机器学习的发展,QC在药物发现化学信息学应用中的可及性将会增加,如DNA编码化合物库量级可达百万级。未来评估这些和其他量子机器学习模型的研究还需要涉及前瞻性预测和实验验证,以便为其药物发现价值提供令人信服的证据。
参考文献
Kushal Batra, Kimberley M. Zorn, Daniel H. Foil, Eni Minerali, Victor O. Gawriljuk, Thomas R. Lane, and Sean Ekins, Quantum Machine Learning Algorithms for Drug Discovery Applications, Journal of Chemical Information and Modeling, 2021, 61, 6, 2641-2647. DOI: 10.1021/acs.jcim.1c00166.