
引言
结合热点区域通常结合低分子量化合物(片段),其重要性在FBLD的相关文献中已被很好地证明。结合热点大多通过实验手段发现,如NMR-SAR方法和基于共晶的MSCS方法都可以确定蛋白表面上的重点结合区域,但实验手段工作量巨大且失败率高,基于计算的片段映射方法可靠性稍逊实验但代价极小,其中GRID和MCSS过于关注局部能量最小值而产生过多假阳性片段,FTMap通过使用较大的分子来探测表面解决了此问题。测试及验证计算方法需要标准或验证数据集,本文构建了一个测试发现结合热点方法的基准数据集Acpharis,并与Astex公司的基准数据集一起完成对FTMap方法的测试。
Acpharis 数据集的产生
第一步从PDB数据库中查询分子量在80-200g/mol之间的“片段”型配体,剩下的分子通过子结构检索找到PDB库中更大的配体,以下分子被排除:
i作为片段结合到同个蛋白上;
ii以相似的朝向或位置结合;
iii明显大于片段(至少一个配体的分子量>250g/mol)。
最终得到包含62个片段-蛋白对的基准数据集,有48个唯一的蛋白和52和唯一配体。
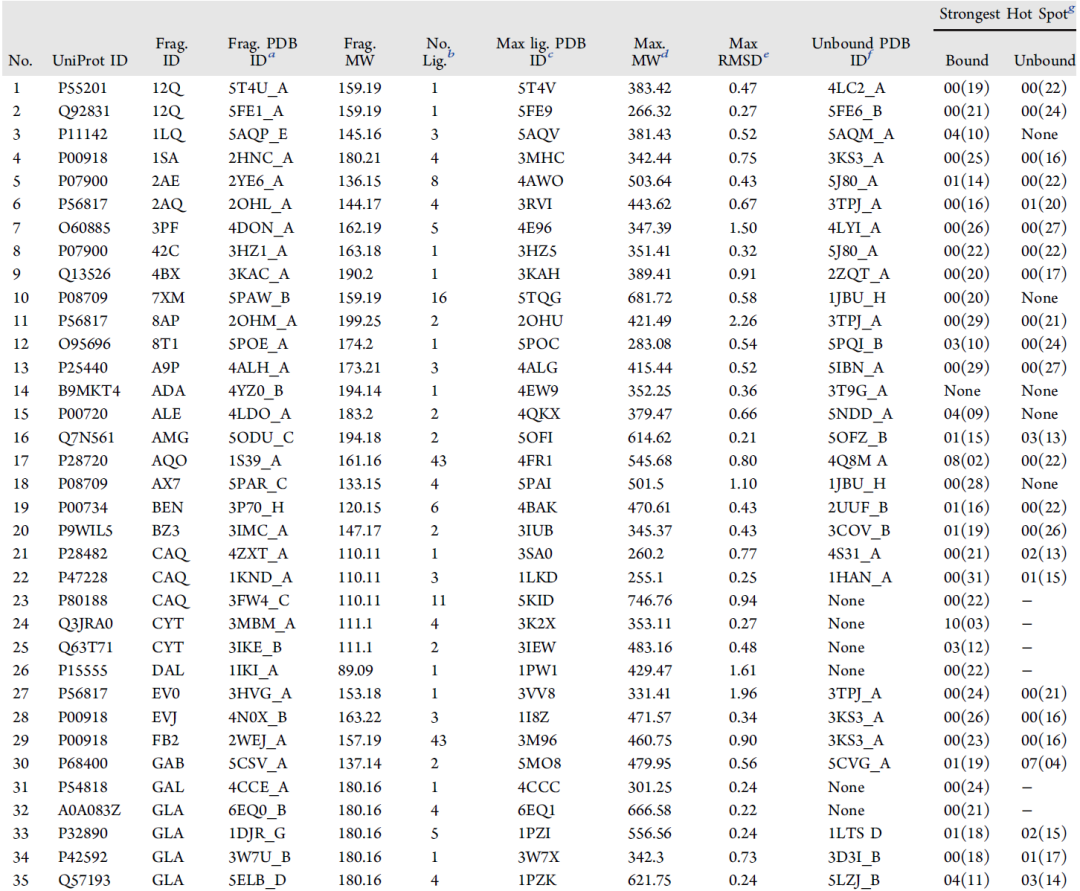
图1. 部分Acpharis数据集.
图片来源:JCIM
结合配体结构的FTMap分析
基于片段结合的口袋包含至少一个重要的共有位点的共识,FTMap几乎能在所有情况下检测到此类口袋。运用FTMap对图1第四列的数据进行分析返回5行结果,以人源碳酸酐酶II(PDB code2HNC)结合配体1SA为例,图2(A)a.第一行表示FTMap给出的从强(00)到弱(06)的共有位点,括号内的数字表示每个位点处的探针簇的数量,大于13时表示位点能够结合具有毫摩尔及以上亲和力的分子,16及以上的探针簇则能预测和具有微摩尔级别化合物的结合。第二行表示每个热点覆盖片段的百分比,图2 (B)a.青色(00)表示100%覆盖片段1SA的原子,白色(04)表示部分覆盖,第三行表示的关系则相反,表示有多少比例的热点被片段占据。后两行是片段生长为配体时的热点分析,人源碳酸酐酶结构中最大的配体是ARZ,1SA是其子结构,第四行表示每个热点覆盖最大配体的百分比,图2 (B)b.白色(04)和橙色(06)覆盖率为68%和14%,第五行表示配体占据每个热点的百分比,ARZ的10%占据了88%的04。
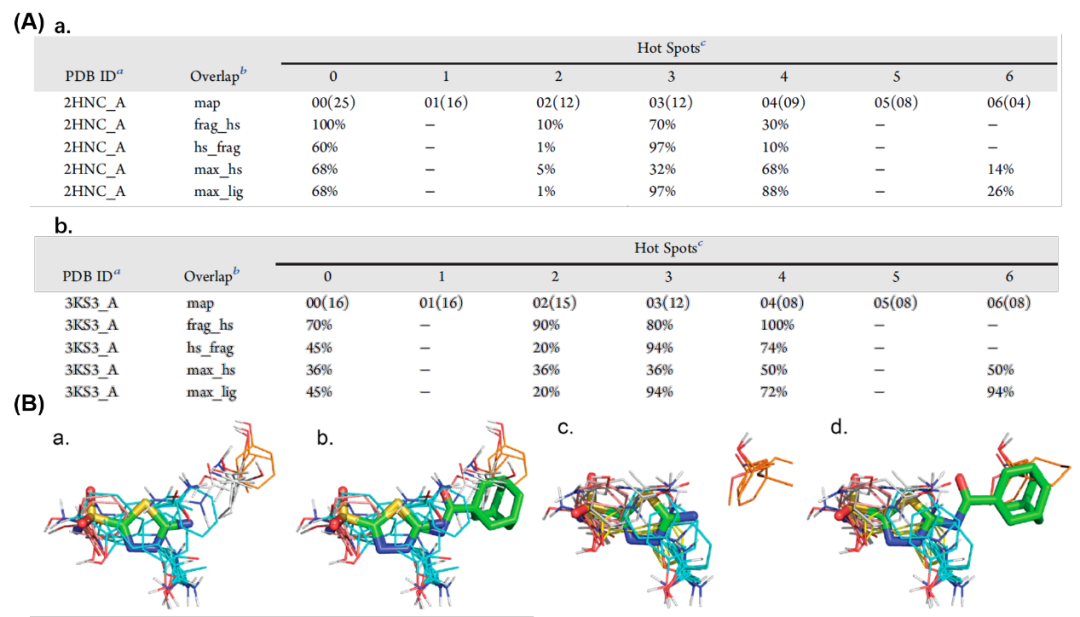
图2. 人源碳酸酐酶II的热点分析.
图片来源:JCIM
未结合配体结构的热点分析
大多数情况下,热点区域分析的目的是找到没有已知配体信息的蛋白上的配体结合位点。因此,Acpharis数据集还新增了这部分未结合配体的信息,共有44种蛋白表示了48个蛋白-片段对。结合位点与结合配体的结构做了叠合,人源碳酸酐酶II的热点分析显示,最强热点探针簇00(16)比结合配体状态时少,图2 (B)c.显示热点00覆盖了1SA70%的片段,片段却只占据45%的热点,00仅覆盖最大配体ARZ的36%,图2 (B)c.显示配体(橙色)也与热点06反应。
Astex和Acpharis数据集的比较
Astex公司的Verdonk及同事构建了一个相似的蛋白数据集,他们的主要目的是区分一个区域是热点还是温点,而不是和作者一样为了测试验证热点的方法。该数据集包含52个结合热点和温点的样本。作者为了知道是否任何一个强热点能覆盖片段结合位点,设立了热点覆盖片段的50%和80%两个阈值,探针簇13和16的热点被认为具有成药可能,这两种标准均被认为是成功找到的正确热点。结果显示,任何热点中,Acpharis和Astex表现相似。考虑最强热点00时,Acpharis中,有结合结构的成功率比没有结合配体的结构高,Astex则相反。这可能是后者中更大的配体已与蛋白共结晶,结合这样的配体可能会使区域远离结合片段的口袋即热点。
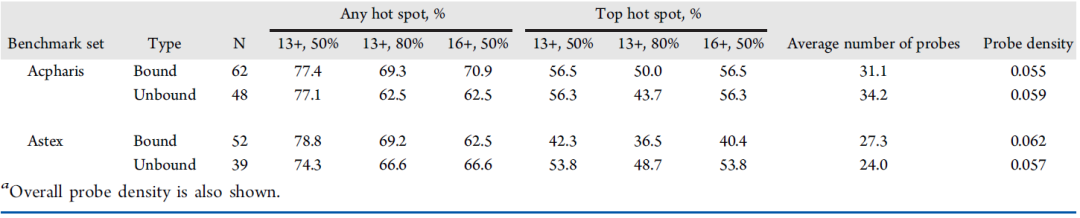
图3. Acpharis和Astex数据集覆盖热点的能力对比.
图片来源:JCIM
总结
本文创建了包含62种高质量蛋白-片段复合物的数据集Acpharis并用其测试了热点鉴定方法FTMap。另外,还讨论了另一个区分热点和温点的数据集Astex。尽管数据集的构造方法不同,但不考虑最强热点时,两者表现十分相似。
参考文献
Wakefield AE, Yueh C, Beglov D, et al. Benchmark Sets for Binding Hot Spot Identification in Fragment-Based Ligand Discovery. J Chem Inf Model 2020. https://doi.org/10.1021/acs.jcim. 0c00877