
引言
与高通量筛选相比,基于片段的药物设计使用的化合物库要小得多,但如此小的库仍能有效地探索巨大的化学空间,而且片段往往具有较低的靶标亲和力和较高的配体效率和更好的理化特性。除实验技术外,FBDD常采用分子对接技术来确定单个分子与蛋白的潜在结合模式或者用于片段库的虚拟筛选。目前只有少数小分子对接工具涉及到片段对接且相关数据早已过时或不可得。德国Björn Windshügel和Laura Chachulski构建了LEADS系列的第二个基准数据集LEADS-FRAG作为LEAD-PEP的补充,且评价了4款对接工具再现晶体结合模式的能力和打分性能。
基准数据集LEADS-FRAG的产生
晶体结构选择使用KNIME分析平台完成,第一步按以下条件从PDB数据库中进行查询:
i不含DNA,RNA或DNA/RNA杂合;
ii有实验数据;
iii结构最多包含两条链;
iv Rfree<0.3;
v分辨率<2Å;
vi 配体的分子量在100~300Da。
共查询到11639条代表唯一的蛋白质的数据。接着无偏差地除去仅包含缓冲分子或配体不合适的条目,该步骤使用ELD的过滤结构来实现,过滤之后仅剩1832个PDB,随后去掉配体缺失/共价/明显非特异性的条目后使用CD-HIT根据序列相似性做聚类分析得到162个聚类,基于共结晶配体的结构使用MOE的tanimoto得到101个聚类,最终剩下93个高质量的蛋白-碎片复合物。大多数碎片满足RO3可旋转键数≤3且PSA≤60Å2,平均分子量187.2Da,重原子数7~22,clogP在-4.1~3.5之间。所有分子描述符(logP, MW, SASA)都使用MOE计算得到且为避免干扰自对接,给所有配体重新生成坐标。蛋白准备使用MOE的Protonate3D方法完成。
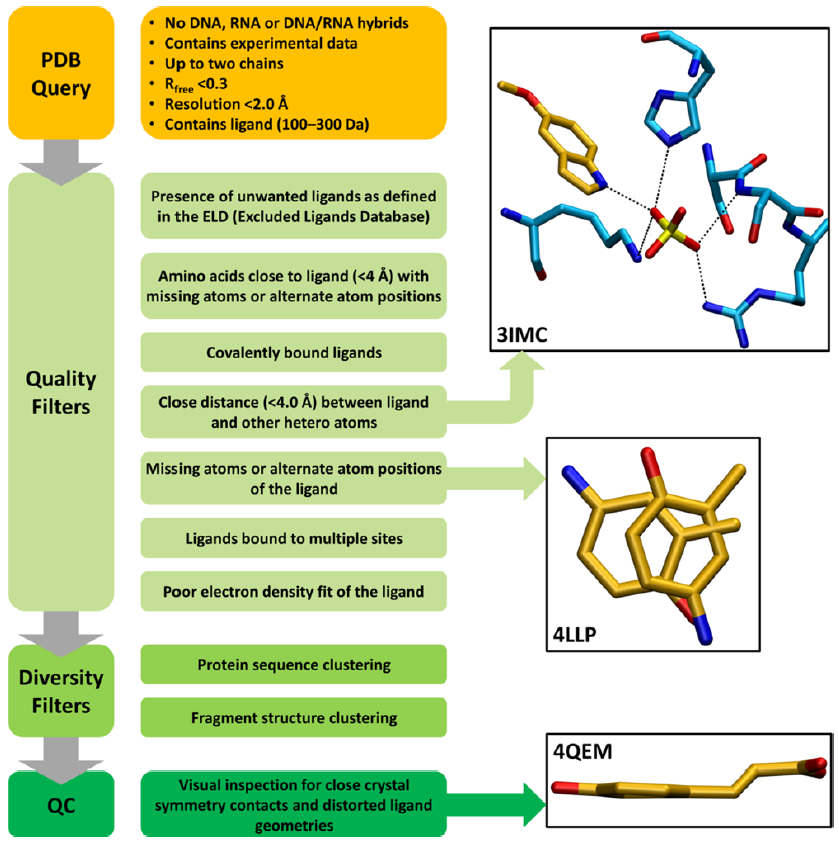
图1. LEAD-FRAG的生成.
图片来源:JCIM
片段对接性能评价
待评测的工具有AutoDock, AutoDock Vina, LeadIT中的FlexX和GOLD。对AutoDock,和AutoDock Vina的SA和HA两种不同的精度进行了测试,对GOLD的四个打分函数(ASP, ChemPLP,ChemScore和GoldScore)和FlexX的四个打分函数(FlexX-Score,ChemScore,PLP和ScreenScore)都单独进行了测试。每次对接最多产生30个构象,选择实验结合模式与对接结合模式的均方根偏差RMSD来衡量性能。
就CPU对接耗时而言,AutoDock Vina SA表现出最快的对接速度(平均5.2s),其他均在10s以上,AutoDock对接最为耗时,平均为252.5s(SA)和2490.5s(HA)。当仅考虑最佳得分构象与晶体构象的RMSD时,GOLD(ChemPLP)是最准确的(1.0 Å ),AutoDockHA和FlexX的所有四个打分函数的测评结果RMSD均2.7 Å。当考虑30个构象中RMSD最小的构象时,平均RMSD与最佳得分构象相比有明显降低,GOLD:CP表现最佳(0.6 Å ),紧随其后的是GOLD:ASP, GOLD:CS和 GOLD:GS。当从对接工具准确复现结合模式(RMSD Å)的数目而言, GOLD(ChemPLP)表现最佳,其次是AutoDock Vina SA和GOLD:GS。最佳得分构象条件下复现率从33.3%(FlexX:CS)到53.8%(GOLD:CP),最小RMSD构象条件下复现率下从52.7%(FlexX:FS和FlexX:SS)到86.0%(AutoDock VinaSA)不等。
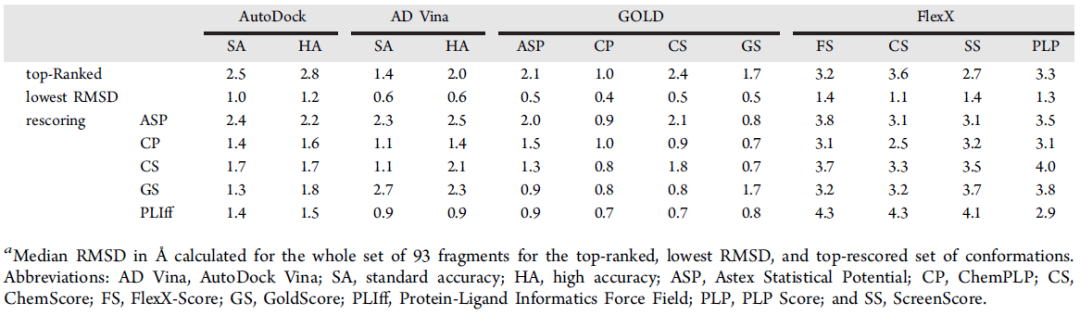
图2. 不同对接工具和打分函数的总体均值RMSD情况.
图片来源:JCIM
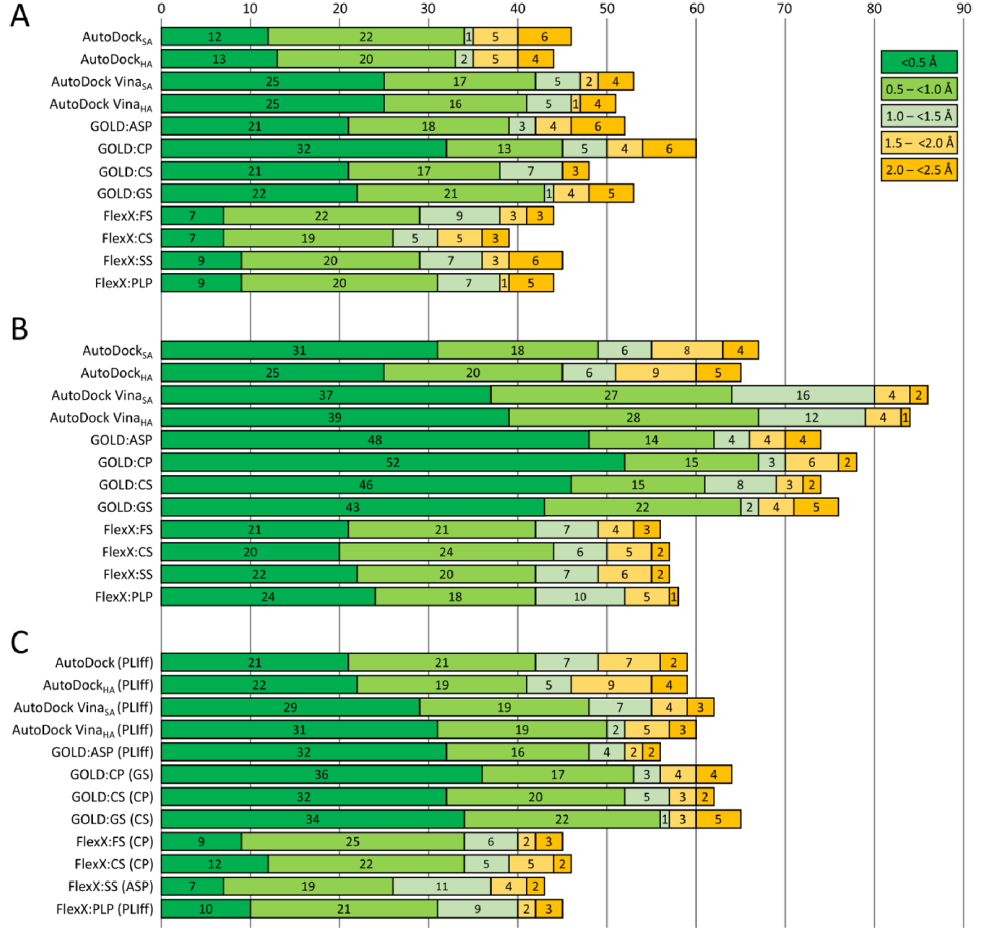
图3. 不同工具复现天然结合模式的能力.
图片来源:JCIM
最佳得分构象与最小RMSD构象之间的差异表明需要对所有对接构象优化以获得相对于最佳得分构象而言整体的对接性能提升,使用GOLD的四个打分函数及一个外部打分函数Pliff重打分,ChemPLP对FlexX:CS产生的构象重打分时,Pliff将平均RMSD降低了,近天然配体结合模式的构象数量提高到40, 其他评分均对RMSD有所增加。
总结
本文使用合理且无偏差的程序KNIME创建了包含93种高质量蛋白-片段复合物的数据集LEADS-FRAG。另外,在此基准数据集上评估了4款对接工具。GOLD的ChemPLP打分函数在测评中表现最佳,AutoDockVina不同准确度的表现几乎一样,AutoDock和FlexX的片段对接性能较差。
参考文献
Chachulski L, Windshugel B. LEADS-FRAG: A Benchmark Data Set for Assessment of Fragment Docking Performance. J Chem Inf Model 2020. DOI: 10.1021/acs.jcim. 0c00693