
引言
蛋白质疗法的聚集稳定性一直是个巨大挑战,例如用于糖尿病治疗的胰岛素在酸性pH值、温度升高或搅拌下会表现出聚集现象,导致生产、储存、纯化和输液过程中的复杂性。先前研究发现胰岛素类似物的低聚集稳定性可能与胰岛素单体形式参与聚集的早期阶段有关,特别是折叠-展开途径上的部分折叠中间体(PFI)。合理设计聚集抑制剂的一套方法包括开发破坏纤维的分子,例如设计肽或非肽β-片段类似物,以及筛选破坏芳香堆积相互作用的分子。最近,一种基于结构的计算机辅助设计,成功地扩展了筛选方法,从大量设计肽中识别新的抑制剂。本文设计早期聚集抑制剂的方法是基于破坏易聚集的胰岛素二聚体。蛋白质-蛋白质相互作用(PPI)已被证明通常由少量残基介导,这就提供了用小肽取代大蛋白以模拟这些“热点”残基所产生的相互作用的可能性。
在本研究中,作者将N−PFI胰岛素同源二聚体中天然单体(N)的结合界面残基作为模板,设计对胰岛素易聚集的PFI具有高特异性的肽。确定了一个最稳定的N−PFI同源聚体的集合,然后根据界面上埋置的残基面积确定结合界面的热点片段。首先根据与天然单体起始结构域的构象相似性,从这些天然单体的界面片段中筛选出肽。第二次筛选是通过选择与源结构域相同的PFI残基结合的肽,即模拟N-PFI界面上的相互作用。用尺寸排阻色谱法和远紫外圆二色谱法分别评价所选肽对胰岛素聚集和变性的抑制作用。通过等温滴定量热法评价了所设计肽的特异性及其胶束化热力学。最后,用酶标仪分光光度法定量测定肽对HepG2细胞葡萄糖摄取的影响。
先睹为快
作者信息
印度理工学院化学工程系的Anurag S. Rathore和Gaurav Goel教授
蛋白靶点
胰岛素的部分折叠中间产物(PFIs)
计算方法
蛋白-蛋白对接,分子动力学
计算软件
HEX:用于生成N−PFI复合物的对接构象
PEPFOLD:用于生成每个肽的低能构象
GROMACS:用于分子动力学模拟
计算流程
基于先前对胰岛素蛋白的模拟,筛选出14个PFIs结构,用于对接胰岛素天然单体N,确定可能参与形成N−PFI复合物的关键残基; 接下来,将N−PFI复合物的结合界面残基作为模板,设计对PFI具有高特异性的肽,并利用累积结合界面面积ΔAsegmentN- PFI对片段进行排序; 经过进一步的筛选和修饰,选出三个9肽分子进行下一步功能验证,并通过MD模拟证明了其与PFI可以稳定结合; 结合实验结果,最终获得了两个可有效地抑制胰岛素聚集的九肽分子:(R6)P3 (EAAYL AGLY)、
(R6)P19(AAYLVAGLY)。
研究内容
作者先前研究了在淀粉样蛋白形成条件下(PH 2,330K)测定了胰岛素折叠结构上的五个亚稳态集合,一个最低自由能自然状态,一个未折叠状态,以及三个亚稳态中间产物。这些中间产物与胰岛素聚集有关,相对于未折叠状态具有更高的天然接触,以下称为部分折叠中间产物(PFIs)。每个PFI集合由一组动态的结构组成,吸引子(attractor)是系综中最低的自由能结构。从三个PFIs集合中获得了32 个PFI结构用于本文中的分析。使用gromos算法对这些PFI进行分组,得到14个簇PFI1-PFI14,Cα均方根偏差(RMSD)截止值为0.3 nm。基于Cα接触图(距离截止,0.95nm)和残基聚集倾向(A3D评分),进一步评估14个PFI构象的结构相似性, 显示这些PFI构象非常明显(支持信息中表S1)。表现出完全的二级结构损失,并且比原生系综具有更高的溶剂可及表面积SASA(支持信息中表S2)。图1a和b分别显示了最稳定溶液结构(自然系综吸引子)和一个PFI中易聚集残基的表面分布。图1c显示了14个PFIs中每个残基的平均(μ)A3D评分和相关标准差(σ)。正如预期的那样,PFIs中更大比例的残基具有正的A3D评分,并且与原生状态相比,总体数量也更高,说明具有大的正A3D值的残基与蛋白质聚集有关。此外,PFI具有由空间连接的易于聚集的残基形成的疏水斑块,因此有望表现出更高的聚集倾向。作者还注意到,多个残基具有大的相关标准差,表明PFIs的结合界面存在显著的不均匀性。然而,差异主要在于A3D评分的大小而不是符号。同一组22个残基在14个PFIs中有11个的A3D评分为正,而同一组16个残基的A3D评分在14个PFIs中有12个为负(支持信息中图S2)。此外,不同的PFI在运动上彼此可接近,因此,14个PFI可被视为一个单一的系综来测定缔合热点残基。
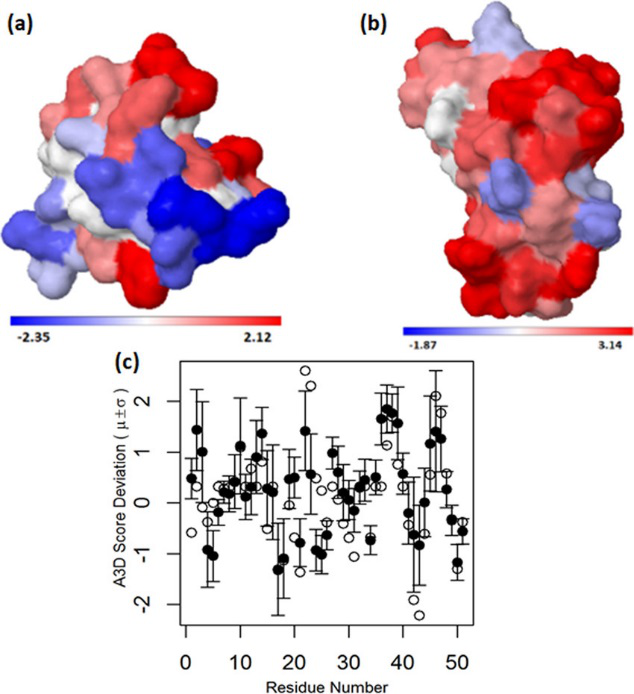
图1. 结构不同的PFI构象研究
图片来源JCIM
天然单体与易聚集的中间产物的结合被认为是胰岛素聚集的早期成核步骤。如图2a中的程序示意图,将天然单体与每个PFI对接(总共28个),以确定最有可能参与形成这种N−PFI复合物的残基。通过刚性对接获得的总共14000个N−PFI结构,使用gromos聚类法将其分为4353个亚组,Cα-RMSD截止值为0.3nm。利用给定簇中结构的结合自由能计算每个子集的簇自由能分数Gc。图2c显示,对于前几个最稳定的簇,Gc有很大的增量,随后的簇的间距要小得多。最稳定的团簇(最低Gc,如图2b所示)的中心结构的稳定性通过GROMACS进行了分子动力学模拟进行评估。N和PFI单体沿垂直于结合界面的方向分离时,重组为二聚体,并且在5个独立的200ns模拟中都保持完整。图2b显示了这种最稳定的复合物中的关键稳定相互作用,结合界面由来自天然单体N(I10、C11、S12、L13、Y14、L16、F22、V23、H31、L32、E34、A35和L38)的13个残基和PFI(G1、I2、Q5、C6、C7、T8、H26、L27、C28、H31和E34)的11个残基组成。天然单体中的大部分界面残基属于A链C末端的α螺旋(R12−R20)和B链α-螺旋(R30−R40),这两个区域在低pH下参与胰岛素原纤维的形成。进一步发现N−PFI复合物,主要由四对疏水残基稳定,即L13N−I2PFI、L13N−L32PFI、F22N−L27PFI和V23N−L27PFI。作者还确定了H31N和T8PFI侧链之间和Y14N侧链与I2PFI主链之间的两个界面氢键。H31被证明在胰岛素核聚集的稳定中起关键作用,这表明在聚集过程中静电相互作用的重要性。为了获得更大的重要分子间相互作用的光谱,作者用15个最稳定的团簇的中心结构来识别N和PFI单体上的结合域。
图3a,b分别显示了15个N−PFI二聚体中天然单体和PFI的每个残基的结合界面面积的平均值和标准偏差。N和PFI单体的标准化结合界面面积(ΔAiN- P F I)具有较大标准差表明结合界面残基存在显著的不均匀性。N和PFI单体分别具有43和50个不同残基,其中至少一个二聚体具有正的ΔAiN- P F I。这些结果与PFIs中残基聚集倾向的较大变化一致(图1c)。
作者使用了平均15个N−PFI二聚体的ΔAiN- P F I作为确定聚集热点的代理,图3c,d显示了ΔAN- P F I和MM/PBSA结合能之间的良好相关性。残基L38、L13、H31、E34、I10、Y37、Y47和P49对天然单体具有最大的ΔAiN- P F I,残基F46、Y37、I2、L27、E34、R43、F45、N18、Q5、H31、T48和C28对PFI具有最大的ΔAiN- P F I (见支持信息表S5),这些残基之间的一些相互作用被认为是胰岛素聚集核形成和原纤维形成的重要因素。结合界面上极性残基的存在表明疏水和静电相互作用将共同控制胰岛素原纤维形成的初始阶段。作者还发现N−PFI二聚体中的天然单体界面与胰岛素的受体结合域有密切的对应关系,从而将胰岛素的活性构象与其较高的聚集倾向联系起来。以上结果表明,天然单体上相对明确的区域与PFIs的不同区域结合。此外,尽管在一级序列水平上结合界面的组成存在异质性,但这些不同区域在残基极性方面具有相似的组成。
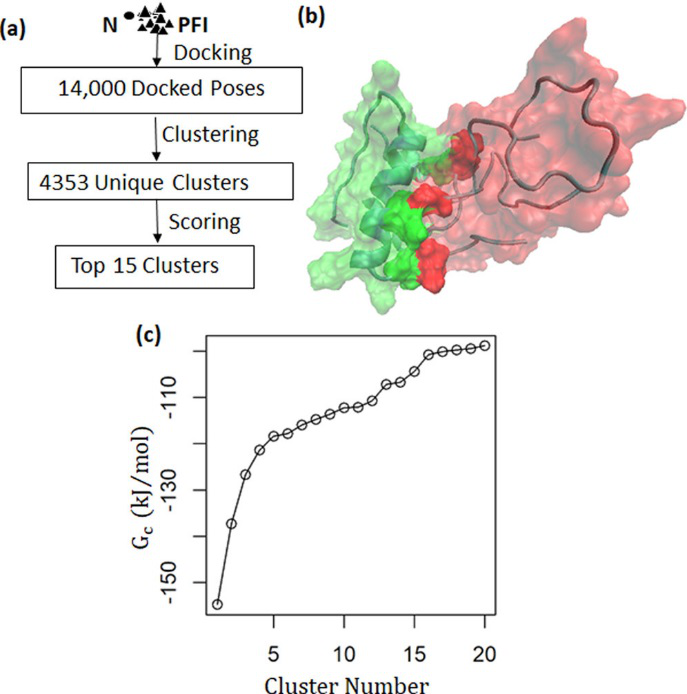
图2. 稳定的N−PFI二聚体研究
图片来源JCIM
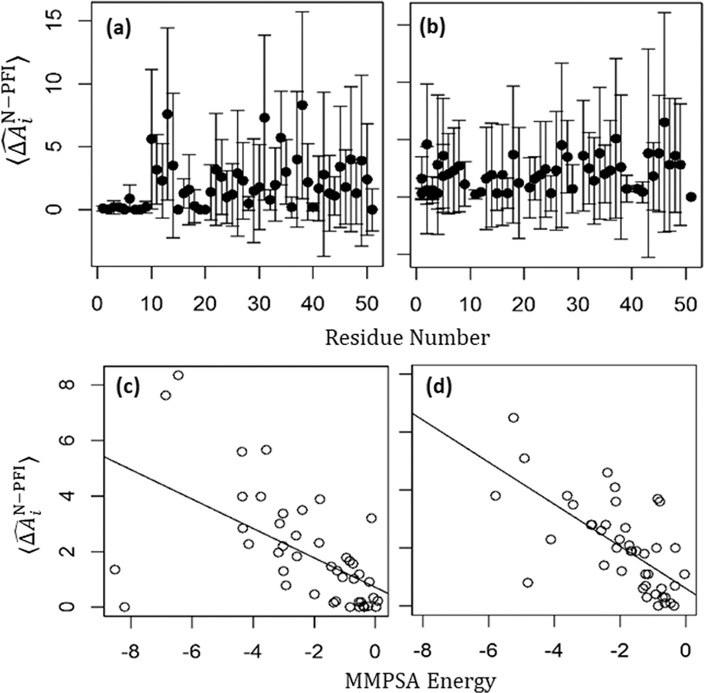
图3. N−PFI二聚体中结合界面残基的研究
图片来源JCIM
作者将N−PFI胰岛素同二聚体中天然单体(N)的结合界面残基作为模板,设计对胰岛素易聚集的PFI具有高特异性的肽。利用累积结合界面面积ΔAsegmentN- PFI对片段进行排序。对于任何9-mer 片段,R31-R39具有最高的ΔAsegmentN- PFI=33.4;对于任何5-mer 片段,R10-R14和R45-R49具有最高ΔAsegmentN- PFI=22.2、14.4。涉及这些片段的蛋白间相互作用已被证明可以稳定成熟的胰岛素纤维。来自这些片段的肽,被证明可以降低胰岛素聚集率和/或增加聚集延迟时间。两项研究中的肽,仅从天然单体的一个顺序连接的易聚集区获得,并与天然单体和PFIs结合。在本文研究中,作者利用空间上相邻的,顺序连接的片段来设计对胰岛素PFIs具有更高特异性的肽。肽序列是通过连接两个空间相邻的片段在一个残基对组成。这是通过根据残基对的Cα原子之间的距离插入甘氨酸残基、GA二肽或GAA三肽来实现的(见图4a、b)。作者总共获得了12101个长度为2-40个残基的肽,利用序列中残基对标准化结合界面面积(ΔApeptideN- PFI)的总贡献排序。由于长度相似的肽被证明能有效地抑制胰岛素聚集,因此其大小被限制在9个残基范围内。共得到665个ΔApeptideN- PFI 数值在9.8到34.5之间的九聚体,其中有20个较优的九聚体(见支持信息图S4)。接下来去除顺序相同的肽,获得八个唯一序列,即P1、P2、P3、P13、P16、P18、P19和P20。
根据它们的构象和肽-PFI复合物构象对这八种肽进行下一步的筛选,综合以下两点(i)肽-PFI复合物具有良好的结合自由能;(ii)肽-PFI复合物与N-PFI二聚体界面处的PFI残基高度相似,选出了高cosθ数值的肽P1(ICSLGAVLY)和P19(AAYLVAGLY),以及最突出的序列P3 (EAAYLAGLY)来表征抑制胰岛素聚集的功效。肽P1、P3和P19的最低RMSD构象与天然单体的相应界面区域的重叠分别如图4c-e所示。
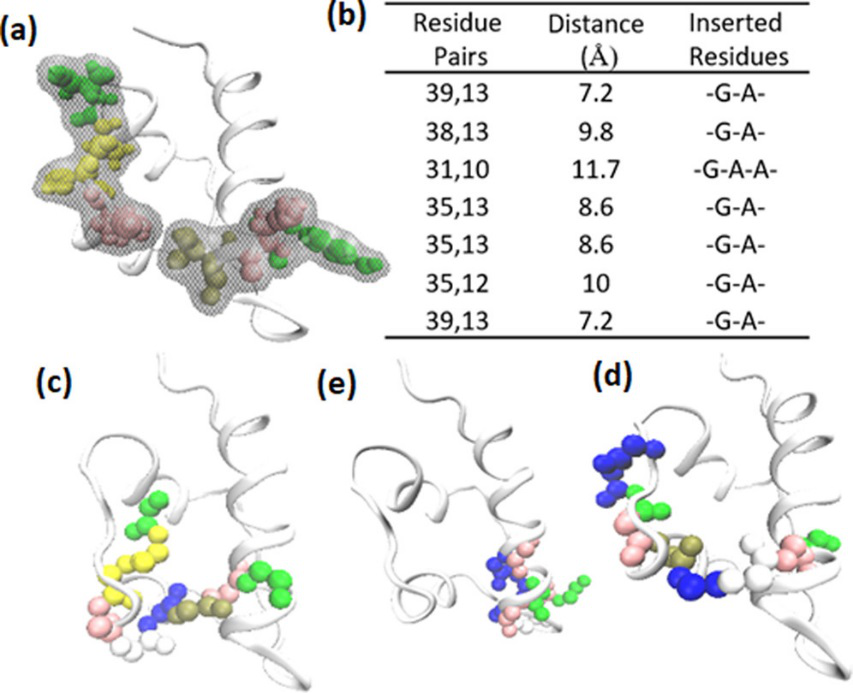
图4. 设计抑制聚集核形成的肽
图片来源JCIM
图5a显示,这三个肽中至少有一个对每个N−PFI二聚体具有高cosθ。图5b-d显示了肽-PFI复合物和N-PFI复合物的重叠图,分别对应于分别对应于肽P1、P3和P19的最高cosθ。正如预期的高cosθ,设计的肽模拟N−PFI相互作用,因此在与天然单体相同的位置与PFI结合。然而,所设计肽中高比例的疏水残基使其与水性制剂不相容。使用Innovagen进行溶解度计算,建议插入三个精氨酸残基增加水溶性。由于先前的研究表明以六聚精氨酸作为破坏结构域可获得显著的聚集抑制作用,作者在设计的肽的N端添加了相同的结构域。由较短片段组成的极性破坏结构域,对聚集抑制的影响较小。两亲性肽的最终序列为R6ICSLGAVLY((R6)P1)、R6EAAYLAGLY((R6)P3)和R6 AAYLVAGLY((R6)P19)。作者进一步对三种肽结构中最稳定、最高的cosθ的肽-PFI复合物进行显式溶剂MD模拟。通过轨迹分析,肽(R6)P1和(R6)P3保持与PFI的联系与PFI结合(见支持信息图S5),而(R6)P19先是远离后再与PFI的重新形成接触。
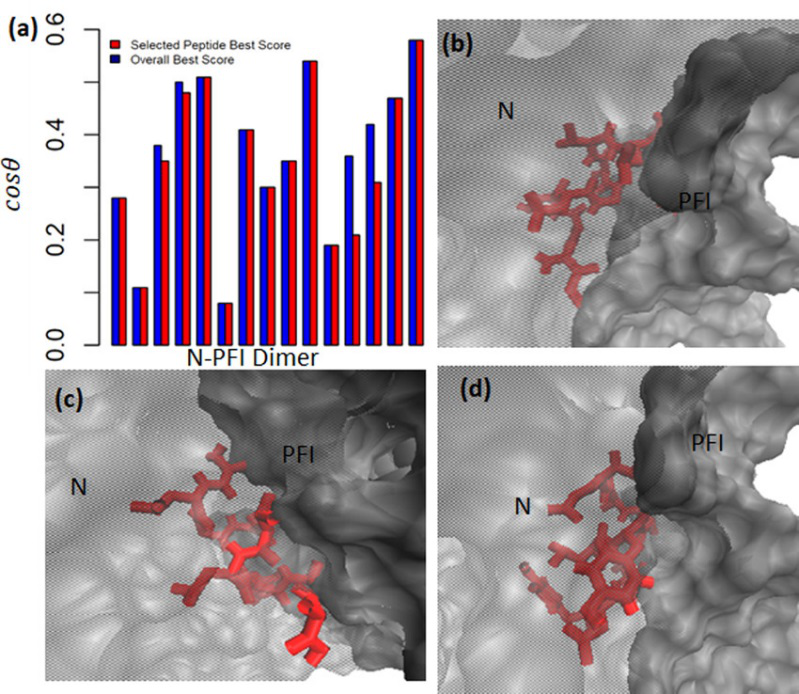
图5. 肽-PFI和N-PFI界面的比较
图片来源JCIM
作者利用生物分析技术研究了设计的肽分子对胰岛素稳定性和受体结合特性进行了评估。图6a-c显示单体胰岛素的损失和寡聚物在肽存在和不存在的情况下的数据。所有三种肽都以浓度依赖的方式在不同程度上保留单体胰岛素(图6a),(R6)P1的有效浓度范围存在显著差异,与这些肽的胶束化行为有关。肽的最佳浓度配方的远紫外镉光谱如图6d所示。综上所述,上述数据表明肽(R6)P3和(R6)P19有效地抑制胰岛素聚集,以单体形式保留90%的胰岛素,并几乎完全保留其天然的富含α-螺旋的构象。此外,长时间内单体损失率处于平稳状态(图6b),这表明了一种机制,即通过与抑制剂分子结合来隔离易聚集的PFI,抑制胰岛素聚集成核的早期阶段。为了进一步阐明(R6)P3和(R6)P19的稳定模式,作者使用ITC研究了它们与天然胰岛素的结合(见支持信息图S8)。ITC测量前后样品细胞的SEC色谱图(此处未显示)重叠,表明整个测量过程中只存在天然胰岛素单体。由于本研究中的肽和蛋白质在pH值范围2−3范围内都带有高度正电荷,肽-PFI复合物的形成将以疏水相互作用为主,这可以解释观察到的最佳稳定浓度接近于每个肽的临界胶束浓度。图7显示了不同胰岛素制剂(含或不含肽)在412nm处的相对吸光度,这些数据与图6中的数据一致。含有(R6)P3和(R6)P19的配方在培养72小时后仍保持78%和85%的胰岛素活性,而对照胰岛素配方(无肽)和含有(R6)P1的配方分别仅保留6%和20%的活性。
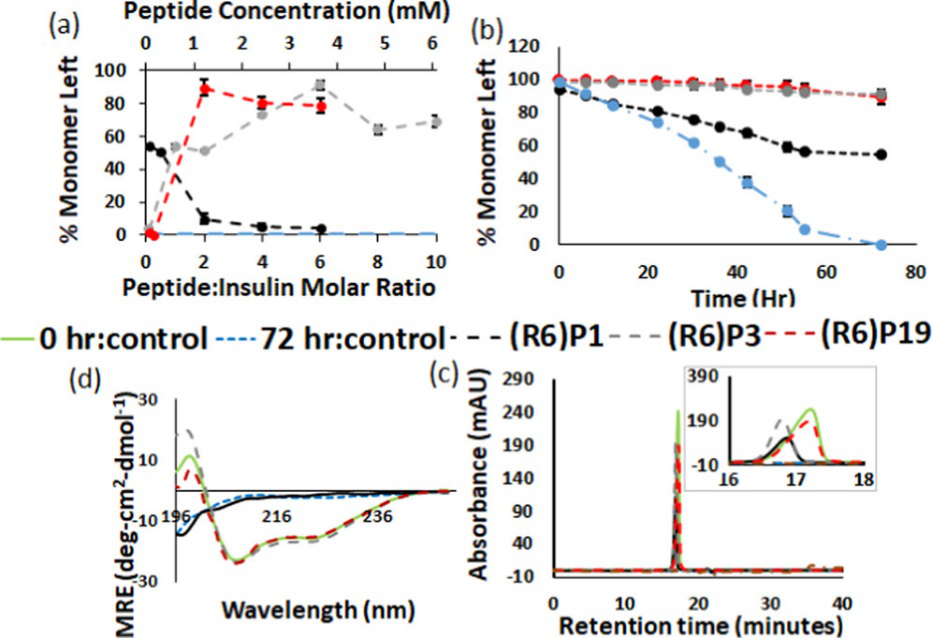
图6. 设计的肽分子对胰岛素制剂稳定性影响研究
图片来源JCIM
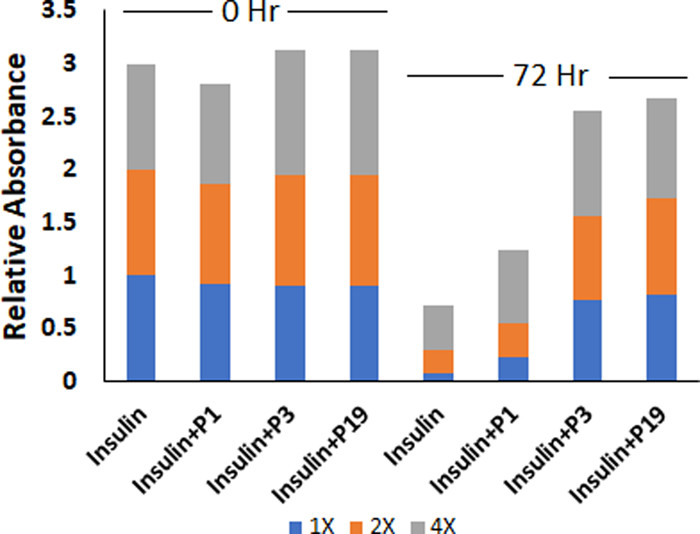
图7. 设计的肽分子对胰岛素制剂活性影响研究
图片来源JCIM
结论
作者将N−PFI胰岛素同源二聚体中天然单体(N)的结合界面残基作为模板,理性设计了一系列对胰岛素易聚集的PFI具有高特异性的肽,限制胰岛素的同型二聚化。当肽/胰岛素摩尔比分别为6:1和2:1时,15-mer肽(R6)EAAYLAGLY((R6)P3)和(R6)AAYLVAGLY((R6)P19)最为有效。这些肽组成的制剂以单体形式保留了>90%的胰岛素,并且在淀粉样变性条件下培养72小时后,在CD光谱中显示出特征性的α-螺旋倾斜。在相同条件下,不含肽的制剂显示单体胰岛素和胰岛素活性完全丧失。由与(R6)P3和(R6)P19组成的配方在72 h孵育后分别保留了78%和85%的天然胰岛素活性,因此有希望进一步开发。
参考文献:
Mishra, Avinash , et al. Structure-Based Design of Small Peptide Ligands to Inhibit Early-Stage Protein Aggregation Nucleation. J. Chem. Inf. Model. 2020, 60, 3304−3314