
引言
大规模的构象变化对于关联蛋白质的结构和功能都至关重要,虽然分子模拟能在原子层面观察构象的转变,但许多构象是短暂形成的,分布具有随机性,确定相关结构和它们相互转换的机制仍然是巨大的挑战。
本文中,研究人员以一种参与免疫反应并与HIV感染相关的蛋白CypA为例,介绍了结合加速MD(aMD)模拟和Markov模型(MSM)探索构象空间的方法。通过构建MSM模型确定了五个主要的优势构象状态以及状态相互转换的通路,并且通过理性设计,寻找到2个全新的突变位点,其中D66A会改变蛋白构象,而突变H70A可以稳定构象。aMD/MSM方法对于设计用于蛋白质工程和药物开发中蛋白质自由能分布的研究具有广阔的前景。
静态晶体结构的局限性
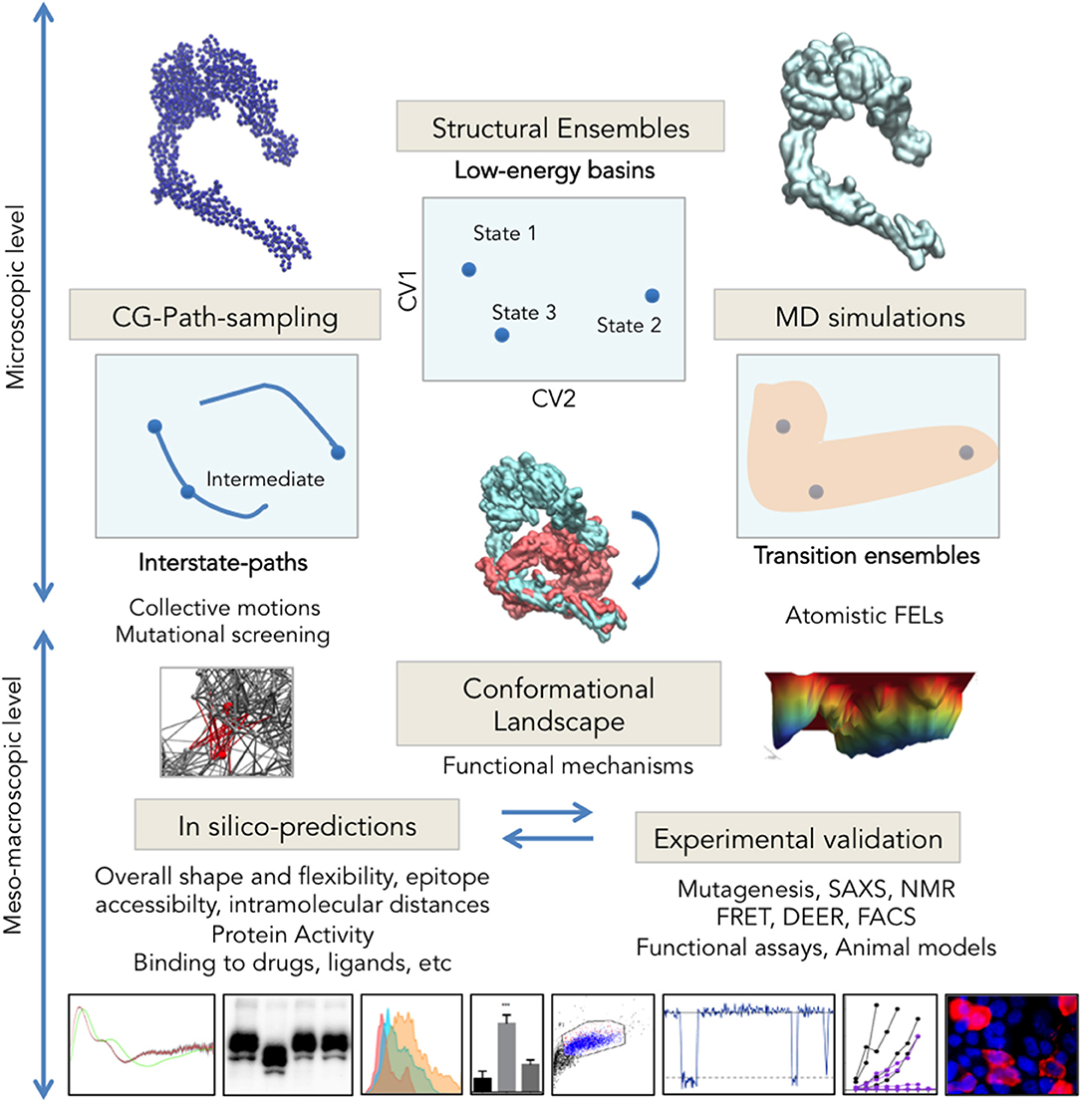
图1:综合多尺度的结构生物学
图片来源:Frontiers in Molecular Biosciences
分子动力学构象采样
分子动力学(MD)模拟在过去40年中经历了巨大的发展,可以称之为”计算显微镜“。随着分子力场的发展,高性能计算机的发展,MD模拟已经成为了研究和描述生物大分子结构变化以及动力学行为的重要工具。如果对局部结构的探究,MD模拟时长往往在几百纳秒的尺度就足够。但是对于全空间或者某些感兴趣构象的采集,则需要更长的模拟时间,一般是为毫秒的时间尺度。而且由于过渡态的随机性,时间尺度和本身较高的能垒,使得对过渡态的采样难上加难。
目前流行的是两种类增强采样方法,要么通过增加模拟覆盖有效时间,能够详尽地采样,比如元动力学(metadynamics),伞形采样(umbrella sampling); 要么显着改变力场的势能函数(scaled MD或者加速MD(aMD)。前一类方法要求预先提供感兴趣运动的自由度,其前提是已经知道相关运动。后一类方法可能会高估势能面(Free energy Surface)的高能区域,因此会探索构象空间的非物理区域。比如加速MD方法(aMD)就是在体系能量下降时改变盆地的相对高度,从而使FEL(Free energy landscape)局部变得平缓。
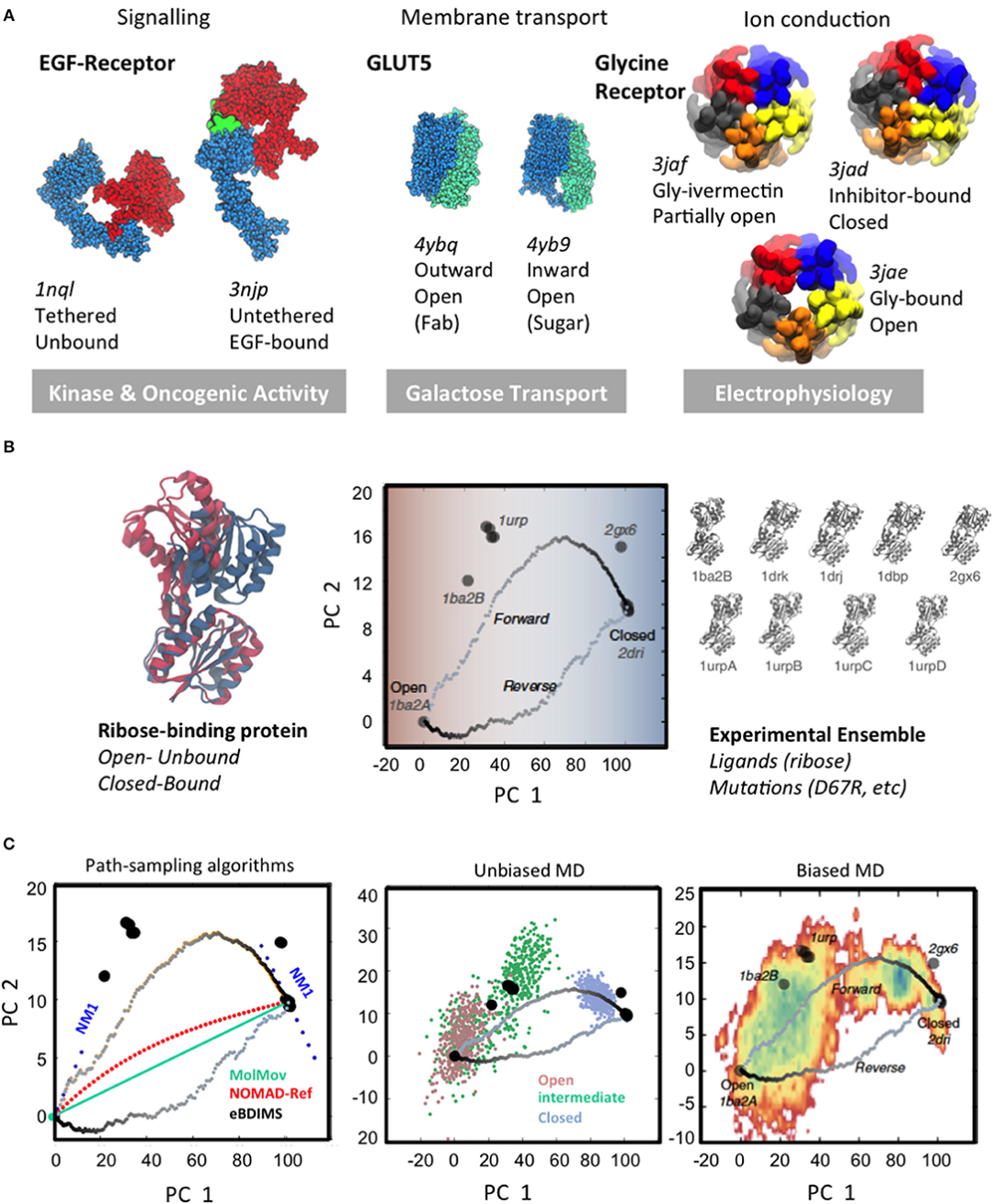
图2:大规模构象变化和不同规模的采样方法
图片来源:Frontiers in Molecular Biosciences
马尔可夫模型
为了改进这些方法,最近5年,基于马尔可夫模型(MSM)的采样方法逐渐流行,可以在毫秒级的时间尺度上采样蛋白质动态。MSM本身是一种统计方法,串联过渡来描述不同的状态。MSM可以从一组较短的模拟中推断出长期的动态变化,为采样难题提供了又一个捷径
如何在分子模拟中构造马尔科夫模型(MSM)
建MSM模型,一般要选择n个状态,使得它们涵盖了整个动力学行为,并且滞后时间τ足够长以成为马尔可夫模型,但又短得足以解决系统动力学问题。如果能够成功做到这一点,则MSM仅从其过渡矩阵提供有关系统的有价值的信息, 如下图所示,a图表示的是八段不同的短时间的模拟轨迹,每一段模拟中可以选取不同帧的由一个模拟时间步分开。B图表示将轨迹分解为若干离散状态,其中已识别出四个状态(绿色,蓝色,紫色和粉红色)。C图表示的是观察到的转换计数矩阵,d图是可逆转换概率矩阵, e图是总体概率分布的饼图,这是概率矩阵的第一个特征向量。占比比较高的状态在热力学上也更趋于稳定。
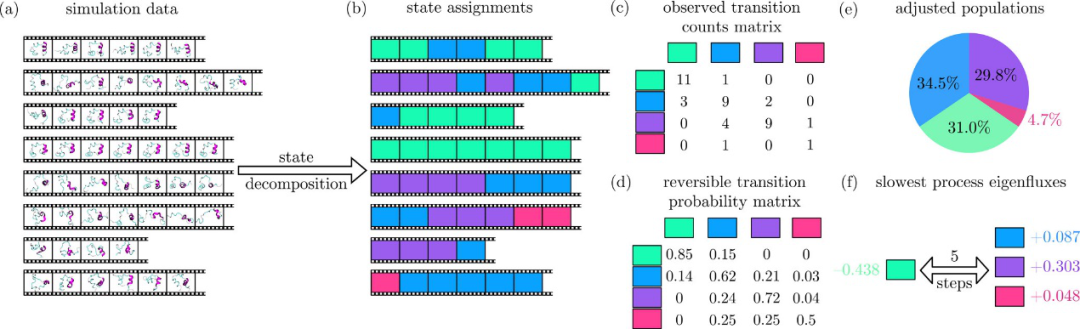
图3:马尔可夫模型构建
图片来源:J. Am. Chem. Soc.
方法与软件
分子动力学模拟:AMBER16和GROMACS5.0
模拟轨迹分析与处理:MDTRAJ
MSM构建:PyEMMA 2.3.0
化学位移计算:ShiftX2和MultiEx
晶体结构优化: CCP4
结果
图片来源:Chemical Science
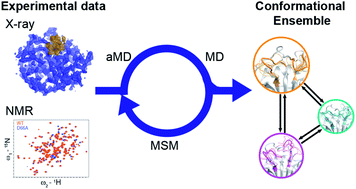
在这项工作中,研究人员设计出合适的过渡态采样流程(图4b)。这是通过利用aMD高效探索构象空间的能力来实现的,同时依靠MSM程序来描述体系的更准确的热力学和动力学行为。研究人员将aMD / MSM方法应用到人亲Loop蛋白A(CypA)的势能面搜索中。CypA酶是人类脯氨酰异构酶,将其掺入新的病毒装配中对于HIV-1感染和HCV复制是必不可少的,这使它成为主要的药物靶标。之前的研究揭示了变构调控的复杂网络,这个酶具有两个功能Loop,残基65-77组成的70S loop(图4a)和100-110残基组成的100sLoop, NMR实验发现它可在毫秒级的时间尺度上经历动力学变化。研究人员探究了其中100s和70s回路在open 状态和closed状态之间的交换。
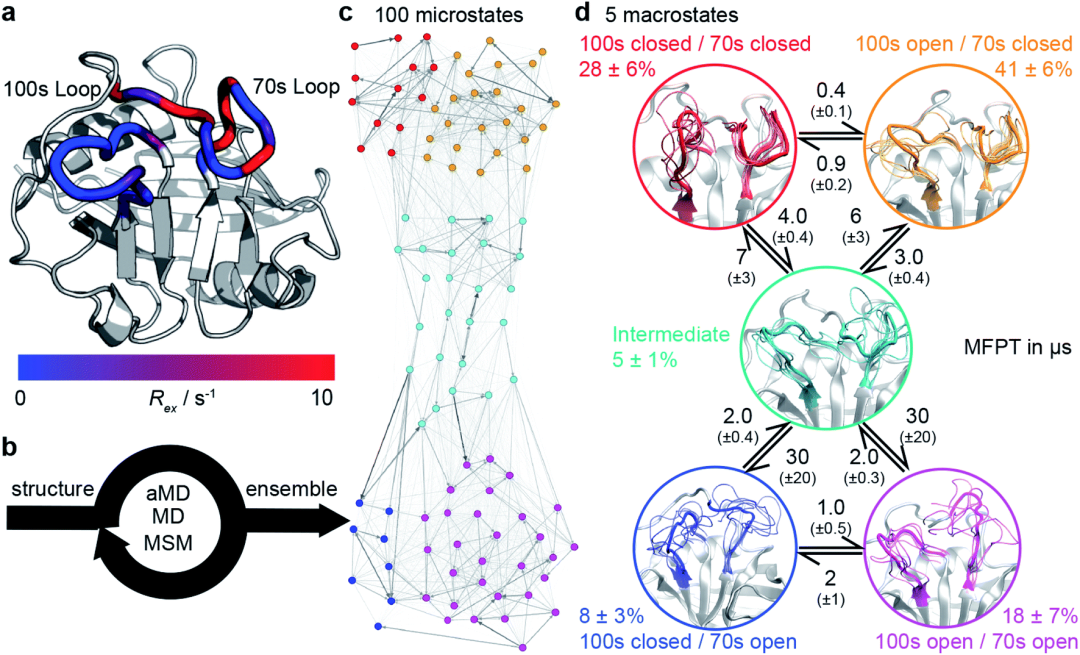
图4:计算WT-CypA的动态运动过程
图片来源:Chemical Science
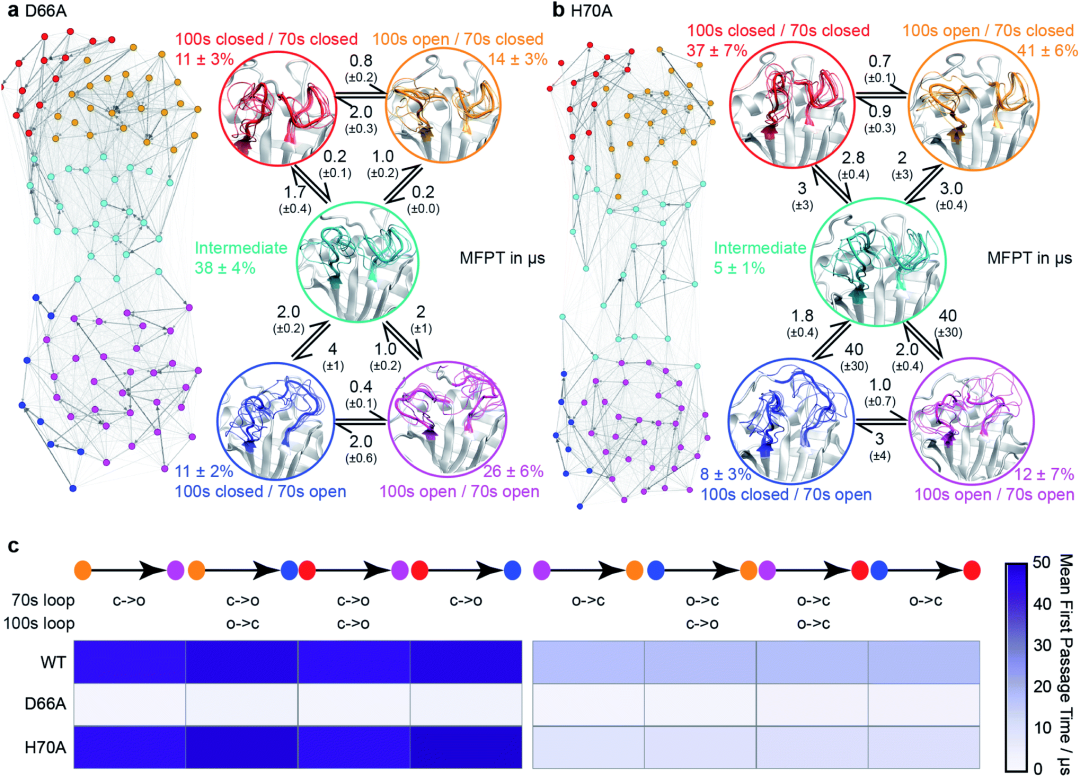
为了能够更好的研究势能面,研究人员实施了一种基于氢键模式的设计策略,以稳定特定稀疏的具有毫秒时长的构象(图5)。该过程使得研究人员发现了新的突变体D66A。WT的70sLoop主要处于闭合状态,这与迄今为止已经解析的大多数CypA结构一致,而D66A预计将主要占据70sLoop的开放状态。类似地,作为阴性对照,aMD / MSM预测突变H70A与WT非常相似。为了构建MSM模型,研究人员100个MSM微观状态汇总5个宏观状态模型中。这五个状态的构象态势揭示了100s和70s循Loop都可以采用“开放”和闭合”的构象。虽然两个Loop的相互转化在很大程度上是独立的,但100sLoop的相互转化比70sLoop的相互转化快一个数量级(图5d),这表明蛋白更慢的动力学行为和70s loop 的打开与闭合相关。为了验证这些发现,研究人员找了一个突变位点,该突变可以稳定70S的开放构象(青色/蓝色/紫色)。基于开放和闭合的构象这两种形式评估了每个残基与70s loop形成的氢键数量(图5B)。值得注意的是,发现D66在闭合构象中采用了大量的氢键,而在开放构象中的氢键数量则少得多(图5C),这使该残基脱颖而出,成为“可设计的”位点。随后NMR的结果也验证了H键数量差异的情况。
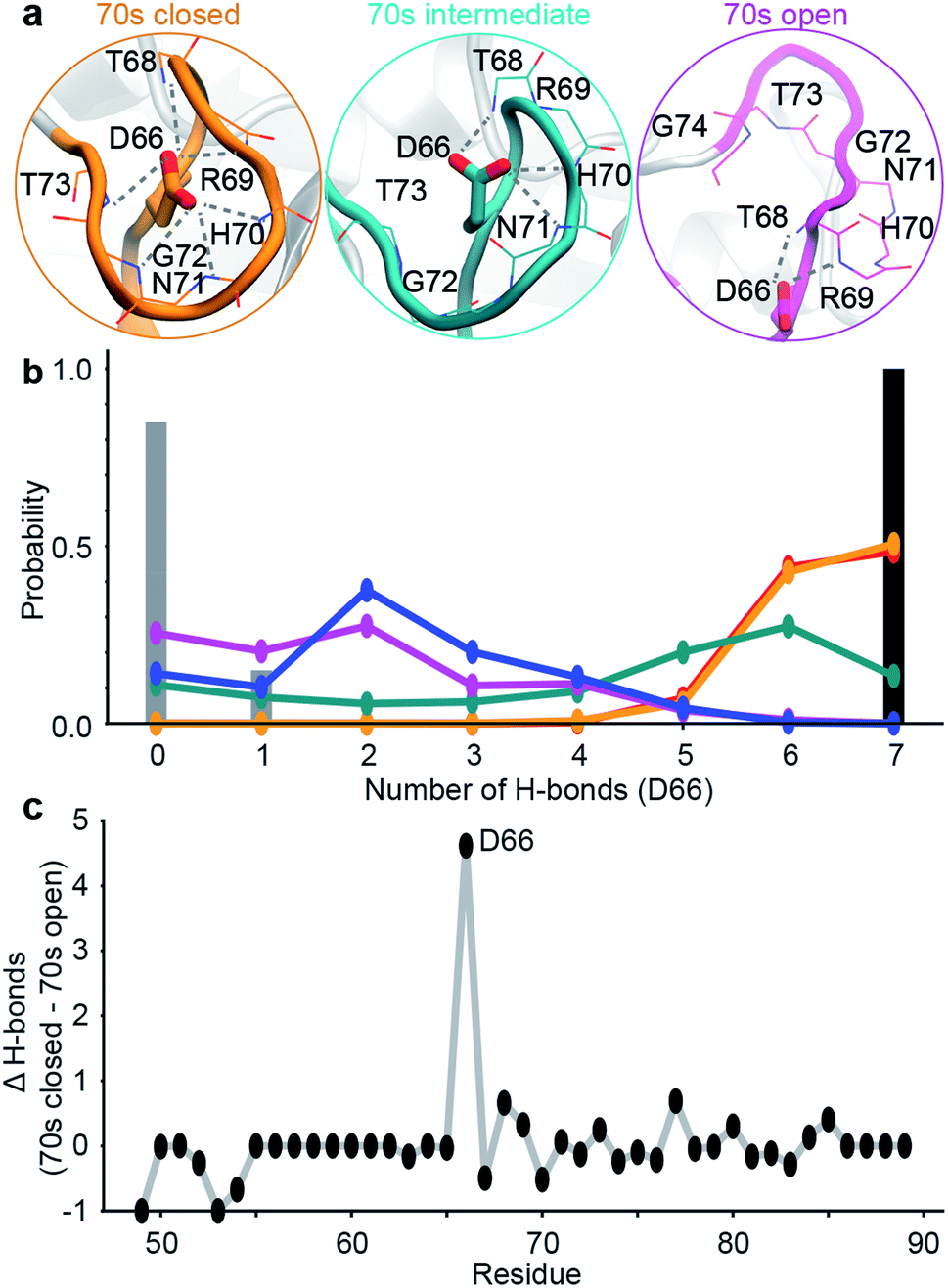
图5:突变的设计
图片来源:Chemical Science
参考文献:
1.Husic, B. E.; Pande, V. S. Markov State Models: From an Art to a Science. J. Am. Chem. Soc. 2018, 140, 2386-2396.
2.Orellana, Laura. 2019. “Large-Scale Conformational Changes and Protein Function: Breaking the in Silico Barrier.” Frontiers in Molecular Biosciences 6: 117.
3. Juarez-Jime ́ nez, J.; Gupta, A. A.; Karunanithy, G.; Mey, A. S. J. ́S.; Georgiou, C.; Ioannidis, H.; De Simone, A.; Barlow, P. N.; Hulme, A. N.; Walkinshaw, M. D.; Baldwin, A. J.; Michel, J. Dynamic Design:Manipulation of Millisecond Timescale Motions on the energy Landscape of Cyclophilin A. Chem. Sci. 2020, 11, 2670−2680.