
文章简介
作者采用PM6方法计算了分子几何优化构型和电子性质,计算基数为PubChem Compounds(截止2016年8月29日的数据)的9,160万个分子的94%分子。除了中性状态的分子,作者还计算了56.2%的阳离子状态、49.7%的阴离子状态和41.3%的自旋翻转状态的分子。因此,作者采用PM6方法计算的分子总数达2.21亿个。作者对比了260万个分子的PM6法和B3LYP/6-31G*法的几何优化构型结果,键长和键角的标准差分别为0.016 Å和1.7°;并对两种方法的HOMO能级和LUMO能级进行线性回归分析,得出HOMO能级关系为EB3LYP(HOMO) = 0.876EPM6(HOMO) + 1.975(eV),计算确定的系数为0.803;LUMO能级关系为EB3LYP(LUMO) = 1.069EPM6(LUMO) – 0.42(eV),计算确定的系数为0.842。作者还生成了4个子数据集,每个子集中的分子质量都低于500,子集一包含 C、H、O、N四个元素,子集二包含C、H、N、O、P、S六个元素;子集三包含C、H、N、O、P、S、F、Cl八个元素;子集四包含C、H、N、O、P、S、F、Cl、Na、K、Mg、Ca十二个元素。
背景介绍
随着有机薄膜太阳能电池、电发光材料、有机非线性光学材料、分子传感器和创新药的开发和发展,新的有机分子的发现越来越重要。太阳能电池的化学性质如激发能、最高占据分子轨道(HOMO)与最低未占据分子轨道(LUMO)的能隙等非常重要,而量子化学从头计算法不需要昂贵的物理或化学实验就可以精确预测化学性质。遗憾的是,这种量子化学计算方法的计算费用虽然已经降低很多,但对于研究分子化合物的化学空间来说仍然太慢。根据Lipinski成药性规则,目前候选药物预计已达到1060个,数量相当庞大。机器学习被证实能弥补量子化学计算方法缺陷,该方法在过去十年已经广泛应用于分子和固体研究,尤其是深度学习,已经引起相当大的关注。深度学习基于定量结构-性质关系(QSPR)数据训练,采用多层人工神经网络创建预测模型,而不是采取“第一性原理”计算化学性质。总的来说,为了使用有监督深度学习方法来开发精准模型,我们需要大量的可信赖数据用于训练。
我们报告了大规模的量子化学数据集——PubChemQC PM6数据集,数据集采用PM6方法对PubChem Compounds(提取日期2016年8月29日)上的分子进行几何结构最优化、电子结构和其他性质的计算。PubChem Compounds是最全面的化合物数据库之一,截止2016年8月29日已收录了大约9,160万个分子,且每天都在增加。在撰写本文时,作者已经成功完成计算的分子包括86,213,135个中性分子、51,555,911个阳离子态分子、45,581,750个阴离子态分子及37,839,619个自旋翻转态分子,合计计算了221,190,415个分子的最优几何结构和电子结构。PubChemQC PM6覆盖了超过94%的中性分子。此外,作者为了方便用户使用,通过限制元素和分子量、去除盐基等维度创建了4个子数据集。数据集网址:http://pubchemqc.riken.jp/pm6_datasets.html。
建立这些数据集的目的是向研究人员或从业人员提供全面的量子化学数据集,应用领域包括但不限于机器学习、高通量虚拟筛选等。目前互联网上已有多个量子化学数据集,但它们的范围和规模仅限于几百上千个有机小分子。据目前所知,在半经验量子化学计算方法创建的数据集中,PubChemQC PM6数据集是最大的。如果忽略计算方法及其计算准确性的不同,PubChemQC PM6数据集里的记录数量大大超过了任何通过量子化学计算获得的数据集。采用密度泛函理论(DFT)方法创建的数据集比PM6半经验方法的准确性高,如哈佛清洁能源项目数据库(Harvard Clean Energy Project Database)和ANI-1数据集,前者收录了230万个由26个构建基块组成的有机光伏候选化合物,后者收录了57,462个有机小分子的2,000万个经计算的非平衡构象。然而,和PubChemQC PM6相比,这两种数据库的分子种类非常有限,PM6数据集包含了2.21亿个分子几何和电子结构,覆盖70种元素。作者使用PM6方法建立数据集的原因有3个,第一,计算成本大大低于那些更准确的计算方法;第二,作为分子结构优化的初步推测,计算结果更优越,因为采用PM6方法的分子几何优化能用于构象分析,能用于减少各种高精密计算方法的计算成本(如B3LYP/6-31G*, CCSD(T)/ccpVDZ, 等等)。值得一提的是,PM6方法为包括蛋白质在内的分子提供了相当优异的几何结构;第三,计算结果本身就具有价值,其能用于预测分子的电子结构。据文献报告,PM7方法结合机器学习可能可以高准确性的预测电子结构,值得一提的是PM6和PM7方法在小分子的几何优化方面有着相似的准确性。为了确认计算出的分子几何结构的准确性,作者对比了260万个PM6法和B3LYP/6-31G*法优化的几何结构。B3LYP/6-31G*法计算的键长和键角的标准差分别约为0.016Å and 1.7°,这些值几乎和Stewart的原始结果一样。
创建PubChemQC PM6数据集的方法
——选择分子的标准
分子的数量极大,即使限制原子种类和组成的原子数量,分子的数量仍然是个天文数字,例如Ruddigkeit等人创建的数据集GDB-17,里面列举了1660亿个由C、N、O、S和卤素等17种原子组成的有机小分子,这个数据集的问题就在于不能直接判断哪个分子必不可少。Ramakrishnan等人建立了GDB-17的子集,包括QM7、QM7b、QM8和QM9数据集,里面采用量子化学方法计算了几何和电子结构。其中QM9数据集是最大的,但里面只有13.4万个分子。此外,当分子大小稍微大一点点,采用枚举法的GDB-17数据集可能就不能正常运行,比如C32的异构体已经能列出27,711,253,769个,尽管这些异构体间的区别几乎可以说是微不足道。又如顺反异构,N个双键的顺反异构体有2N个,但是有些异构体不能展示出来,不能直接决定一对异构体中应该选择哪个。有些异构体非常不同,有些异构体却相似,如顺式不饱和脂肪酸有利于维持良好的胆固醇水平,而反式脂肪酸则被认为是有害的;1, 3二氯丙烯作为农业化工品使用时,顺反结构没有明显的区别,通常混合用于农药。因此,与其主观选择分子,作者决定基于现有的已收录必要分子的数据库来开发化学数据库,并尽可能多的对数据库中的分子进行量子化学计算。目前已有很多的化学数据库包括CAS、ChEMBL、ChemSpider、Zinc等等,作者选择PubChem化合物库是因为其可及性、大小和多样性,Zinc、ChEMBL和PubChem可免费试用,其他数据库有部分限制。CAS是专有数据库,ChemSpider需要权限才能组装超过5000个结构的数据库。PubChem(2016年)收录了大约1亿个分子,而ChEMBL15只有180万个分子,Zinc15有1.2亿个分子。尽管PubChem和Zinc15的数据库大小相当,但作者仍选择了PubChem,因为PubChem数据来源多样,收录的信息不止来自化学品供应商目录和期刊出版商,甚至来自于Zinc,ChEMBL和ChemSpider。PubChem是一个开放式化学数据库,隶属于NIH(美国国立卫生研究院)。自2004年起,PubChem就不断更新来自全世界的分子信息,数据采集自数百个大数据源包括大学、药企、政府机构、化学品供应商、科学文献和其他管理机构。PubChem包含3个子数据库:Substances, Compounds,和BioAssays,Substances收集申报数据,Compounds收录了从Substances数据库提取的独特的标准化合物数据。截止2016年8月16日,PubChem Compounds收录了91,679,247个化合物,每一个化合物都有InChI和SMILES编码以及PubChem CID号码,分子式和其他信息。
创建PubChemQC PM6数据集的方法
——分子的展示
一个分子没有严格的定义,但是我们可以在一定的假设条件下定义一个分子。在Born-Oppenheimer近似气相、非相对论极限和点电荷模型中,我们可以在笛卡尔坐标系中从一组原子(核电荷)中确定一个分子的汉密尔顿量以及系统中的电子数量。然后我们可以通过解薛定谔方程来获得波函数和量子数。用这种方法,一个分子就由汉密尔顿量、波函数和量子数定义,这种定义的弊端是不易区分两个相同的分子或者在其他方面不同的分子如同分异构体。另一方面,最方便的展示方法是有一个通用名,而每个新引入的化合物都必须命名。我们可以简单采用IUPAC命名法作为系统命名法,因为大部分情况下原子、原子间的连接、键级和其他立体信息等就足以确定一个分子。但是我们不容易在电脑运行IUPAC名字。基于此作者决定采用人类和机器可读的分子编码系统如InChI和SMILES,能与IUPAC命名法兼容,采用类似化学分子式的系统方法来编码分子。PubChem Compounds采用InChI和SMILES两种方法来编码化合物,作者在计算过程中广泛使用了这些编码,即使用SMILES系统生成初始几何结构预测,用InChI系统确认最优结果。需要注意的是这些编码系统也有一些模糊的地方,只能确定一组原子、电荷和原子连接的一些信息以及立体异构。
PubChemQC PM6数据集的详细信息
——创建数据集
首先,从PubChem站点下载所有分子的SDF文件,解析和提取每一个分子的CID、分子量、InChI和SMILES信息、分子式、电子电荷和自旋量子数,通过异构体的SMILES编码计算电子电荷,根据系统的电子数量的奇偶性将自旋量子数设置为0或1。然后根据分子量进行降序排列,剔除分子量大于1000g/mol的分子(上限高于Lipinski成药性原则的500g/mol)后剩余PubChem Compound中的604,330个(占比0.66%)分子。同时剔除2,188,881个(占比2.39%)带电分子。输入文件由Open Babel生成,采用分子的异构SMILES编码,选项为-addh和-gen3d。通过对比原始数据集中的分子式和分子的SMILES编码生成的结构来确认氢原子数量,因为Open Babel有时会给分子添加过多或过少的氢原子。作者没有用PubChem3D来展示分子几何结构,原因在于(1)SMILES和InChI表示法比分子几何结构小得多,因此更容易处理;(2)和PM6分子几何优化相比,Open Babel计算初始几何预测值所需要的计算资源是微不足道的;(3)PubChem3D的分子几何构型的可及性受限。最终,作者使用Gaussian09来进行每个分子的PM6几何构型的优化计算。成功之后,采用PM6几何优化模型对阳离子态、阴离子态和自旋翻转状态的分子也进行了几何构型最优化,得到初始预测值。接下来使用Open Babel对分子的中性状态进行确认,确认计算好的最优几何构型的InChI是否与原始InChI一致。采用sed脚本来描述2个InChI表示法,并验证输出的特征。作者忽略了游离氢和原子的形式电荷,因为不能预测或确定它们的量子化学计算输出的意义。作者还忽略了总电荷,总电荷在几何优化过程中应不变。同样的,立体异构体、几何异构体和构象异构体也不在考虑范围内。每一个CID数据如下:输入文件,xyz格式的原子坐标和JSON文件。所有计算都在RIKEN HOKUSAI BigWave超级计算机(Intel Xeon Gold 6148 2.4GHz, 1680CPUs, 33600 cores),QUEST集群 (Intel Core2 L7400 1.50 GHz, 700 nodes, 1400 cores), 和 RIKEN RICC 超级计算机 (Intel Xeon 5570 2.93 GHz, 1024 nodes, 8192 cores)运行。总的计算时间为HOKUSAI BigWave 95天、QUEST 346 天、RICC 126天,2016年12月30日开始计算,到2018年6月9日结束,2018年10月12日完成项目总结。
PubChemQC PM6数据集的详细信息
——数据
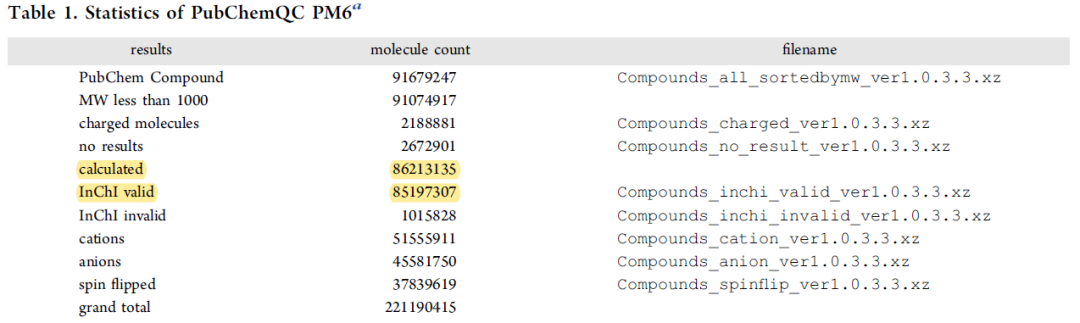
表1. PubChemQC PM6的数据量,总共计算了86,213,135个中性分子,其中85,197,307个分子的原始InChI和PM6优化几何结构计算得出的InChI的化学分子式和主层原子连接相一致。
表格源自JCIM

表2. PubChemQC PM6数据集中每个分子可用的数据,包括振动频率、振动强度、SCF能量。
表格源自JCIM

表3. PubChemQC PM6子数据集的数据,每个子集包含的分子质量都低于500,所有分子都是中性状态和单峰状态,CHON意思是化合物只包含C, H, N, O原子,前三个子集均去除了盐基。
表格源自JCIM
表1和2列出了PM6几何结构优化数据,包括大量化合物和包含详细数据的文件名字,比如PubChem CID、分子量、InChI、异构体的SMILES以及分子式。比如,“MW less than 1000”列出了PubChem Compound库中分子量低于1000的分子数量;“charged molecules”列出了带电分子的数量和包含详细数据的文件名;“no results”列出了PM6几何结构优化失败的分子数量;“InChI (in)valid”列出了在PM6优化几何构象中原始InChI和经计算过的InChI的化学分子式和主层原子连接(不)一致的分子数量;“cations” “anions” “spin flipped”是计算的阳离子、阴离子和自旋翻转状态的分子,这些分子都是来自PM6优化过的几何中性状态分子,较中性分子不稳定,因此更难计算。“grand total”是中性状态、阳离子状态、阴离子状态和自旋翻转状态的分子总和。为了用户方便,作者创建了4个较小的数据集(表3),选择分子质量低于500的分子,符合Lipinski原则之一。第一个子集包含了生物体中最常见的元素,第二个子集包含了生物分子的重要元素,第三个子集加入了轻卤素原子F和Cl,第四个子集包含了人机体的基础元素,除了氟。
PM6和B3LYP分子计算的对比
——分子的几何构型
为了看看PM6和B3LYP/6-31G*优化几何结构计算方法的区别,作者选取键长和键角的标准差(RMSD)进行比较,这两个指标能获得相对准确的实验结果,因此常用于评估量子化学方法的准确性。另一方面,比较分子的标准差较难,不做分子的标准差对比的原因如下:第一,实验已表明乙烷在室温条件下容易发生异构翻转,烯醇的互变异构化也频繁发生,因此较难获得精确的结果;第二,构象异构体的搜索从计算方面来讲具有挑战性,即使是如L-半胱氨酸这样的小分子,也需要耗费2个月的时间来获得稳定的构象。因此,作者只对比PM6和B3LYP/6-31G* 这两种几何优化方法所预测的2,606,946个分子的键长和键角。加入对比的分子数量少于原始PubChemQC数据集(包含3,981,230个分子),原因如下:第一,基础数据集不同,B3LYP/6-31G*方法用的是截止2014年7月PubChem compounds里的数据,而PM6用的是2016年8月的数据,两个数据集有很大的不同。虽然两个数据集共用了2,777,085个分子(CIDs),与2014年7月的版本相比,2016年8月的版本去掉了超过一百万个分子或移到不同的CIDs。第二,作者优化过程中去掉了每个CID对应的变形分子,确切的说,剔除了PM6和B3LYP优化方法计算后SMILES不一致的分子。
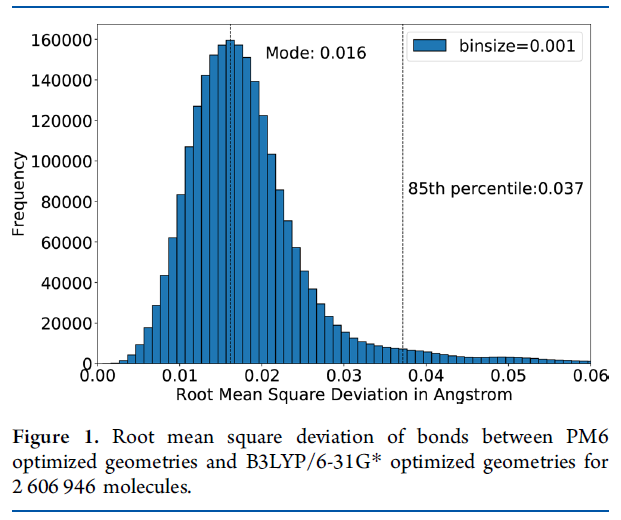
图1. PM6和B3LYP/6-31G*方法对2,606,946个分子的几何优化构象的键长标准差。
图片源自JCIM
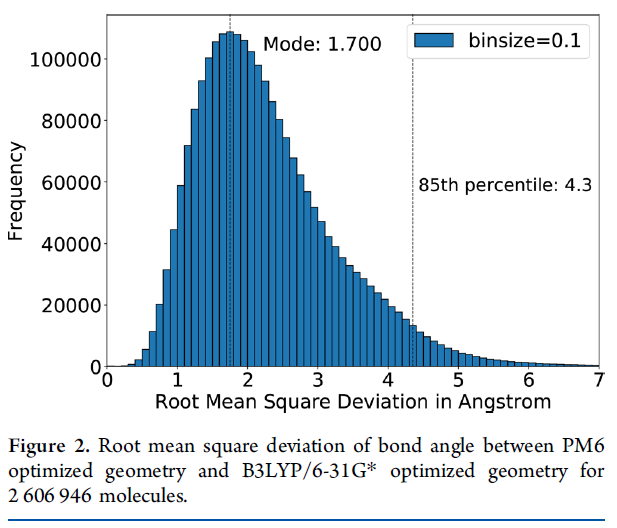
图2. PM6和B3LYP/6-31G*方法对2,606,946个分子的几何优化构象的键角标准差。
图片源自JCIM
图1描绘了两种方法分子优化后的键长(单位:埃,Å)标准差的直方图。键长标准差直方图的一个箱宽度(bin size)为0.001 Å,众数是0.016 Å,尖峰明显,第85百分位数为0.037 Å。结果显示PM6分子几何构象略微好于Stewart的方法,PM6计算的由C、H、O、N组成的化合物的平均不知名错误为0.025 Å。图2描绘了两种方法分子优化后的键角(单位:埃,Å)标准差的直方图。键角标准差直方图的箱宽度为0.1°,众数为1.70°,尖峰明显,第85百分位数为4.3°,结果主要与原始论文结果一致,100个由C、H、O、N组成的化合物中键角计算的平均不知名错误为3.1°。作者发现PM6和B3LYP/6-31G*两种方法计算的键长和键角的标准差以在0.016 Å和1.70°出现尖峰为特征,这些数值几乎与PM6的原始论文报告相同,同时作者也在260万个分子中验证过除氢键外的所有键长和键角。有趣的是,所有的计算结果和B3LYP几何结构优化结果一致,键角和键长与实验结果相差也只在几度和0.02 Å范围内。
HOMO、LUMO的能级和HOMO-LUMO的能隙
HOMO能级,LUMO能级和HOMO-LUMO能隙是很重要的信息,与反应性、光激发和电荷运输有关,对于开发有机发光二极管、有机光伏设备、有机薄膜晶体管等材料很重要。分子的HOMO-LUMO能隙越大,分子越稳定。当一个分子在HOMO能级释放电子,另一个分子在LUMO能级接受电子时发生电荷运输。光激发发生在一个分子吸收光子产生电子-空穴对,电子靠近HOMO能级,而空穴靠近LUMO能级。因此,比较PM6和几何优化法和B3LYP/6-31G*几何优化法获得的分子的HOMO、LUMO的能级和HOMO-LUMO的能隙会很有意思。根据分析,作者预估两者的HOMO能级,LUMO能级和HOMO-LUMO能隙关系分别如下式(图3-5):EB3LYP(HOMO) = 0.876EPM6(HOMO) + 1.975;EB3LYP(LUMO) = 1.069EPM6(LUMO) – 0.42和EB3LYP(GAP) = 0.959EPM6(GAP) − 3.165。同时三个关系式对应的系数分别为0.803, 0.842和0.779。
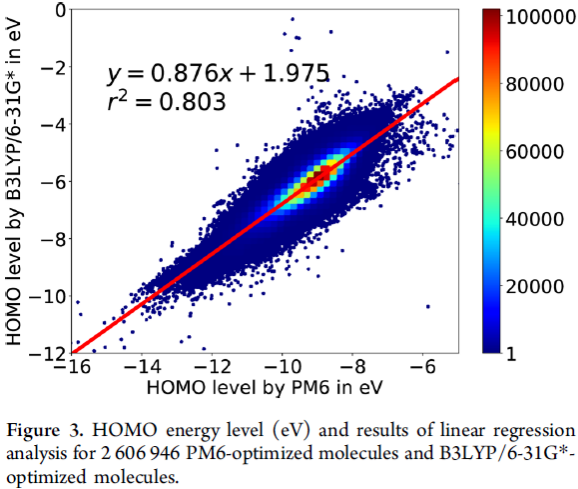
图3. PM6和B3LYP/6-31G*优化的HOMO能级和线性回归分析结果。
图片源自JCIM
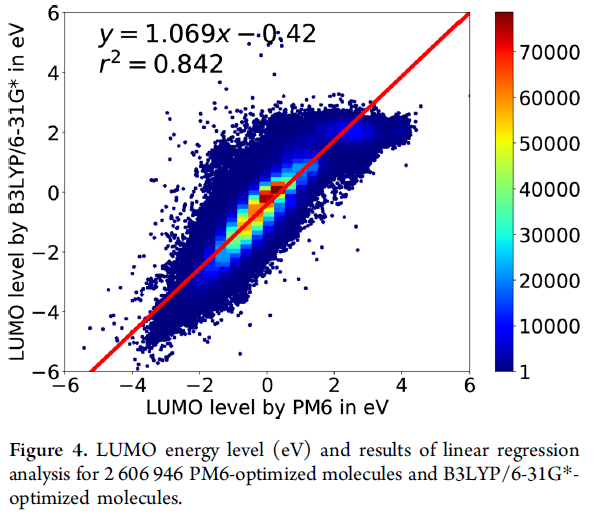
图4. PM6和B3LYP/6-31G*优化的LUMO能级和线性回归分析结果。图片源自JCIM
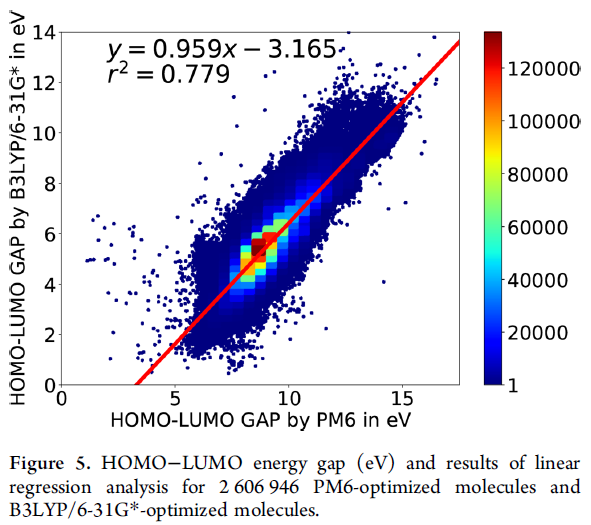
图5. PM6和B3LYP/6031G*优化的HOMO-LUMO能隙和线性回归分析结果。
图片源自JCIM
前述确定的系数大部分与Pereira等人的研究成果(Pereira, F.; Xiao, K.; Latino, D. A. R. S.; Wu, C.; Zhang, Q.; Aires-de Sousa, J. Machine Learning Methods to Predict Density Functional Theory B3LYP Energies of HOMO and LUMO Orbitals. J. Chem. Inf. Model. 2017, 57, 11−21.)一致,他们使用JChem软件以经验为主地生成单一构象异构体,采用半经验PM7法优化几何构型,再采用B3LYP/6-31G*理论计算单点;他们确定的HOMO、LUMO、HOMO-LUMO能隙的系数分别为0.799、0.895、0.656。作者的方法和Pereira等人的方法稍微不同,和他们直接对比是不可能的,第一个是计算方法的不同,Pereira等人用的是PM7,作者用的是PM6;第二是分子几何,Pereira等人用PM7优化几何适用于B3LYP/6-31G*计算,作者采用了2种不同的几何构型——PM7优化几何和B3LYP/6-31G*优化几何;第三用于对比的分子数量也不同,Pereira等人用了111,725个分子,而作者使用了2,606,946个分子,前两点可能对结果影响不大,但是使用相当大数量的分子作为对比使作者的结果更准确。因此可以预测B3LYP/6-31G*法的HOMO能级、LUMO能级和HOMO-LUMO能隙与Pereira等人机器学习的结果相似。
展望
在PubChemQC PM6数据集的帮助下,许多研究方向都可以开展,作者列出了主要的四个方向。(1)更详尽的分析,基于PubChemQC PM6的分子几何优化和电子性质计算结果,可以分析更多详细的分子性质,作者准备发表更多关于最优振动强度、模式、偶极瞬间、结构转化等分析结果,这对于材料开发应该有用。(2)使用更精细的方法进行计算。虽然PM6是一种比较完善的半经验量子化学计算方法,但增强计算资源仍有更多精细的方法能更准确的优化几何结构。事实上作者基于PubChemQC PM6优化的几何构型,正在试图结合B3LYP方法开发计算方法,使用目前的结果创建更全面的数据集。众所周知色散相互作用对于获得分子的平衡几何结构很重要,但PM6方法和B3LYP方法均不能恰当的处理色散相互作用。因此能恰当处理色散相互作用(如PM6-D3H4, DFT-D, ωB97X-D, ωB97M-V, vdW-DF-04, VV10, MP2, CCSD等)的计算方法将是另一个非常有价值的研究。因此相信PubChemQC PM6的结果能减少计算费用。(3)气相外的计算。目前所有的PubChemQC PM6的计算结果都是用于气相环境中,但是分子的几何结构在溶液和晶体环境中与在气相中有时会有很大的不同。因此在气相环境外的计算和创建相对应的数据集将非常有用。溶液(如水、乙醇等)环境的计算虽然会较昂贵,但PubChemQC PM6能在一定程度上降低计算负担。另外,如果没有事先了解晶体的空间群或晶胞内的分子数,晶体环境中的计算会更加有挑战性,因为会产生组合爆炸。(4)比较PubChemQC PM6的计算结果和实验确定的结构。量子化学计算有时会偏离实验数据,因此,系统对比PubChemQC PM6的计算结果和实验结果有助于验证和提高量子化学计算理论和实践。然而,据我们所知,目前还没有全面的实验数据库公开发表。
参考文献
Nakata Maho, Shimazaki Tomomi, Hashimoto Masatomo and Maeda Toshiyuki, PubChemQC PM6: Data Sets of 221 Million Molecules with Optimized Molecular Geometries and Electronic Properties. J. Chem. Inf. Model. 2020, ASAP.