
引言
在困扰于新疾病的现实世界中,我们必须加快药物设计进程以开发针对这些新疾病的新疗法。近年来,基于深度学习的方法在基于配体的药物设计中逐渐崭露头角。然而,这些方法在针对新靶标设计药物时面临数据匮乏的问题。在本工作中,作者利用了深度学习和分子建模方法的巨大潜能来开发药物设计流程,这对于靶标特异性配体数据集有限或不可用的情况很有用。在靶蛋白的活性位点筛选靶蛋白同系物的抑制剂以产生初始靶标特异性数据集,之后使用迁移学习用于学习靶标特定数据集的特征,然后利用深度预测模型预测新设计分子的对接打分,最后通过强化学习将这两种模型结合起来,以优化的对接打分设计新的化学小分子。通过设计针对人类JAK2蛋白的抑制剂来验证该方法工作流程,并且其中没有使用现有的JAK2抑制剂数据进行训练。结果显示该方法能够从验证数据集中重现现有抑制剂分子并设计出具有更好结合能力的分子,进而证明了该方法的实用潜力。
背景介绍
传统的药物设计方法通过筛选可在公共或商业分子库中获得的化合物或者通过使用片段和药效团模型从头产生分子来识别对目标靶蛋白具有特异性的分子。但是,使用传统方法仅仅探索了化学空间的一部分,即大约十亿个小分子,而实际的化学空间大约为~10^63个分子或更多。深度学习方法在一定程度上可以弥合这一差距,并设计具有所需类药物特性的新化学实体。
人工智能(AI)和大数据领域的最新进展表明,有可能从根本上改变计算模型在包括药物发现在内的医疗领域的准确性和可靠性。简化的分子输入线输入系统(SMILES)表示或分子图表示通常用于训练深度神经网络模型以学习特征表示。尽管AI早期研究的重点是用于虚拟筛选库的生成,但为优化性能而引入强化学习(reinforcement learning)的方法有助于使模型产生偏向,从而生成具有目标特性的化合物。此外,通过使用记忆增强神经网络(memory-augmented neural networks),可以显著提高模型产生化学有效分子的效率。尽管基于AI的方法的应用取得了一些进步,但是蛋白质特异性药物发现的数据可用性目前仍然是一个巨大的挑战。
本文研究中,作者开发了一种从头设计药物的流程,该流程可用于针对新型靶标蛋白(其3D结构已知或已建模并且可以表征活性位点)。作者先使用生成模型进行训练以学习已知的类药物分子的语法,然后应用分子建模技术来编译特定于靶标蛋白的数据集,并且使用迁移学习(transfer learning)学习已编译数据集的特征。最后,强化学习被用于组合生成模型和预测模型,从而优化打分函数以产生预计可与靶标蛋白更好结合并具有所需药物特性的小分子。
方法
整个从头药物开发流程可分为以下几个部分:(1)数据集管理、预处理和训练生成模型:从ChEMBL数据库获得用于训练生成模型和案例研究的数据集,这些分子以SMILES格式表示,学习SMILES语法的问题被转换为Seq2Seq问题(机器翻译),生成模型用来对具有堆栈增强递归神经网络的SMILES数据集进行模拟。通过应用顺序过滤器对SMILES数据集进行预处理,以除去立体化学、盐和具有不利原子或基团的分子。将获得的SMILES字符串规范化,并删除重复项,仅收集长度≤100的分子;(2)生成靶标特异性配体数据集并进行迁移学习:来自相似蛋白质的小分子精选数据集经过了广泛的预处理,选择具有高生物活性(pChEMBL分数≥6.0)的分子来训练生成模型。通过使用AutoDock Vina将这些分子对接到靶标蛋白质的活性位点上来进一步提高该数据集的靶标选择性。具有高对接得分的分子用于重新训练生成模型,以通过迁移学习来捕获特定于目标受体的分子特征;(3)训练预测模型:预测模型用于学习小分子与它们相应的实验确定的属性值(如生物活性)之间的映射关系;(4)增强学习来生成特性优化的分子:使用强化学习将迁移学习后获得的生成模型与预测模型相结合,以生成具有模型偏向且感兴趣属性的分子;(5)通过理化性质过滤产生的分子;(6)应用基于规则的过滤器除去具有不良基团的分子:使用泛化学干扰化合物(PAINS)过滤器、BRENK过滤器、NIH过滤器和ZINC过滤器对经过理化性质过滤器应用后获得的分子集进行进一步筛选;(7)通过分子模型验证过滤后的分子:为了研究溶剂的影响并计算结合自由能,使用GROMACS 2016和CHARMM36力场进行分子动力学模拟。整个从头小分子设计流程如图1所示。
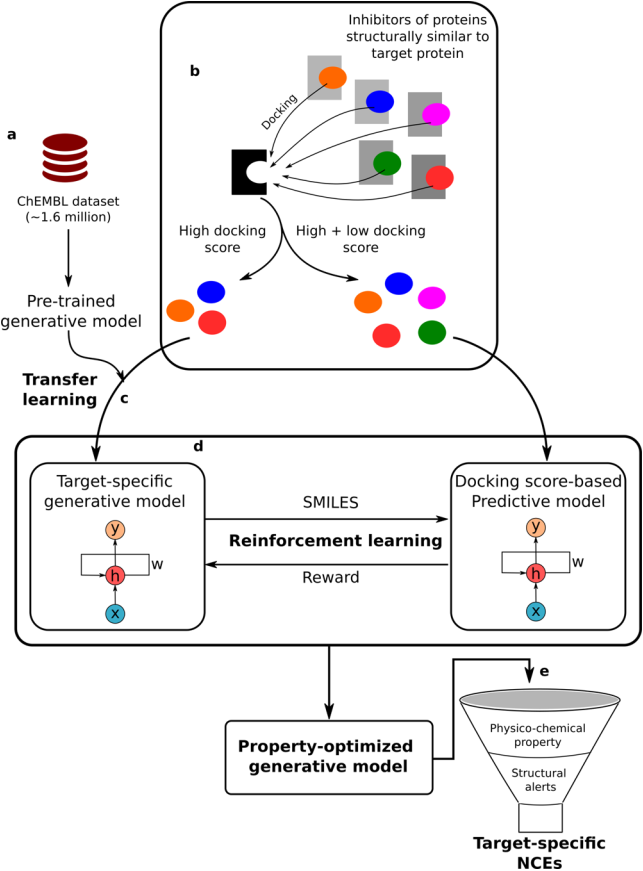
图1. 从头开始的小分子设计流程。其中x,h,y和w分别对应于输入,隐藏状态,输出和权重
图片源自JCIM
结果1. 设计具有Janus Kinase 2(JAK2)蛋白特有的最佳对接得分的小分子
选择已知抑制JAK1、JAK3和TYK2蛋白的配体来构建靶标特异性训练数据集。经过预处理、规范化和除去JAK1、JAK3和TYK2抑制剂中的多余分子后,最终获得了4167个分子的数据集,其中3711个分子的pChEMBL得分≥6.0。为确保3711个抑制剂分子的特异性,使用AutoDock Vina通过将其对接到人JAK2蛋白的活性位点来进一步筛选数据集,仅将虚拟筛选分数≤-7.0的3681个分子用于通过迁移学习训练生成模型。从3681个分子的选定数据集中,进行100个epochs的迁移学习,直到推断出的分子与训练数据集之间的Tanimoto系数分布没有进一步的改善(图2a)。我们选择了对接得分预测模型,因为它比实际对接过程快几个数量级。经过广泛的超参数调整后,预测模型可以在0.5的均方根误差(rmse)范围内预测SMILES字符串的对接得分。接下来,将迁移学习后获得的靶标特定生成模型与预测模型进行对接分数优化,使用正则强化学习持续70个epochs。强化学习之前和之后的预测对接分数的分布被视为终止训练过程的标准(图2b)。强化学习后,从训练后的生成模型中采样了10000个分子,其中有93%(9290)在化学上是有效的。去除多余的(15.76%)和训练固定的相同分子(2.45%)后,获得了7469个小分子。通过使用属性过滤器获得了6691个分子的数据集。虽然药物相似性得分的定量估计值分布(图2c)仍然相似,但最终的分子集合也比训练数据集具有更好的可合成性得分(图2d)。
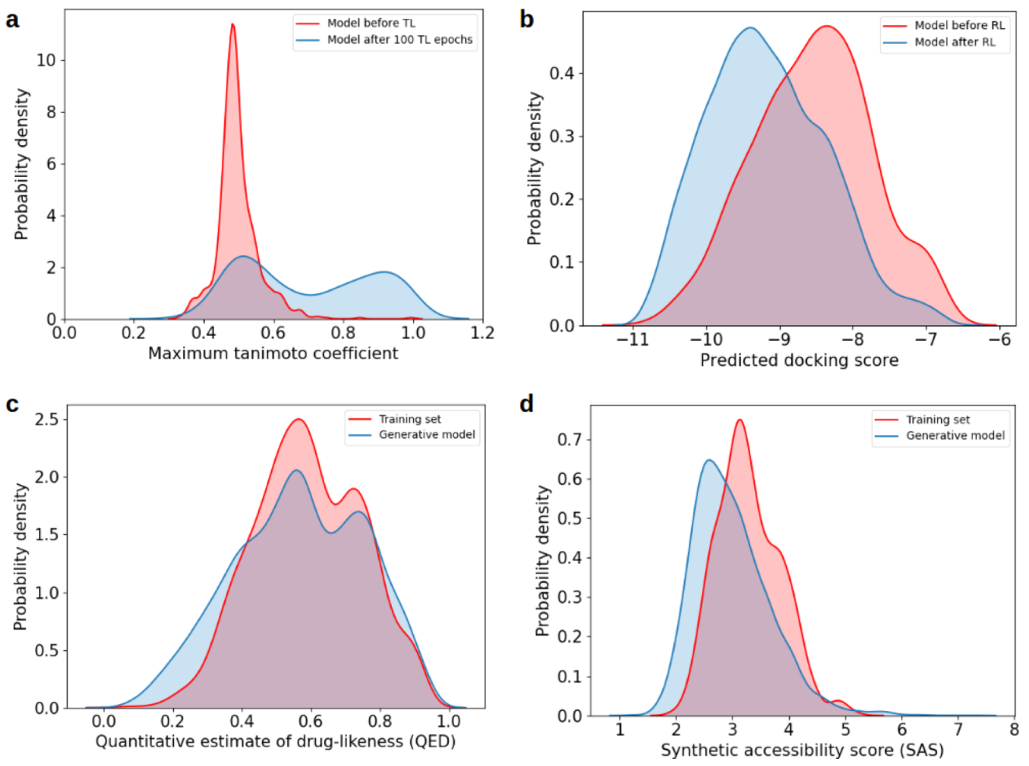
图2. 迁移学习和强化学习期间分子属性分布的变化
图片源自JCIM
结果2. 生成模型捕获训练数据集的特征
为了了解所生成分子的亚结构特征,我们计算了每个batch的不同分子片段的频率,并计算所有batches的平均片段频率。表1中列出了前10个片段的平均频率。有趣的是,鉴定出的所有前10个片段通常用于设计和合成高选择性JAK2抑制剂。值得注意的是,叔胺已显示出增加JAK2抑制剂的选择性和合成的容易性,并且已知双环基团相对于JAK1、JAK3和TYK2可以提高抑制剂对JAK2的选择性。
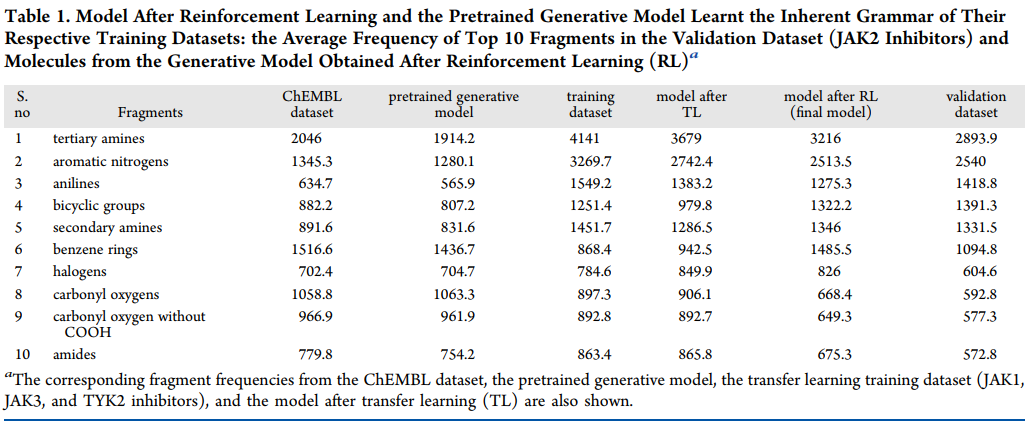
表1. 验证数据集中前10个分子片段的平均频率
表格源自JCIM
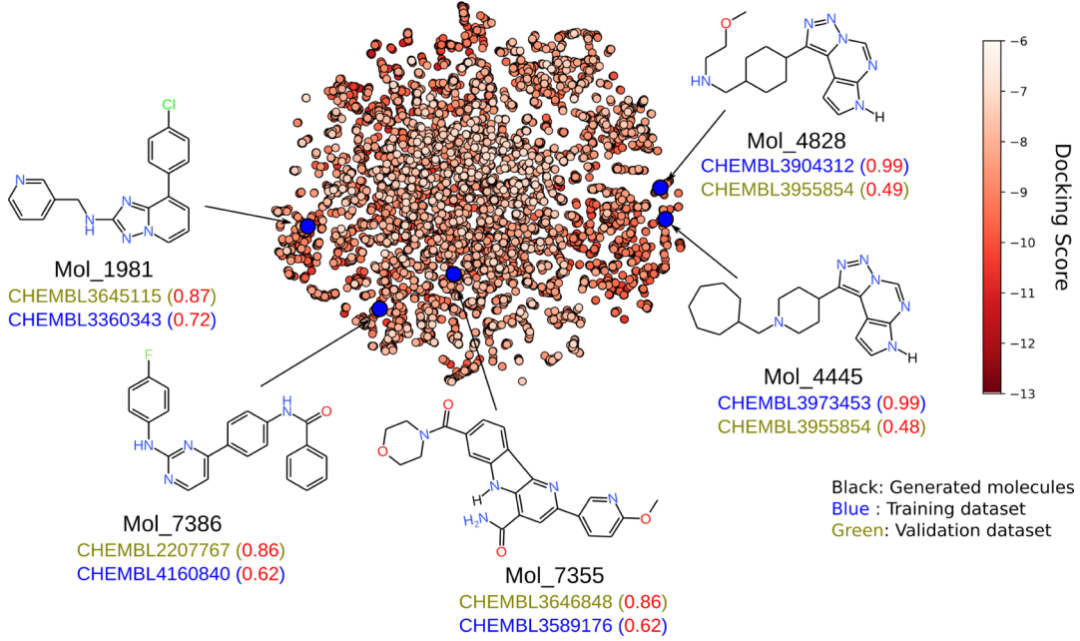
图3. 优化的生成模型从训练数据集中学习并生成接近验证数据的分子
图片源自JCIM
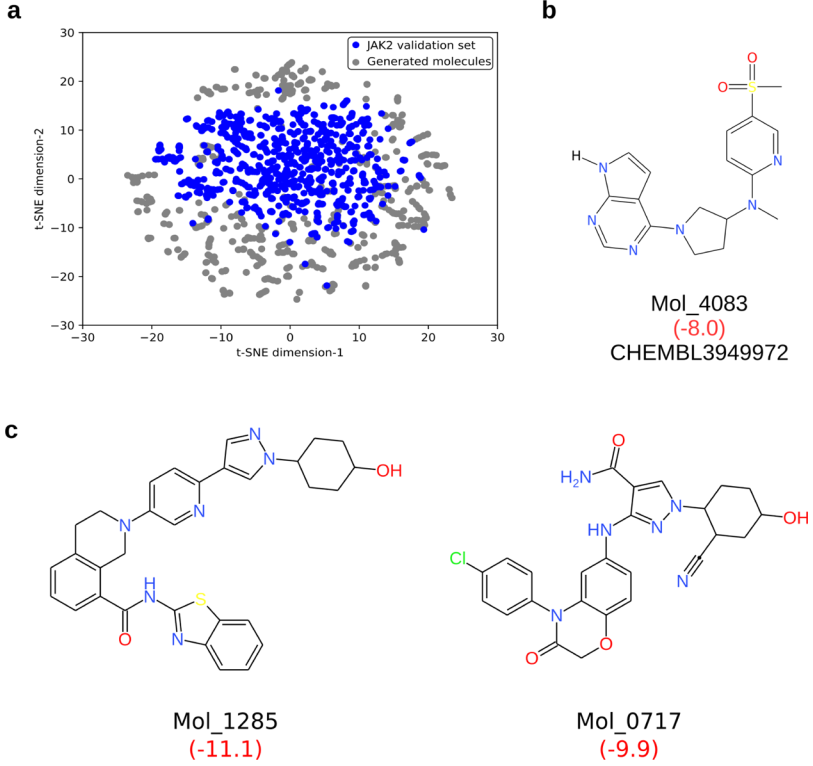
图4. 生成与验证数据集相同的小分子以及具有改进的对接得分的分子
图片源自JCIM
将迁移学习和强化学习后的平均片段频率与训练数据集和验证数据集的频率进行比较可以发现,迁移学习后,来自模型的平均片段频率类似于训练数据集的频率,而强化学习后,来自模型的平均片段频率类似于验证数据集的频率。例如,转移学习后的叔胺频率为3679,而强化学习后的频率为3216。强化学习后的频率降低表明该模型正在生成更接近验证数据集而不是训练数据集的分子。通过各种过滤器的强化学习模型中的小分子显示为低维embedding(图3)。从图中可以推断,生成模型能够生成与训练数据集和验证数据集高度相似(使用Tanimoto系数量化)的小分子。
结果3. 生成的小分子与验证数据集的相似性
为了进一步了解生成的分子相对于验证数据集的分布,图4a显示了较低维的embedding。扩展连接指纹(ECFP4)被用作embedding的描述符。从embedding中可以推断出,尽管与验证数据集的子集相似,但这些分子也已从特异JAK2抑制剂占据的化学空间的更优化子空间中采样(图4a)。这可以通过生成模型在增强学习后生成具有高于预期的对接得分和具有更好的理化性质的分子的能力来证实。将虚拟筛选后最终获得的6106个分子与特异JAK2抑制剂的验证数据集进行比较可以发现,相对于来自验证数据集的分子,生成的分子集中的310个分子(5%)的Tanimoto系数高于0.75。在这310个分子中,还发现一个分子与验证数据集相同(图4b)。基于虚拟筛选得分,与已知抑制剂分子相比,新分子可能是更好的JAK2抑制剂,且这些代表性分子的子集如图4c所示。根据MM/PBSA计算,与现有的JAK2抑制剂(-28.9 kJ/mol)相比,新分子的预测结合自由能(Mol_1285和Mol_0717分别为-87.6和-75.3 kJ/mol)表现更好。
总结
作者最后总结了使用本文提出的基于深度学习的方法的优势:(1)能够有效探索化学空间。传统的从头药物设计方法主要集中在与现有抑制剂具有高度骨架相似性的新型小分子的产生上,而深度学习模型显示了通过生成模型生成完全新颖的骨架和小分子的能力。从研究结果可以看到,与训练和验证数据集相比,所生成分子的骨架中有25.21%是新颖的;(2)靶标特定分子设计。通过使用迁移学习,深度学习模型能够从靶标特异性小分子数据集中捕获药效学表现形式;(3)物理化学性质的动态控制。可以使用增强学习将对接分数替换为所需的理化特性,以进行实时性能优化。
参考文献
Sowmya Ramaswamy Krishnan, Navneet Bung, Gopalakrishnan Bulusu, and Arijit Roy, Accelerating De Novo Drug Design against Novel Proteins Using Deep Learning, J. Chem. Inf. Model. 2021, 61(2), 621-630. DOI: 10.1021/acs.jcim.0c01060.